摘要gydF4y2Ba
染色体外DNA (ecDNA)上的癌基因扩增是一种常见的事件,导致肿瘤侵袭性生长,耐药和缩短生存期。目前,人们对非染色体癌基因遗传的影响——通过血统随机识别——知之甚少。ecDNA对体细胞变异和选择的影响也不清楚。在这里,我们整合了随机分离、无偏置图像分析、基于crispr的ecDNA标记与活细胞成像和CRISPR-C的理论模型,证明了随机ecDNA遗传导致广泛的肿瘤内ecDNA拷贝数异质性和对代谢应激和靶向治疗的快速适应。观察到的ecdna有利于宿主细胞的生存或生长,并且可以在单个细胞周期内发生变化。ecDNA遗传可以先验地预测含有ecDNA的癌症的一些侵袭性特征。这些特性是由ecDNA快速适应基因组的能力促进的,而这种能力是不可能通过染色体癌基因扩增实现的。这些结果显示了ecDNA的非染色体随机遗传模式如何导致癌症患者的不良预后。gydF4y2Ba
主要gydF4y2Ba
遗传、变异和选择是达尔文有机体进化的基本原则,这些原则被用来解释癌症是如何出现、发展和适应的gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.遗传同一性的概念是进化理论应用于癌症的核心,它提出了在细胞分裂过程中通过染色体遗传进行同一性的物理基础,从而解释了肿瘤中常见的克隆轨迹gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba.然而,有几个问题对目前的肿瘤克隆进化模型提出了挑战。首先,某些侵袭性癌症保持高水平的瘤内拷贝数异质性,而不是像预测的那样进行选择性清扫gydF4y2Ba8gydF4y2Ba.这对于放大的致癌基因尤其如此,尽管具有适应度优势,但其细胞间的变异性很高gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba.因此,维持异质性癌基因扩增事件的机制一直难以建立。其次,某些癌症通过改变基因组,特别是改变扩增致癌基因的拷贝数,来迅速适应不断变化的条件(包括治疗)的能力,目前的基因遗传模型并没有很好地解释gydF4y2Ba9gydF4y2Ba.第三,通过选择单个细胞或少数细胞中产生的耐药突变来预测耐药的滞后时间,在某些癌症中没有看到,这引发了肿瘤是否正在经历遗传瓶颈的问题gydF4y2Ba9gydF4y2Ba,gydF4y2Ba13gydF4y2Ba.ecDNA扩增的存在可能解释了这些矛盾的特征。缺乏着丝粒的圆形颗粒上的染色体外癌基因扩增现在被认为是人类癌症中的一种常见事件,与患者的不良预后和治疗耐药性有关gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba.有人认为,ecdna由于缺乏着丝粒,在细胞分裂过程中不平等地分离到子细胞gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba.然而,非染色体癌基因遗传在肿瘤-瘤内遗传异质性、加速肿瘤进化、增强抗环境应激能力和快速基因组改变对治疗耐药性的随机识别中的影响尚不清楚。在这项研究中,我们整合了计算机模拟、数学建模、进化理论、无偏倚图像分析、基于crispr的ecDNA标记与活细胞成像和CRISPR-C生成ecDNA,以及对患者肿瘤的纵向分析(扩展数据图)。gydF4y2Ba1gydF4y2Ba),以更好地理解ecDNA遗传及其功能后果。gydF4y2Ba
ecDNA在人类癌细胞中的随机分离gydF4y2Ba
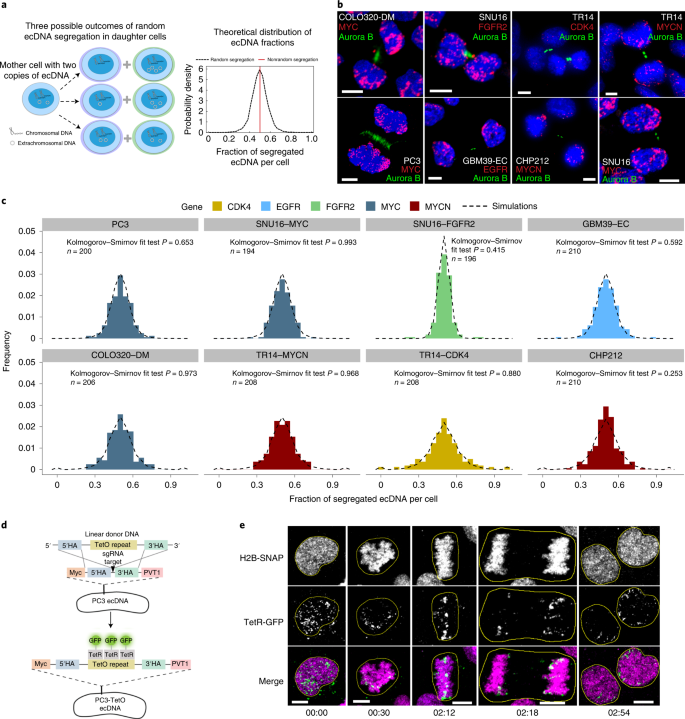
首先,我们测试了不同的ecdna扩增癌基因在细胞分裂后是否随机分离,或者我们是否可以观察到癌基因特异性差异。有丝分裂细胞分裂期间的染色体分离确保每个子细胞具有相同的DNA含量(图。gydF4y2Ba1gydF4y2Ba(红线),尽管在癌症中,染色体分离的失调也会导致分离错误gydF4y2Ba19gydF4y2Ba.如果ecDNA分离是完全随机的,在子细胞之间继承ecDNA的概率相等,那么我们预测有丝分裂后ecDNA的每个细胞含量近似高斯分布(图2)。gydF4y2Ba1gydF4y2Ba及补充资料gydF4y2Ba1.1gydF4y2Ba).因此,我们开发了一种基于FISH的方法,结合无偏置图像分析来量化细胞分裂后子细胞中的ecDNA,使用FISH探针检测这些ecDNA上的扩增癌基因,并使用Aurora B激酶免疫染色来识别有丝分裂晚期的子细胞gydF4y2Ba20.gydF4y2Ba(无花果。gydF4y2Ba1 bgydF4y2Ba).在不同组织学类型的癌细胞系中,包括前列腺、胃、结肠、神经母细胞瘤和胶质母细胞瘤,在ecDNA上携带不同的癌基因,包括含有多种含癌基因ecDNA的癌细胞系,我们量化了每个细胞系大约100对有丝分裂后子细胞的ecDNA分布,这允许足够的分辨率(扩展数据图)。gydF4y2Ba2gydF4y2Ba及补充资料gydF4y2Ba2.3gydF4y2Ba).这些实验揭示了一个广泛的近似高斯分布,与癌细胞类型或ecDNA上包含的癌基因无关。gydF4y2Ba1 b, cgydF4y2Ba和扩展数据图。gydF4y2Ba2摄氏度gydF4y2Ba).每个子细胞分离的ecDNA的比例(直方图)与随机分离的理论预测(虚线)高度一致(Kolmogorov-Smirnov检验)gydF4y2BaPgydF4y2Ba> 0.05,扩展数据图gydF4y2Ba2摄氏度gydF4y2Ba)(图。gydF4y2Ba1 cgydF4y2Ba而且gydF4y2Ba方法gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba, ecDNA分离示意图和ecDNA组分的预测分布。gydF4y2BabgydF4y2Ba在有丝分裂晚期的多个癌细胞系中,通过Aurora B中体染色鉴定ecDNA分布到子细胞的代表性图像。gydF4y2BacgydF4y2Ba中分析的癌细胞系中ecDNA片段的频率直方图gydF4y2BabgydF4y2Ba,显示模拟随机隔离(点状分布)和观测(彩色分布)之间的一致性(Kolmogorov-Smirnov检验)gydF4y2BaPgydF4y2Ba> 0.05)。gydF4y2BadgydF4y2Ba,用于前列腺癌细胞中ecDNA活细胞成像的基于crispr的遗传方法示意图。HA,同源臂。gydF4y2BaegydF4y2Ba,活细胞延时成像显示ecDNA在子细胞间分布不均。时间戳,hh:mm。比例尺,5 μm。gydF4y2Ba
为了证实这些相关的观察,我们设计了一个活细胞成像系统来可视化细胞分裂过程中的ecDNA动态。我们使用CRISPR-Cas9(参考。gydF4y2Ba21gydF4y2Ba)将TetO阵列插入到之间的基因间区gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaPVT1gydF4y2BaPC3前列腺癌细胞中ecDNA的表达(图;gydF4y2Ba1 dgydF4y2Ba).通过PCR、Sanger测序和TetO-MYC dual FISH证实了该阵列的插入。gydF4y2Ba3模拟gydF4y2Ba).随后,绿色荧光蛋白(GFP)的表达融合到Tet阻遏物tetra -GFP,它结合TetO阵列,使ecDNA在整个细胞周期的跟踪(图。gydF4y2Ba1 dgydF4y2Ba).用新开发的JF669 SNAP标签配体标记的组蛋白H2B-SNAP标签融合检测染色质gydF4y2Ba22gydF4y2Ba.PC3-TetO细胞的活细胞延时成像显示细胞分裂过程中ecDNA的随机遗传模式(图2)。gydF4y2Ba1 egydF4y2Ba及补充视频gydF4y2Ba1gydF4y2Ba).在证明了ecDNA在细胞分裂过程中是随机分离的之后,我们研究了ecDNA随机分离如何影响达尔文进化论的其他支柱,即变异和选择。gydF4y2Ba
ecDNA导致瘤内异质性gydF4y2Ba
肿瘤内异质性在治疗耐药和肿瘤进化中起着重要作用gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba.为了更好地理解ecDNA的随机分离如何促进肿瘤内异质性,我们进行了基于个体的随机计算机模拟,假设细胞分裂过程中随机ecDNA分离gydF4y2Ba2.1gydF4y2Ba).我们制定了ecDNA的每个细胞分布的动态(图。gydF4y2Ba2gydF4y2Ba及补充资料gydF4y2Ba3 gydF4y2Ba),基于所观察到的随机偏析模式。假设独立复制和随机分离,ecDNA动力学可以转化为一组耦合微分方程,其中gydF4y2BaNgydF4y2BakgydF4y2Ba(gydF4y2BatgydF4y2Ba)表示含有的单元格数gydF4y2BakgydF4y2Ba每次复制ecDNAgydF4y2BatgydF4y2Ba而且gydF4y2Ba年代gydF4y2Ba是选择系数。单元格的数目gydF4y2BakgydF4y2BaecDNA随着时间的变化:gydF4y2Ba
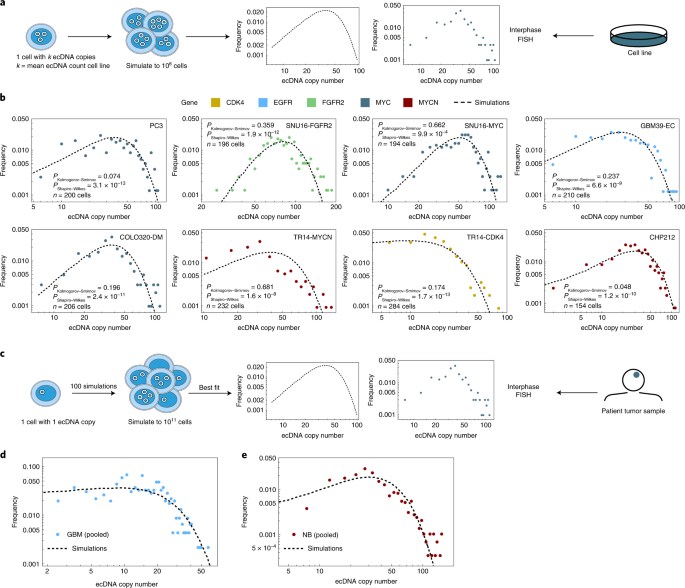
一个gydF4y2Ba,通过随机ecDNA分离和ecDNA模拟,显示ecDNA拷贝数异质性量化的示意图gydF4y2Ba+gydF4y2Ba细胞系。gydF4y2BabgydF4y2Ba,癌细胞系间期FISH检测的ecDNA癌基因拷贝数。观察到的(彩色直方图)和模拟的(虚线直方图)之间的一致性表明,癌基因拷贝数异质性在很大程度上遵循预测的分布。未经调整的gydF4y2BaPgydF4y2Ba给出了Shapiro-Wilks和Kolmogorov-Smirnov检验的值。gydF4y2BacgydF4y2Ba,通过随机ecDNA分离和ecDNA模拟,显示ecDNA拷贝数异质性量化的示意图gydF4y2Ba+gydF4y2Ba患者数据。gydF4y2BadgydF4y2Ba, 6例GBM患者的ecDNA拷贝数分布(点)gydF4y2BaegydF4y2Ba, 4例出现NB的患者(点)均来自同样的随机ecDNA分离过程(黑色虚线)。gydF4y2Ba
为了使这个问题在计算上易于处理,我们使用了Metropolis Hastings的Gillespie算法实现gydF4y2Ba25gydF4y2Ba.为了模拟肿瘤生长,我们用含有1个ecDNA副本的单个细胞开始模拟,并运行到不同的群体大小,最多10个gydF4y2Ba11gydF4y2Ba在中性(gydF4y2Ba年代gydF4y2Ba= 1或正s > 1)选择。我们从1个细胞开始模拟细胞系实验gydF4y2BakgydF4y2BaecDNA的拷贝,其中gydF4y2BakgydF4y2Ba是感兴趣的细胞系的平均ecDNA拷贝数(补充gydF4y2Ba信息gydF4y2Ba),让人口增长到10人gydF4y2Ba6gydF4y2Ba细胞。对于这些模拟,ecDNA拷贝数分布非常广泛。许多细胞被预测携带少量ecDNA副本,而少数细胞携带许多(多达数百个)ecDNA副本。然而,在中性选择和阳性选择下,ecDNA动力学存在根本差异。如果ecDNA处于正选择状态,预计其分布将及时向更高的拷贝数转移,而中性进化时分布仍将保持在初始的ecDNA拷贝数。gydF4y2Ba
然后,我们将模拟预测的ecDNA拷贝数分布与来自6个ecDNA的经验数据进行了比较gydF4y2Ba+gydF4y2Ba具有已知ecdna扩增癌基因的不同癌症类型的肿瘤系。对于胶质母细胞瘤(GBM)和成神经细胞瘤(NB)这两种癌症类型,我们有真正的肿瘤组织和临床数据来扩展我们的研究gydF4y2Ba9gydF4y2Ba.我们还选择了两种具有两种不同ecDNA的癌细胞系(图。gydF4y2Ba2 bgydF4y2Ba).在这些癌细胞系模型中,观察到的每个细胞ecDNA拷贝数分布也非常宽,与我们模拟预测的分布相匹配的细胞与细胞之间的极端差异(图2)。gydF4y2Ba2 bgydF4y2Ba, Kolmogorov-Smirnov检验gydF4y2BaPgydF4y2Ba> 0.05和扩展数据图gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
然后,我们通过量化6例GBM患者肿瘤切片上表皮生长因子受体(EGFR) FISH探针染色的细胞分布,将我们的分析扩展到临床样本,我们还量化了4例NB患者肿瘤切片上MYCN FISH探针的细胞分布。这些患者的ecDNA上都有扩增的癌基因(图。gydF4y2Ba2摄氏度gydF4y2Ba和扩展数据图。gydF4y2Ba5gydF4y2Ba).尽管这些组织样本很小,导致每个样本的细胞数量少得多,但我们仍然观察到ecDNA拷贝数分布再次显示出极端的细胞间差异,与我们模拟预测的分布相匹配(Kolmogorov-Smirnov检验)gydF4y2BaPgydF4y2Ba> 0.05,扩展数据图gydF4y2Ba5 bgydF4y2Ba;无花果。gydF4y2Ba2 d, egydF4y2Ba和扩展数据图。gydF4y2Ba5 bgydF4y2Ba).gydF4y2Ba
然后,我们询问是否在我们的细胞系数据和患者样本中都有ecDNA扩增癌基因阳性选择的迹象,或者观察到的ecDNA异质性模式是否可以仅用中性进化和随机分离来解释。我们的理论模型在积极选择或中性选择下对ecDNA进行动态预测(扩展数据图)。gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba6gydF4y2Ba).两个主要的区别是ecDNA的比例gydF4y2Ba+gydF4y2Ba细胞和大细胞群中ecDNA的平均拷贝数。在ecDNA进化的中性模型中,ecDNA的部分gydF4y2Ba+gydF4y2Ba在大群体中,细胞数量下降并接近0,而平均ecDNA拷贝数与时间无关且不变,例如,如果群体由具有1个ecDNA副本的单个细胞启动,则它将保持1。相反,对于正选择下的ecDNA, ecDNA的比例gydF4y2Ba+gydF4y2Ba在大群体中,细胞接近1,平均ecDNA拷贝数随着群体规模的增加而增加。细胞系和患者数据均与阳性选择下的ecDNA模型一致(扩展数据图。gydF4y2Ba6 b, cgydF4y2Ba).然而,这些观察是间接的和定性的,随着时间的推移,平衡选择的作用不能排除。gydF4y2Ba
药物诱导的ecDNA选择gydF4y2Ba
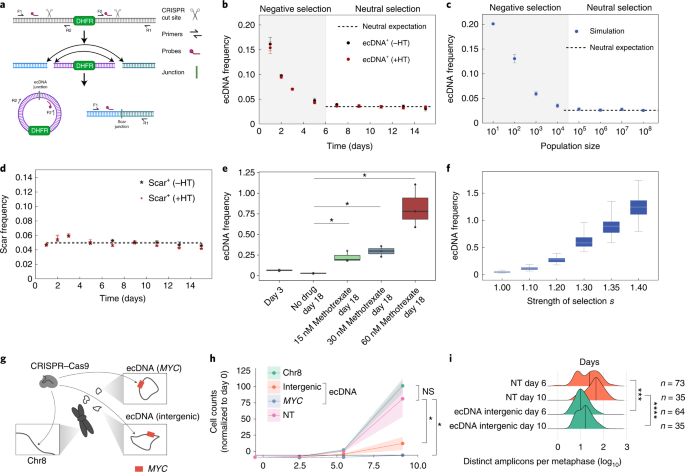
为了直接检验我们模型的预测并解决ecDNA分布的时间动态,我们开始从实验上量化ecDNA的进化。我们使用了CRISPR-CgydF4y2Ba26gydF4y2Ba产生含有二氢叶酸还原酶的ecdna (gydF4y2BaDHFRgydF4y2BaHAP1癌细胞系是一种近单倍体的慢性骨髓性白血病人癌细胞系(图2)。gydF4y2Ba3gydF4y2Ba).在CRISPR-C后,大约15%的细胞含有ecDNA,每个携带ecDNA的细胞恰好有一个副本。因此,我们能够使用数字液滴PCR来测量ecDNA是如何随着时间的推移而进化的。在CRISPR切割和宗教染色体后留下的染色体“疤痕”的存在,使同一细胞群中的染色体外和染色体动力学的直接比较成为可能。此外,通过生成包含的ecdnagydF4y2BaDHFRgydF4y2Ba基因,我们也能够模拟ecDNA的中性选择或阳性选择的影响,分别在没有或存在甲氨蝶呤时,它针对DHFR并中断核苷酸代谢gydF4y2Ba27gydF4y2Ba.ecDNA诱导对细胞不利,ecDNA拷贝最初丢失(图。gydF4y2Ba3 bgydF4y2Ba).在没有甲氨蝶呤的情况下,在最初选择能够耐受和维持ecDNA拷贝的细胞后,平均ecDNA拷贝数保持不变,与我们的模型预测的中性选择一致(图3)。gydF4y2Ba3 cgydF4y2Ba).染色体瘢痕频率在整个实验过程中也保持不变,这与中性选择一致(图2)。gydF4y2Ba3 dgydF4y2Ba).相反,我们观察到在甲氨蝶呤治疗后,ecDNA拷贝数出现了强烈的剂量依赖性上升(图2)。gydF4y2Ba3 egydF4y2Ba),这与不同正选择强度的模拟高度一致,并提供了明确的证据,证明含有gydF4y2BaDHFRgydF4y2BaecDNA克服甲氨蝶呤治疗(图;gydF4y2Ba3 fgydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba,描述用于在HAP1细胞中生成单个ecDNA的CRISPR-C策略的示意图gydF4y2BaDHFRgydF4y2Ba基因。gydF4y2BaDHFRgydF4y2BaecDNA和染色体疤痕通过ddPCR检测新的连接位点。gydF4y2BabgydF4y2Ba,在CRISPR-C诱导ecDNA 0天后,用ddPCR追踪HAP1细胞中ecDNA的平均拷贝数。中性选择gydF4y2BaDHFRgydF4y2Ba通过次黄嘌呤和胸苷缺失或包含的相似性观察到的ecDNA。c,模拟实验条件下的平均ecDNA数的模拟gydF4y2BabgydF4y2Ba.消极的选择gydF4y2Ba年代gydF4y2Ba= 0.5,中性选择gydF4y2Ba年代gydF4y2Ba= 1。gydF4y2BadgydF4y2Ba, ddPCR测定染色体疤痕在疤痕交界处的平均频率。gydF4y2BaegydF4y2Ba,第0天ecDNA诱导后的平均ecDNA拷贝数±第4天开始的甲氨蝶呤治疗。gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BaegydF4y2Ba, 3个生物重复的CRISPR-C数据;数据以均数±s.e.m表示;gydF4y2BaPgydF4y2Ba双面的值gydF4y2BatgydF4y2Ba测试。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba的箱形图,线在中位数处,框在25 - 75百分位,须向最极值延伸。gydF4y2BafgydF4y2Ba,模拟实验中ecDNA平均拷贝数的模拟gydF4y2BaegydF4y2Ba.消极的选择gydF4y2Ba年代gydF4y2Ba= 0.5,持续4 d,然后按照14 d的指示进行不同水平的选择强度。箱形图显示中位数处的直线和从第25至第75百分位的方框,胡须延伸到最极端的值。gydF4y2BaggydF4y2Ba,描述基于crispr的策略,以测试给予COLO320-DM细胞的选择优势gydF4y2BaMYCgydF4y2BaecDNA。箭头表示sgRNA靶向的区域。gydF4y2BahgydF4y2Ba,基因组编辑gydF4y2BaMYCgydF4y2Ba编码在ecDNA上导致了细胞数量的大量减少,这超过了基因间编辑ecDNA的影响,这表明了对ecDNA上的癌基因的强选择。数据以平均值±标准差表示。与gydF4y2BaPgydF4y2Ba双面的值gydF4y2BatgydF4y2Ba测试;数据来自两个独立的副本。NS,不显著;NT, nontransfected。gydF4y2Ba我gydF4y2Ba,在CRISPR转染后6和10 d,每个中期ecDNA数量的量化。数据用竖线标记中位数,gydF4y2BaPgydF4y2Ba曼-惠特尼值gydF4y2BaUgydF4y2Ba测试。*gydF4y2BaPgydF4y2Ba≤0.05;**gydF4y2BaPgydF4y2Ba≤0.005;***gydF4y2BaPgydF4y2Ba≤0.0005;****gydF4y2BaPgydF4y2Ba≤0.00005。gydF4y2Ba
为了进一步分析携带癌基因的ecdna是否处于阳性选择状态,我们部署了靶向COLO320-DM不同基因组区域的引导rnagydF4y2BaMYCgydF4y2BaecDNA (ecDNA和gydF4y2BaMYCgydF4y2BaecDNA上的基因体)和8号染色体的一个未扩增的基因间区(图。gydF4y2Ba3 ggydF4y2Ba).我们用Cas9和单导向rna (sgRNAs)通过慢病毒载体感染细胞,定量细胞增殖和ecDNA拷贝数。cas9靶向切割8号染色体对细胞增殖的影响最小,而在基因间区域靶向ecDNA则影响更大gydF4y2BaMYCgydF4y2Ba在ecDNA上,导致了极端的生长缺陷(图。gydF4y2Ba3 hgydF4y2Ba).当我们定量这些细胞中的ecDNA拷贝数时,我们发现在初次感染后6 d, ecDNA显著减少。gydF4y2Ba3我gydF4y2Ba和扩展数据图。gydF4y2Ba6 dgydF4y2Ba).综上所述,这些CRISPR-C数据(图。gydF4y2Ba3 fgydF4y2Ba)证实ecDNA及其所含的癌基因处于强大的选择压力下,这影响了肿瘤中ecDNA癌基因的平均拷贝数和每个细胞的分布。细胞系模型和肿瘤样本中ecDNA拷贝数的相关分析与我们的模拟和肿瘤中携带癌基因ecDNA的强阳性选择模型高度一致(扩展数据图)。gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba6摄氏度gydF4y2Ba).然而,悬而未决的问题依然存在。随着在不久的将来获得更多的数据和更高的单细胞分辨率,探索在癌症患者中平衡或其他形式的拷贝数依赖的ecDNA选择的潜在作用,并确定形成这些过程的力量将非常重要。绘制不同ecDNA扩增癌基因在单细胞分辨率下的时间分辨的ecDNA拷贝数分布将是未来更好地理解更复杂适应度模型的关键工作。gydF4y2Ba
ecdna能使肿瘤快速适应应激gydF4y2Ba
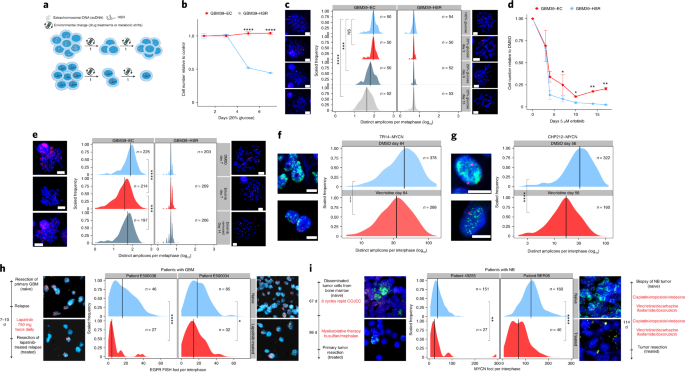
在证明了ecDNA对达尔文进化论的三大支柱——遗传(即通过遗传随机识别)、变异和选择——都有贡献之后,我们提出了一个问题,即这些ecDNA特征是否比染色体遗传更能使肿瘤更快地适应应激(图2)。gydF4y2Ba4gydF4y2Ba).我们利用来自GBM患者的等基因细胞系对gydF4y2Ba9gydF4y2Ba研究ecDNA在驱动快速适应中的重要性。GBM39-EC是一种患者来源的神经球模型,其平均拷贝数约为100份gydF4y2BaEGFRvIIIgydF4y2Ba,一种存在于ecDNA上的功能获得性EGFR突变gydF4y2Ba15gydF4y2Ba,gydF4y2Ba28gydF4y2Ba.gbm39 -均匀染色区(HSR)是一个等基因模型gydF4y2BaEGFRvIIIgydF4y2Ba扩增子位于染色体HSRs上,具有相同的DNA序列,具有相同的平均拷贝数(扩展数据图)。gydF4y2Ba7一个gydF4y2Ba)gydF4y2Ba28gydF4y2Ba.重要的是,异质性gydF4y2BaEGFRvIIIgydF4y2BaGBM39-EC的拷贝数与流式细胞术评估的EGFRvIII蛋白表达的异质性相关(扩展数据图)。gydF4y2Ba7 b, cgydF4y2Ba).GBM39-EC细胞具有高度糖酵解性gydF4y2Ba9gydF4y2Ba.因此,我们测试了葡萄糖限制对GBM39-EC和GBM39-HSR细胞的差异影响。我们从培养基中提取了正常葡萄糖水平的80%,并看到了一个显著的差异——GBM39-HSR细胞对葡萄糖的提取非常敏感,而GBM39-EC细胞对细胞生长没有明显的下降(图3)。gydF4y2Ba4 bgydF4y2Ba).GBM39-EC细胞适应葡萄糖限制的能力反映在平均水平和整体分布的快速下降gydF4y2BaEGFRvIIIgydF4y2Ba每个细胞含有- ecdna(图;gydF4y2Ba4摄氏度gydF4y2Ba).值得注意的是,这种基因组转变发生在几个细胞周期内。相比之下,GBM39-HSR细胞,相对于gydF4y2BaEGFRvIIIgydF4y2Ba对葡萄糖限制高度敏感(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba,描述ecDNA的随机分离和随之而来的异质性如何驱动快速适应和抗性的示意图。gydF4y2BabgydF4y2Ba,含有ecdna的GBM细胞对葡萄糖戒断有抗性,而相同癌基因以几乎相同的拷贝数附着在染色体位点(GBM39-HSRs)上的GBM细胞不耐受葡萄糖戒断;来自三个独立重复的数据;表示为平均值±标准差。gydF4y2BacgydF4y2Ba含有ecdna的细胞对葡萄糖戒断的适应与每个细胞扩增子分布的快速变化有关,不像高度敏感的含有hsr的细胞那样。不调制扩增子拷贝数。实验的时间轴在左边。红色的FISH信号来自gydF4y2Ba表皮生长因子受体gydF4y2Ba鱼类调查。gydF4y2BadgydF4y2BaEGFRvIII在ecDNA上扩增的GBM细胞,在最初反应后,迅速对EGFR酪氨酸激酶抑制剂厄洛替尼产生耐药性,而GBM39-HSR细胞仍然高度敏感。数据以均数±标准差表示;数据来自2个独立重复(4个重复第7天)。gydF4y2BaegydF4y2Ba, GBM细胞与gydF4y2BaEGFRvIIIgydF4y2Ba在ecDNA上的扩增迅速改变了分布gydF4y2BaEGFRvIIIgydF4y2Ba每个细胞的扩增子,在第7天测量,也可以通过停药在1周内迅速逆转。实验的时间轴在左边。红色信号是gydF4y2Ba表皮生长因子受体gydF4y2Ba鱼类调查。gydF4y2BafgydF4y2Ba, NB细胞系TR14改变了的拷贝数分布gydF4y2BaMYCNgydF4y2Ba用43 nM长春新碱治疗12周后的ecDNA。gydF4y2BaggydF4y2Ba, NB细胞系CHP212改变了的拷贝数分布gydF4y2BaMYCNgydF4y2Ba经5.3 nM长春新碱处理8周后,ecDNA的变化。gydF4y2BahgydF4y2Ba的分布比较gydF4y2Ba表皮生长因子受体gydF4y2Ba2例GBM患者在治疗前(初治期)和拉帕替尼治疗7-10 d后进行了每个细胞的扩增。红色的FISH信号来自gydF4y2Ba表皮生长因子受体gydF4y2Ba鱼类调查。绿色的FISH信号来自Chr. 7控制探头。gydF4y2Ba我gydF4y2Ba,比较gydF4y2BaMYCNgydF4y2BaecDNA拷贝数评估gydF4y2BaMYCNgydF4y2Ba(绿色)2例NB患者接受长春新碱等化疗前后的FISH。红色信号来自Chr. 2控制FISH探测器。比例尺,5 μm;临床组织图像中没有标度信息。gydF4y2BaPgydF4y2Ba数值用曼-惠特尼计算gydF4y2BaUgydF4y2Ba-测试分布和双面的比较gydF4y2BatgydF4y2Ba-测试单元格数的比较。*gydF4y2BaPgydF4y2Ba≤0.05;**gydF4y2BaPgydF4y2Ba≤0.005;***gydF4y2BaPgydF4y2Ba≤0.0005;****gydF4y2BaPgydF4y2Ba≤0.0005。gydF4y2Ba
我们之前已经证明,GBM39-EC细胞可以通过降低ecDNA拷贝数对EGFR酪氨酸激酶抑制剂厄洛替尼产生可逆抗性gydF4y2Ba9gydF4y2Ba.因此,我们检测了GBM39-EC细胞是否比GBM39-HSR细胞更快地对厄洛替尼产生耐药性。与葡萄糖剥夺一样,GBM39-EC通过改变其ecDNA拷贝数来适应不断变化的条件。在最初细胞数量减少后,GBM39-EC细胞在仅两周的治疗后就对厄洛替尼产生耐药性,以可逆的方式改变其细胞ecDNA分布(图2)。gydF4y2Ba4 d, egydF4y2Ba和扩展数据图。gydF4y2Ba7 dgydF4y2Ba).相比之下,GBM39-HSR细胞没有移位gydF4y2BaEGFRvIIIgydF4y2Ba染色体拷贝数,并对厄洛替尼保持高度敏感(图。gydF4y2Ba4 d, egydF4y2Ba和扩展数据图。gydF4y2Ba7 fgydF4y2Ba).然后,我们分析了从GBM患者的肿瘤中提取的两个样本,如前所述gydF4y2Ba2gydF4y2Ba.我们比较了原发肿瘤切除(初治)和切除复发(切除前用EGFR酪氨酸激酶抑制剂拉帕替尼治疗7-10 d)。我们发现这些患者肿瘤中的平均EGFR拷贝数和ecDNA分布显著降低(图2)。gydF4y2Ba4 hgydF4y2Ba).为了将我们的分析扩展到其他含有ecdna的癌症类型,我们研究了长春新碱的作用,这是一种拮抗的化疗药物gydF4y2BaMYCNgydF4y2Ba放大gydF4y2Ba29gydF4y2Ba.体外实验中,在NB细胞系TR14和CHP212中与gydF4y2BaMYCNgydF4y2Ba在ecDNA上扩增后,长春新碱对低拷贝数的肿瘤细胞产生反应(图2)。gydF4y2Ba4 f, ggydF4y2Ba).当我们比较未经治疗的NB活检和接受包括长春新碱在内的治疗后的原发肿瘤切除术时,我们发现在平均拷贝数和ecDNA分布上有类似的显著下降gydF4y2BaMYCNgydF4y2Ba在这些患者的两种肿瘤中都有较低的拷贝数,与细胞系数据平行(图2)。gydF4y2Ba4我gydF4y2Ba).有趣的是,当使用CDK4/6抑制剂abemaciclib和更大程度上的palbociclib治疗TR14细胞时,在耐药肿瘤细胞中检测到CDK4 ecDNA分布向更高拷贝数的转移(扩展数据图)。gydF4y2Ba7 d, egydF4y2Ba而且gydF4y2Ba8gydF4y2Ba),这与先前关于不同肿瘤的报道一致,即CDK4拷贝数和高表达促进了对CDK4/6抑制剂的耐药性gydF4y2Ba30.gydF4y2Ba.gydF4y2Ba
总之,这些数据表明了一个清晰的模式,即ecDNA如何实现高水平的异质性,从而增加对环境或治疗挑战的初始抗性。此外,基于ecdna的癌基因的持续随机遗传允许通过一种在染色体改变驱动的细胞中不可能实现的机制快速适应和耐药性的形成。gydF4y2Ba
讨论gydF4y2Ba
ecDNA已成为一个重大挑战,迫使我们重新考虑对癌症的基本认识。新兴数据表明,改变的ecDNA拓扑驱动增强的染色质可及性,并重新连接基因调控,以驱动致癌转录gydF4y2Ba28gydF4y2Ba.此外,ecDNA粒子形成枢纽的独特高级组织gydF4y2Ba31gydF4y2Ba进一步有助于ecdna介导的发病机制。本文提出的发现揭示了ecDNA独特地塑造了达尔文进化的每个基本原则,即通过血统随机遗传,通过随机分离和选择增强变异,从而加速肿瘤细胞的进化和增强适应性。这些观察可以解释为什么针对癌基因扩增事件的治疗在ecdna如此普遍的GBM等肿瘤中的临床活性如此有限。治疗此类癌症可能需要在未来针对ecdna的独特适应性。gydF4y2Ba
方法gydF4y2Ba
我们的研究符合所有相关的道德准则。GBM的FISH图像来自于在UCLA接受治疗的患者,这些患者参加了由北美脑瘤联盟NABTC 04-01赞助的lapatinib的多机构II期临床试验,这是lapatinib GW572016 (lapatinib)在复发性GBM中的生物标志物和II期研究。患者样本的收集和使用得到了加州大学洛杉矶分校机构审查委员会的批准。这些样本之前已经被描述过,包括Nathanson等人。gydF4y2Ba9gydF4y2Ba.从NBs中获取FISH图像作为常规分子肿瘤诊断的一部分。患者根据儿童肿瘤学会欧洲成神经细胞瘤网络HR-NBL-1试验(NCT01704716)或德国儿童肿瘤和血液学学会(GPOH) NB2004试验的试验方案进行注册和治疗。本研究是根据世界医学协会赫尔辛基宣言(2013)和良好临床实践进行的;所有患者或其监护人均知情同意。维也纳圣安娜儿童医院、Charité-Universitätsmedizin柏林医院和科隆大学医学院的机构审查委员会批准了患者标本的收集和使用。标本和临床数据由Charité-Universitätsmedizin柏林、圣安娜儿童医院或德国公共卫生总局的国家成神经细胞瘤生物库和成神经细胞瘤试验登记处(科隆大学儿童医院)存档并提供。gydF4y2Ba
细胞培养gydF4y2Ba
细胞株是从ATCC或dsmz -德国微生物和细胞培养集(莱布尼茨研究所)购买的,或者是J.H.舒尔特的善意礼物。如前所述,GBM39-HSR和GBM39-EC来自一名GBM患者gydF4y2Ba9gydF4y2Ba.Hap1细胞(Horizon Discovery)维持在添加GlutaMAX和10% FCS (Gibco)的IMDM中。gydF4y2Ba
PC3细胞在含10% FCS的DMEM中培养。COLO320-HSR和COLO320-DM在DMEM/F12 50:50 + 10% FCS中培养。SNU16生长在Roswell Park Memorial Institute (RPMI 1640),添加10% FCS。GBM39-HSR和GBM39-EC神经球在含B27、GlutaMAX、肝素(5 μg ml)的DMEM/F12中培养gydF4y2Ba−1gydF4y2Ba)、EGF (20 ng mlgydF4y2Ba−1gydF4y2Ba)和成纤维细胞生长因子(20ng mlgydF4y2Ba−1gydF4y2Ba).TR14细胞在含20% FCS的RPMI 1640中培养。用TC20自动细胞计数器(Bio-Rad Laboratories)计算细胞数。对于药物治疗,每3-4 d更换一次药物。gydF4y2Ba
中期染色体扩散gydF4y2Ba
在中期用100 ng ml的KaryoMAX Colcemid (Gibco)处理细胞gydF4y2Ba−1gydF4y2Ba3小时至一夜之间(取决于细胞周期速度)。用PBS清洗细胞一次,单细胞悬液在75 mM KCl中37°C孵育15分钟。然后用Carnoy固定液(3:1甲醇:冰醋酸)固定细胞并旋转。细胞用固定液清洗三次。然后将细胞放入加湿的载玻片上。gydF4y2Ba
鱼gydF4y2Ba
固定样品放在盖玻片或载玻片上,在2× SSC缓冲液中进行短暂平衡。然后分别在上升的乙醇浓度为70、85和100%的条件下脱水约2分钟。FISH探针在杂交缓冲液(Empire Genomics)中稀释,并添加到样品中,添加盖片或载玻片。样品在72°C下变性2分钟,然后在37°C的潮湿黑暗室中杂交过夜。然后用0.4× SSC和2× SSC 0.1% Tween 20洗涤样品(所有洗涤时间约为2min)。4,6-二氨基-2-苯基吲哚(DAPI) (100 ng mlgydF4y2Ba−1gydF4y2Ba)作用于样品上10 min。样品再次用2× SSC 0.1% Tween 20,再用2× SSC洗涤。样品在双蒸馏H中短暂洗涤gydF4y2Ba2gydF4y2BaO和安装延长黄金。玻片用指甲油密封。gydF4y2Ba
双重immunofluorescence-FISHgydF4y2Ba
异步细胞在聚l -赖氨酸包被的覆盖物上生长(层粘连蛋白用于GBM39-EC)。PBS清洗细胞一次,用4%多聚甲醛(PFA)在室温下固定10-15分钟。样品在室温下用0.5% Triton X-100在PBS中渗透10分钟,然后用PBS洗涤。然后用3% BSA在PBS 0.05% Triton X-100中在室温下封闭样品30分钟。样品在一抗中孵育,在阻断缓冲液(1:10 - 1:200)中稀释,在室温下孵育1小时或在4℃下孵育一夜。样品在PBS 0.05% Triton X-100中洗涤三次。样品在二抗中孵育,在阻断缓冲液中在室温下稀释1小时(所有后续步骤都在黑暗中),然后在0.05% Triton X-100 PBS中洗涤三次。用PBS清洗细胞一次,用4%的冷PFA在室温下重新固定20分钟。细胞用PBS洗涤一次,然后用2× SSC缓冲液洗涤一次。FISH进行如上所述,区别如下:变性在80°C进行20分钟。gydF4y2Ba
显微镜gydF4y2Ba
常规荧光显微镜使用Olympus BX43显微镜;图像是用QIClick冷却相机获取的。共聚焦显微镜使用具有闪电反褶积的徕卡SP8显微镜进行(加州大学圣地亚哥医学院显微核心)。NB细胞株用Leica TCS SP5显微镜、HCC PL APO lambda blue ×63 1.4油透镜或DeltaVision Elite cell Imaging System (Applied Precision)和显微镜(IX-71型;由SoftWoRx软件v.6.5.2(应用精度)和一个60倍物镜与CoolSNAP HQ2相机(Photometrics)控制。gydF4y2Ba
NB患者组织gydF4y2Ba
FISH分析在福尔马林固定的石蜡包埋块的4µm切片上进行。将载玻片脱蜡、脱水,在95-99℃的预处理溶液(Dako)中孵育10分钟。样品在37°C下用胃蛋白酶溶液处理2 min。对于杂化,ZytogydF4y2Ba光gydF4y2Ba规范gydF4y2BaMYCNgydF4y2Ba/2q11双色探针(ZytoVisiongydF4y2Ba)gydF4y2Ba是使用。37°C孵育过夜,然后用DAPI复染。对于每个病例,使用荧光显微镜(BX63自动荧光显微镜;奥林匹斯山)。使用SoloWeb成像系统(BioView Ltd)进行计算机文件记录和图像分析gydF4y2BaMYCNgydF4y2Ba放大(gydF4y2BaMYCNgydF4y2Ba鱼gydF4y2Ba+gydF4y2Ba)被定义为gydF4y2BaMYCNgydF4y2Ba/2q11.2 .比率>4.0,如INRG报告所述gydF4y2Ba32gydF4y2Ba.gydF4y2Ba
FISH病灶的定量gydF4y2Ba
FISH焦点的量化是使用ImageJ Find插件maxima函数在监督方式下执行的。为了量化像素强度,使用ImageJ像素强度函数。这两名GBM患者的组织FISH图像是作为先前描述的拉帕替尼GBM II期临床试验的一部分获得的。简而言之,患者在手术前口服750 mg拉帕替尼,每天两次,持续7-10天(取决于治疗间隔是否超过周末),时间达到稳定状态。在切除时采集血液和组织样本gydF4y2Ba9gydF4y2Ba.gydF4y2Ba
PC3-TetO细胞系的构建gydF4y2Ba
通过crispr - cas9介导的方法插入TetO重复序列。所用质粒pSP2-96-mer TetO-EFS-BlaR和f9 - tetra - egfp - ires - puror是H. Zhao的盛情馈赠gydF4y2Ba21gydF4y2Ba.简单地说,就是基因间区域gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaPVT1gydF4y2Ba基于其在PC3细胞ecDNA上高频扩增,被选为插入区。DNA序列从UCSC Genome Brower中检索;重复和低复杂度的DNA序列被UCSC基因组浏览器中的RepeatMasker注释和屏蔽。通过CRISPRdirect web工具设计了sgrna的引导序列gydF4y2Ba33gydF4y2Ba全基因组测序数据证实了它们的扩增。所选的导向序列构建到pSpCas9(BB)-2A-Puro (PX459)中。pSpCas9(BB)-2A-Puro(PX459)是由F. Zhang (Addgene质粒no.;62988;gydF4y2Bahttp://n2t.net/addgene:62988gydF4y2Ba;研究资源标识符:Addgene_62988)。以pSP2-96-merTetO-EFS-BlaR质粒为模板,以及含有预测切割位点上下行50个核苷酸同源臂的引物,通过PCR扩增得到修复供体。gydF4y2Ba
根据制造商的说明,使用X-tremeGENE HP转染试剂将CRISPR-Cas9质粒和96基TetO EGFP-BlastR供体转染到PC3细胞中,仅使用CRISPR-Cas9质粒或96基TetO EGFP-BlastR作为阴性对照。转染2天后,在培养基中加入芽孢霉素3 d,此时阴性对照组的大部分细胞已经死亡,而转染CRISPR-Cas9质粒和供体的组细胞存活更多。存活的细胞在96孔板中进行有限稀释,同时一直添加芽孢杆菌素。对存活的克隆进行扩增,提取其基因组DNA (gDNA),并用一对引物在插入区域两侧进行基因分型。将基因分型结果的PCR产物进行Sanger测序,以确认插入在预测的切割位点。扩增基因分型带阳性的克隆,收集中期细胞。Double FISH与FISH探针对抗Tet操作符和对抗gydF4y2BaMYCgydF4y2Ba对中期扩散进行FISH探针检测。用含有f9 - TetO - egfp - ires - puror的慢病毒感染含有TetO重复序列的PC3细胞;感染后2 d,在培养基中加入嘌呤霉素,建立稳定的细胞系,在EGFP可视化的帮助下可用于ecDNA成像。gydF4y2Ba
ecDNA的活细胞成像gydF4y2Ba
如前所述,用表达H2B-SNAPf的PiggyBac载体和超级PiggyBac转座酶(2:1)转染PC3 TetO TetR-GFP细胞系gydF4y2Ba34gydF4y2Ba.500µg ml筛选稳定转染物gydF4y2Ba−1gydF4y2BaG418,流式细胞仪分类。为便于长期延时成像,10µg mlgydF4y2Ba−1gydF4y2Ba将人纤维连接蛋白涂在Lab-Tek 8孔腔盖玻璃的每一孔中。成像前,用25 nM SNAP标记配体JF染色细胞gydF4y2Ba669gydF4y2Ba(ref。gydF4y2Ba22gydF4y2Ba)在37°C下洗涤30分钟,然后用常规介质洗涤3次,共30分钟。然后将细胞转移到37°C下含20%血清的1× optical - klear活细胞成像缓冲液中。细胞在蔡司LSM880显微镜上成像,在37°C预稳定2小时。我们用1.5%的488 nm激光和0.75%的633 nm激光照射样品,使用EC Plan-Neofluar ×40/1.30油透镜,分束器MBS 488/561/633和滤波器BP 495-550 + LP 570。z -堆叠的z步长为0.3 μ m,每次体积成像之间间隔4分钟,共16小时。gydF4y2Ba
菌落形成试验gydF4y2Ba
TR14细胞分别取自二甲亚砜(DMSO)、50 nM帕博西利或5 nM阿贝马昔利处理后60 d的细胞,并以每孔20,000个细胞的速度播种到聚d -赖氨酸涂层的24孔板中。24 h后,分别用DMSO、50 nM palbociclib或5 nM abemaciclib处理每个条件的细胞,重复3次,持续20 d。20 d时,进行结晶紫染色。简单地说,吸取细胞培养基,用PBS轻洗细胞,在4% PFA PBS中固定20分钟,用2 ml结晶紫溶液(50 mg在50 ml 10%乙醇milliq水中)染色,用PBS洗涤一次,干燥30分钟。使用ImageJ v2 (NIH)中的ColonyArea插件计算区域强度gydF4y2Ba35gydF4y2Ba.gydF4y2Ba
CellTiter-GlogydF4y2Ba
TR14细胞分别取自DMSO、50 nM palbociclib或5 nM abemaciclib处理60 d后的TR14细胞,以100 μ l的培养基以每孔500个细胞的密度播种到白色平底96孔板(Corning)中。24 h后,细胞分别用50 nM palbociclib或5 nM abemaciclib(每孔50 μ l药物溶液)处理。根据制造商的方案,在药物添加后3、6和9天,使用CellTiter-Glo发光细胞活力测定法(Promega Corporation)测定细胞活力。gydF4y2Ba
流式细胞术gydF4y2Ba
制作单细胞悬液并通过细胞过滤器以确保单细胞悬液。细胞悬浮于流式细胞仪缓冲液(Hanks’Balanced Salt Solution buffer,不含钙镁,1× GlutaMAX, 0.5% (v/v) FCS, 10 mM HEPES)中。EGFRvIII单克隆抗体806(参考;gydF4y2Ba36gydF4y2Ba)以每百万细胞1 μg的浓度加入,冰孵育1 h。细胞在流式细胞仪缓冲液中洗涤,并在抗小鼠Alexa Fluor 488抗体(1:10 00,目录号:488)缓冲液中重悬。A11017;赛默飞世尔科学公司(Thermo Fisher Scientific))在黑暗的冰上进行了45分钟的实验。再次用流式细胞仪缓冲液清洗细胞,并以每毫升约400万个细胞的速度重新悬浮在流式细胞仪缓冲液中。使用Sony SH800 FACS分拣机对细胞进行分类,并进行校准;使用二级阴性对照来通知门控。排序策略如补充图所示。gydF4y2Ba14gydF4y2Ba.gydF4y2Ba
定量聚合酶链反应gydF4y2Ba
DNA提取使用NucleoSpin组织试剂盒(machery - nagel)根据制造商的协议进行。定量PCR (qPCR)使用50 ng或1.5µl模板DNA和0.5µM引物,使用SYBR Green PCR Master Mix (Thermo Fisher Scientific)在FrameStar 96孔PCR板(4titude)中进行。在StepOnePlus实时PCR系统(赛默飞世尔科学公司)上运行和监测反应,用StepOnePlus软件v.2.3(赛默飞世尔科学公司)计算Ct值:CDK4正向:AAAGTTACCACCACACCCCC;CDK4反向:AGTGCTAAGAAAGCGGCACT。gydF4y2Ba
CRISPR-C的指导RNA设计gydF4y2Ba
sgRNAs被设计用于靶向先前报道的含有dhfr的ecDNA扩增子的末端(克隆号:PD29424h)。gydF4y2Ba17gydF4y2Ba;该片段两端的1000个碱基对(bp)序列(Chr5: 79,841,431-81,655,326;hg19)使用Integrated DNA Technologies (IDT) Custom Alt-R CRISPR-Cas9指导RNA软件(gydF4y2Bahttps://www.idtdna.com/site/order/designtool/index/CRISPR_CUSTOMgydF4y2Ba).这些序列被排序为Alt-R sgRNAs (IDT)。gydF4y2Ba
CRISPR-C诱导ecDNAgydF4y2Ba
Hap1细胞胰化,IMDM (GlutaMAX, 10% FCS)淬灭,计数,300离心gydF4y2BaggydF4y2Ba5分钟。细胞在氖重悬缓冲液中重悬至1.1 × 10前用PBS洗涤一次gydF4y2Ba7gydF4y2Ba细胞毫升gydF4y2Ba−1gydF4y2Ba.核糖核蛋白(RNP)复合物的形成过程如下:Cas9 (IDT)在Neon Resuspension Buffer中稀释至36 μM。等体积稀释的Cas9和sgRNA (44 μM, TE, pH 8.0)混合,室温孵育10-20 min。左(DHFR_H2_sgL)、右(DHFR_H2_sgR) sgRNA RNPs分别组装。然后,每个RNP 5.5 μl,电穿孔增强剂5.5 22 μl (10.8 μM;IDT)和99 μl细胞混合,使用100 μl氖移液管尖端按照制造商的说明进行电穿孔,并使用氖转染系统(Thermo Fisher Scientific)进行电穿孔,设置如下:1575 V, 10 ms脉冲宽度,3次脉冲。除使用11 μl适当的sgRNA外,采用上述方法制备单导对照。电穿孔细胞被分配到3.2 ml培养基中(适当补充±次黄嘌呤和胸苷),并分裂成24孔板的6孔。负电穿孔控制细胞在氖重悬缓冲液中重悬,然后直接添加到含有新鲜介质的井中。gydF4y2Ba
中性选择时,细胞在24孔板中培养,每2 d传代一次。传代过程中,每个孔中80-90%的细胞用于gDNA分离,其余细胞转移到含有新鲜培养基的新板中。对于次黄嘌呤和胸腺嘧啶补充井,次黄嘌呤和胸腺嘧啶补充(100X,目录编号:11067030;Gibco)加入到最终浓度100 μΜ次黄嘌呤和16 μΜ胸苷中。gydF4y2Ba
阳性选择:电穿孔后3 d,细胞传代至12孔板,收集第3天时间点。电穿孔后4 d,将细胞置于含有指示浓度甲氨蝶呤(Calbiochem)的培养基中。每2-3天更换一次培养基,在70-80%合流时传代细胞。在甲氨蝶呤孵育14天后(电穿孔后18天)收集细胞。甲氨蝶呤处理井的DMSO最终浓度为0.1%。gydF4y2Ba
在指定的时间点收集细胞:用每1ml预温PBS (Gibco)清洗细胞,然后加入100 μl TrypLE Express (Thermo Fisher Scientific),在37℃孵育5-10分钟。用800 μl IMDM (GlutaMAX, 10% FCS)淬灭TrypLE, 300 μl细胞悬液成粒gydF4y2BaggydF4y2Ba在4°C下加热5分钟。上清液被丢弃,细胞颗粒储存在−80°C。gydF4y2Ba
CRISPR-C原理图是用BioRender创建的。gydF4y2Ba
ddPCR测定ecDNA或染色体瘢痕频率gydF4y2Ba
gDNA使用DNeasy色谱柱(QIAGEN)根据制造商的说明进行分离,包括在蛋白酶K消化步骤中在56°C下孵育10分钟;DNA用100 μl EB缓冲液洗脱。gydF4y2Ba
使用IDT PrimerQuest软件设计ecDNA连接、染色体瘢痕连接和甘油醛3-磷酸脱氢酶(GAPDH)扩增子(gydF4y2Bahttps://www.idtdna.com/PrimerQuest/Home/IndexgydF4y2Ba).使用双淬灭探针(IDT): fam标记探针用于ecDNA和染色体瘢痕连接扩增子,以促进利用hex标记探针与GAPDH扩增子的多路复用。所有探针和引物序列可在补充gydF4y2Ba信息gydF4y2Ba.液滴是使用液滴生成油用于探针,DG8卡带,DG8垫圈和QX200液滴发生器(Bio-Rad实验室);使用ddPCR Supermix for Probes (Bio-Rad Laboratories)进行扩增。ddPCR Supermix扩增反应根据制造商的规范(Bio-Rad Laboratories)进行。在20 μl的反应中使用约60 ng gDNA,最终引物浓度为900 nM(每个引物225 nM), 125 nM FAM探针和125 nM HEX探针。根据制造商的方案(Bio-Rad Laboratories),将反应分成液滴进行扩增。液滴转移到96孔PCR板上,使用PX1 PCR板封口机(Bio-Rad Laboratories)进行热封。液滴在以下循环条件下被放大:95°C 10分钟,40次循环(94°C 30秒,56.1°C 60秒),98°C 10分钟。热循环后,使用QX200液滴数字PCR系统(Bio-Rad实验室)逐个扫描液滴。每个荧光通道(HEX, FAM)中的阳性和阴性液滴是根据荧光振幅来区分的,使用的是一个全局阈值,该阈值是由荧光探针(负液滴)的不完全猝灭所产生的最小本征荧光信号与具有放大模板的液滴中裂解探针的强荧光信号相比所设置的。 The frequency of ecDNA or chromosomal scar was calculated by dividing their measured concentration by the concentration of the GAPDH amplicon.
单细胞ecDNA分离模式的定量gydF4y2Ba
在不同的ecDNA分离模型下,我们通过c++实现的随机计算机模拟,生成了单细胞分裂后ecDNA拷贝数分数的理论预期分布。简单地说,单个细胞由随机数量的ecDNA副本启动gydF4y2BangydF4y2Ba,由均匀分布得出gydF4y2BaUgydF4y2Ba(20200)。2 . EcDNA扩增gydF4y2BangydF4y2BaecDNA副本在双名试验后在两个子细胞之间分离gydF4y2BaBgydF4y2Ba(2gydF4y2BangydF4y2Ba,gydF4y2BapgydF4y2Ba)具有偏析概率gydF4y2BapgydF4y2Ba.在这种情况下,gydF4y2BapgydF4y2Ba= 1/2对应随机分离和gydF4y2BapgydF4y2Ba> 1/2到一个有偏的随机偏析。这将产生两个具有ecDNA拷贝数的子细胞gydF4y2BangydF4y2Ba1gydF4y2Ba≈gydF4y2BaBgydF4y2Ba(2gydF4y2BangydF4y2Ba,gydF4y2BapgydF4y2Ba),gydF4y2BangydF4y2Ba2gydF4y2Ba=gydF4y2BangydF4y2Ba−gydF4y2BangydF4y2Ba1gydF4y2Ba.分离出来的ecDNA,gydF4y2BafgydF4y2Ba,则计算为:gydF4y2Ba
\ (f = \压裂{{n_1}} {{n_1 +甲烷}}\,{{{\ mathrm{和}}}}\,₂= \压裂{{甲烷}}{{n_1 +甲烷}}\)。gydF4y2Ba
10 .迭代过程gydF4y2Ba7gydF4y2Ba的期望分布gydF4y2BafgydF4y2Ba如图所示。gydF4y2Ba1 cgydF4y2Ba.类似地,我们得到了一个期望分布gydF4y2BafgydF4y2Ba染色体遗传模式。为了完美的染色体分离,我们有gydF4y2BafgydF4y2Ba1gydF4y2Ba=gydF4y2BafgydF4y2Ba2gydF4y2Ba= 1/2。为了允许错误分离,我们引入了一个概率gydF4y2BaugydF4y2Ba= 0.05,使得gydF4y2BangydF4y2Ba1gydF4y2Ba=gydF4y2BangydF4y2Ba±1和gydF4y2BangydF4y2Ba2gydF4y2Ba=gydF4y2BangydF4y2Ba−ngydF4y2Ba1gydF4y2Ba.我们使用Kolmogorov-Smirnov统计量来比较理论预期和实验观察到的ecDNA拷贝数分数在这些不同情况下的分布。gydF4y2Ba
ecDNA种群动态的随机模拟gydF4y2Ba
我们在c++中实现了基于个体的ecDNA种群动态随机计算机模拟。对于每个细胞,通过模拟记录ecDNA拷贝的确切数量。使用吉莱斯皮算法随机选择适合增殖的细胞。模拟开始时只有一个单元携带gydF4y2BangydF4y2Ba0gydF4y2BaecDNA的副本。无ecDNA的细胞增殖率设为gydF4y2BargydF4y2Ba−gydF4y2Ba= 1(时间以代为单位)。具有ecDNA的细胞的适应度效应与增殖率相对应gydF4y2BargydF4y2Ba+gydF4y2Ba=。在这个例子中,gydF4y2Ba年代gydF4y2Ba> 1表示适应度优势,0
采样和分辨率限制gydF4y2Ba
我们进行了一个硅试验,以测试我们从不同大小的样本子集中重建真实ecDNA拷贝数分布的能力。我们从2 × 10构建了模拟ecDNA拷贝数分布gydF4y2Ba6gydF4y2Ba细胞使用我们的随机模拟。然后,我们进行了25、50、100和500个细胞的500个随机样本,重建了样本ecDNA拷贝数分布,并使用Kolmogorov-Smirnov统计量比较了与真实拷贝数分布的相似性。随着样本量的增加,分布收敛到真实的分布,相对较小的100-500个细胞的样本足以重建真实的底层ecDNA拷贝数分布。gydF4y2Ba
ecDNA动力学的数学描述gydF4y2Ba
无选择的确定性双种群模型gydF4y2Ba
在模型的最简单表示中,我们区分了携带或不携带ecDNA副本的细胞。我们称具有ecDNA副本的细胞为gydF4y2BaNgydF4y2Ba+gydF4y2Ba(gydF4y2BatgydF4y2Ba)和没有ecDNA副本的细胞gydF4y2BaNgydF4y2Ba−gydF4y2Ba(gydF4y2BatgydF4y2Ba).我们可以写出这些单元格在时间上的变化gydF4y2BatgydF4y2Ba:gydF4y2Ba
在哪里gydF4y2Ba\ \ upsilon \离开({N ^ +左(t \右)}\ \)\)gydF4y2Ba对应随机完全不对称ecDNA分离的损失率。我们发现携带ecDNA的细胞gydF4y2BafgydF4y2Ba+gydF4y2Ba(gydF4y2BatgydF4y2Ba)在指数增长的人口中:gydF4y2Ba
携带ecDNA的细胞比例约为1/gydF4y2BatgydF4y2BaecDNA是否为中性。因此,中性ecDNA的拷贝只存在于肿瘤细胞的一小部分亚群中。gydF4y2Ba
带有选择的确定性双种群模型gydF4y2Ba
上面的方程可以修改,以考虑适应度优势gydF4y2Ba年代gydF4y2Ba携带ecDNA的细胞> 1:gydF4y2Ba
这组方程的解是:gydF4y2Ba
在阳性选择的情况下,具有ecDNA的细胞比例为gydF4y2Ba\(f^ + \to 1\)gydF4y2Ba.在足够长的时间内,肿瘤将由携带ecDNA的细胞主导。gydF4y2Ba
中性ecDNA的随机动力学gydF4y2Ba
我们还对不断增长的种群中ecDNA动力学的随机特性感兴趣。因此,我们转移到一个更细粒度的图像,并考虑单元格的数量gydF4y2BaNgydF4y2BakgydF4y2Ba(gydF4y2BatgydF4y2Ba),gydF4y2BakgydF4y2BaecDNA的拷贝gydF4y2BatgydF4y2Ba.ecDNA中性拷贝的动力学方程为:gydF4y2Ba
使用单元格密度更方便gydF4y2BaρgydF4y2Ba而不是细胞号gydF4y2BaNgydF4y2Ba.将上述方程归一化,我们得到密度gydF4y2BaρgydF4y2BakgydF4y2Ba细胞的gydF4y2BakgydF4y2BaecDNA副本:gydF4y2Ba
中性ecDNA拷贝的矩动力学gydF4y2Ba
用上面的公式求细胞的密度gydF4y2BakgydF4y2BaecDNA副本,我们可以计算出潜在概率密度函数的矩。一般而言,gydF4y2BalgydF4y2Ba矩可通过:gydF4y2Ba
可以证明,所有矩都与gydF4y2Ba\(M^{\左(l \右)}(t)\约t^{l - 1}\)gydF4y2Ba我们明确地发现对于前两个时刻gydF4y2Ba
\ (M ^ {(1)} = 1 \, {{{\ mathrm{和}}}}\,M ^{左(2 \)\}\离开(t \右)= t \)。gydF4y2Ba
在指数增长的种群中,中性ecDNA拷贝的平均ecDNA拷贝数是恒定的。ecDNA拷贝数的方差随时间线性增加。gydF4y2Ba
正选择下ecDNA的随机动力学gydF4y2Ba
上述公式可推广,以适应正选择(gydF4y2Ba年代gydF4y2Ba> 1)用于ecDNA拷贝。细胞密度的动态方程集为:gydF4y2Ba
这些方程的通解很有挑战性。尽管如此,重要的量,例如,矩动力学和缩放行为可以显式计算。gydF4y2Ba
正选择下ecDNA的矩动力学gydF4y2Ba
由上述方程直接推导出力矩动力学的广义方程。我们有:gydF4y2Ba
这首先意味着gydF4y2Ba\ \(压裂{{\部分M ^{\离开(1 \右)}(t)}}{{\部分t}} = (s - 1) \ρ_0M ^{\离开(1 \右)}(t) \)gydF4y2Ba,这个问题可以在第一时间解决:gydF4y2Ba
类似地,第二个弯矩的动力学方程为gydF4y2Ba\ \(压裂{{\部分M ^{\左(右2 \)}(t)}}{{\部分t}} = M ^{\离开(1 \右)}\离开(t \右)+ (s - 1) \ρ_0M ^{\左(右2 \)}(t) \)gydF4y2Ba我们发现gydF4y2Ba
最初,第一时刻呈指数增长。然而,随着平均拷贝数的增加,细胞过渡到无ecDNA状态的速率逐渐降低,平均ecDNA拷贝数的增加逐渐趋于平稳。请注意,gydF4y2Ba年代gydF4y2Ba= 1,我们恢复了之前的中性ecDNA扩增时刻的结果。gydF4y2Ba
利用CRISPR-Cas9核糖核蛋白进行基因组编辑gydF4y2Ba
使用Alt-R s.p. Cas9 Nuclease V3(目录号:col320 - dm细胞)对细胞进行基因组编辑。1081058;IDT)与sgRNA (Synthego)配合,根据Synthego RNP转染协议,使用Neon转染系统(目录编号:MPK5000;赛默飞世尔科学公司)。简单地说,每10 μl反应10 pmol Cas9蛋白和60 pmol sgRNA在Neon Buffer R中室温孵育10 min。用1× PBS清洗细胞,重悬于Buffer R中,每10 μl将20万个细胞与预培养的RNP复合物混合。根据制造商的协议,使用以下设置对细胞混合物进行电穿孔:1700 V, 20 ms, 1个脉冲。细胞培养10 d;在第3、6和10天收集细胞计数和ecDNA拷贝数数据。 To estimate the ecDNA copy numbers, we performed metaphase chromosome spreading followed by FISH as described above. All sgRNA sequences are in Supplementary Table3 gydF4y2Ba.gydF4y2Ba
鱼探针gydF4y2Ba
如下探针用于FISH,如所示:ZytogydF4y2Ba光gydF4y2BaSPEC CDK4/CEN 12双色探针(ZytoVision);ZytogydF4y2Ba光gydF4y2BaSPEC MYCN/2q11双色探针(ZytoVision);Empire Genomics EGFR FISH探针;帝国基因组公司MYC FISH探针;Empire Genomics FGFR2 FISH探针;帝国基因组公司CDK4 FISH探针;帝国基因组公司MYCN FISH探针。gydF4y2Ba
抗体gydF4y2Ba
以下抗体以1:10 - 1:200的浓度用于免疫荧光,以1:10 00的浓度用于免疫印迹(除非另有特殊说明)gydF4y2Ba方法gydF4y2BaAurora B Polyclonal Antibody(目录编号:;a300 - 431 a;赛默飞世尔科技公司;EGFRvIII单克隆抗体806(参考;gydF4y2Ba36gydF4y2Ba);防鼠Alexa Fluor 488。gydF4y2Ba
统计和再现性gydF4y2Ba
分析拷贝数分布的生物实验的样本量是由随机模拟决定的。研究人员并没有对实验组视而不见。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究未产生任何新的核酸测序数据。所有数据和材料,包括细胞结构,将在通信作者的合理要求下提供。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
用于随机ecDNA分离的随机模拟的代码可在GitHub-BenWernerScripts/ ecDNA - dynamics。gydF4y2Ba
参考文献gydF4y2Ba
梅洛,L. M. F.,佩珀,J. W.,里德,B. J.和马利,C. C.癌症作为一个进化和生态过程。gydF4y2BaNat. Rev. CancergydF4y2Ba6gydF4y2Ba, 924-935(2006)。gydF4y2Ba
格里夫斯,M. & Maley, C. C.肿瘤克隆进化。gydF4y2Ba自然gydF4y2Ba481gydF4y2Ba, 306-313(2012)。gydF4y2Ba
McGranahan, N. & Swanton, C.克隆异质性和肿瘤进化:过去、现在和未来。gydF4y2Ba细胞gydF4y2Ba168gydF4y2Ba, 613-628(2017)。gydF4y2Ba
McGranahan, N. & Swanton, C.肿瘤内异质性在癌症进化中的生物学和治疗影响。gydF4y2Ba癌症细胞gydF4y2Ba27gydF4y2Ba, 15-26(2015)。gydF4y2Ba
Abbosh, C.等人。系统发育ctDNA分析描述早期肺癌的进化。gydF4y2Ba自然gydF4y2Ba545gydF4y2Ba, 446-451(2017)。gydF4y2Ba
Barthel, f.p.等。成人弥漫性胶质瘤的纵向分子轨迹。gydF4y2Ba自然gydF4y2Ba576gydF4y2Ba, 112-120(2019)。gydF4y2Ba
贾马尔-汉贾尼,等人。追踪非小细胞肺癌的演变。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba376gydF4y2Ba, 2109-2121(2017)。gydF4y2Ba
Burrell, R. A., McGranahan, N., Bartek, J. & Swanton, C.癌症进化中遗传异质性的原因和后果。gydF4y2Ba自然gydF4y2Ba501gydF4y2Ba, 338-345(2013)。gydF4y2Ba
内桑森,d.a.等人。通过动态调节染色体外突变EGFR DNA介导的靶向治疗耐药。gydF4y2Ba科学gydF4y2Ba343gydF4y2Ba, 72-76(2014)。gydF4y2Ba
Furnari, f.b., Cloughesy, t.f., Cavenee, W. K. & Mischel, p.s.胶质母细胞瘤中表皮生长因子受体信号网络的异质性。gydF4y2BaNat. Rev. CancergydF4y2Ba15gydF4y2Ba, 302-310(2015)。gydF4y2Ba
沃特金斯,t.b.k.等。肿瘤进化中普遍存在的染色体不稳定性和核型顺序。gydF4y2Ba自然gydF4y2Ba587gydF4y2Ba, 126-132(2020)。gydF4y2Ba
金,C.等人。单细胞测序显示三阴性乳腺癌的耐药演变。gydF4y2Ba细胞gydF4y2Ba173gydF4y2Ba, 879 - 893。e13(2018).gydF4y2Ba
Vasan, N. Baselga, J. & Hyman, D. M.关于癌症耐药性的观点。gydF4y2Ba自然gydF4y2Ba575gydF4y2Ba, 299-309(2019)。gydF4y2Ba
Kim, H.等人。染色体外DNA与多种癌症的癌基因扩增和不良预后相关。gydF4y2BaNat,麝猫。gydF4y2Ba52gydF4y2Ba, 891-897(2020)。gydF4y2Ba
特纳,K. M.等。染色体外癌基因扩增驱动肿瘤进化和遗传异质性。gydF4y2Ba自然gydF4y2Ba543gydF4y2Ba, 122-125(2017)。gydF4y2Ba
伦德伯格等人。的二项式有丝分裂分离gydF4y2BaMYCNgydF4y2Ba在成神经细胞瘤中携带双分钟,说明随机性在癌基因扩增中的作用。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba3 gydF4y2Ba, e3099(2008)。gydF4y2Ba
Shoshani, O.等人。嗜色菌病驱动癌症基因扩增的进化。gydF4y2Ba自然gydF4y2Ba591gydF4y2Ba, 137-141(2021)。gydF4y2Ba
Yi, E.等。活细胞成像显示癌症中染色体外DNA元件的不均匀分离和转录活性的染色体外DNA中心。gydF4y2Ba癌症。gydF4y2Ba12gydF4y2Ba, 468-483(2022)。gydF4y2Ba
Thompson, S. L. & Compton, D. A.人类细胞中的染色体误分离是通过特定类型的动丝-微管附着错误引起的。gydF4y2Ba国家科学院学报美国gydF4y2Ba108gydF4y2Ba, 17974-17978(2011)。gydF4y2Ba
富勒,B. G.等。极光B在后期的中间区激活产生细胞内磷酸化梯度。gydF4y2Ba自然gydF4y2Ba453gydF4y2Ba, 1132-1136(2008)。gydF4y2Ba
塔桑,我,等人。CRISPR/ cas9介导的优化TetO重复敲入用于内源性位点的活细胞成像。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e100(2018)。gydF4y2Ba
格林,J. B.等人。红移罗丹明染料优化与功能化的一般方法。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 815-821(2020)。gydF4y2Ba
杰林格,M.等人。多区测序显示肿瘤内异质性和分支进化。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba366gydF4y2Ba, 883-892(2012)。gydF4y2Ba
Qazi, m.a.等人。瘤内异质性:人类胶质母细胞瘤耐药和复发的途径。gydF4y2Ba安。肿瘤防治杂志。gydF4y2Ba28gydF4y2Ba, 1448-1456(2017)。gydF4y2Ba
数值模拟耦合化学反应随机时间演化的一般方法。gydF4y2Baj .第一版。理论物理。gydF4y2Ba22gydF4y2Ba, 403-434(1976)。gydF4y2Ba
Møller, H. D.等。CRISPR- c:基因和染色体在人类细胞中的循环。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e131(2018)。gydF4y2Ba
考夫曼,R. J.,布朗,P. C. & Schimke, R. T.在不稳定的甲氨蝶呤耐药细胞中扩增的二氢叶酸还原酶基因与双分钟染色体相关。gydF4y2Ba国家科学院学报美国gydF4y2Ba76gydF4y2Ba, 5669-5673(1979)。gydF4y2Ba
吴,S.等。环状ecDNA促进可接近染色质和高癌基因表达。gydF4y2Ba自然gydF4y2Ba575gydF4y2Ba, 699-703(2019)。gydF4y2Ba
Paffhausen, T., Schwab, M. & Westermann, F. TargetedgydF4y2BaMYCNgydF4y2Ba表达影响化疗药物在成神经细胞瘤细胞中的细胞毒潜能。gydF4y2Ba癌症。gydF4y2Ba250gydF4y2Ba, 17-24(2007)。gydF4y2Ba
Cen, L. et al. p16-Cdk4-Rb轴控制胶质母细胞瘤异种移植细胞对周期蛋白依赖性激酶抑制剂PD0332991的敏感性。gydF4y2Ba神经肿瘤防治杂志。gydF4y2Ba14gydF4y2Ba, 870-881(2012)。gydF4y2Ba
Hung, K. L.等人,ecDNA中心驱动协同分子间癌基因表达。gydF4y2Ba自然gydF4y2Ba600gydF4y2Ba, 731-736(2021)。gydF4y2Ba
安布罗斯,P. F.等人。成神经细胞瘤分子诊断的国际共识:来自国际成神经细胞瘤风险组(INRG)生物学委员会的报告。gydF4y2BaBr。j .癌症gydF4y2BaOne hundred.gydF4y2Ba, 1471-1482(2009)。gydF4y2Ba
Naito, Y., Hino, K., Bono, H.和Ui-Tei, K. CRISPRdirect:设计具有较少脱靶位点的CRISPR/Cas引导RNA的软件。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 1120-1123(2015)。gydF4y2Ba
谢,等。3D ATAC-PALM:可访问基因组的超分辨率成像。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 430-436(2020)。gydF4y2Ba
Guzmán, C., Bagga, M., Kaur, A., Westermarck, J. & Abankwa, D. ColonyArea:一个ImageJ插件,用于自动量化克隆分析中的菌落形成。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, e92444(2014)。gydF4y2Ba
Jungbluth, a.a.等人。一种单克隆抗体,识别人表皮生长因子受体扩增/过表达的人类癌症。gydF4y2Ba国家科学院学报美国gydF4y2BaOne hundred.gydF4y2Ba, 639-644(2003)。gydF4y2Ba
确认gydF4y2Ba
eDyNAmiC研究是由癌症大挑战(批准no。在英国癌症研究中心和国家癌症研究所的支持下,CGCSDF-2021\100007)。这项研究得到了美国国家脑肿瘤学会和美国国立卫生研究院(NIH)的资助。R01-CA238349, p.s.m.h.y.c是霍华德休斯医学研究所的研究员,也得到了NIH no。R35-CA209919。巴茨慈善讲座MGU045和UKRI未来领袖奖学金支持B.W. UCSD显微镜核心是由国家神经疾病和中风研究所(no。NS047101)。NIH资助号U24CA264379和1R01GM114362资助了V.B.。A.P. Giannini基金会资助了J.C.R.。该项目已获得欧洲研究委员会(ERC)在欧盟地平线2020研究与创新计划(批准号:398299703)下的资助。949172)。 The Cancer Prevention and Research Institute of Texas (no. RR210034) supported S.W. We thank C. Zhang from the Howard Chang Laboratory for providing technical assistance with the CRISPR-C experiments. C.S. is a Royal Society Napier Research Professor (no. RSRP\R\210001). This work was supported by the Francis Crick Institute, which receives its core funding from Cancer Research UK (no. FC001169), the UK Medical Research Council (no. FC001169) and the Wellcome Trust (no. FC001169). This research was funded in whole, or in part, by the Wellcome Trust (no. FC001169). For the purpose of open access, the authors applied a CC BY public copyright licence to any author-accepted manuscript version arising from this submission. C.S. is funded by Cancer Research UK (TRACERx (no. C11496/A17786), PEACE (C416/A21999) and CRUK Cancer Immunotherapy Catalyst Network), Cancer Research UK Lung Cancer Centre of Excellence (no. C11496/A30025), the Rosetrees Trust, Butterfield and Stoneygate Trusts, NovoNordisk Foundation (ID16584), Royal Society Professorship Enhancement Award (no. RP/EA/180007), the National Institute for Health Research Biomedical Research Centre at University College London Hospitals, the Cancer Research UK-University College London Centre, Experimental Cancer Medicine Centre and the Breast Cancer Research Foundation (no. BCRF 20-157). This work was supported by a Stand Up To Cancer (SU2C)‐LUNGevity-American Lung Association Lung Cancer Interception Dream Team Translational Research Grant (grant no. SU2C-AACR-DT23-17 to S. M. Dubinett and A. E. Spira). SU2C is a division of the Entertainment Industry Foundation. Research grants are administered by the American Association for Cancer Research, the Scientific Partner of SU2C. C.S. is in receipt of an ERC Advanced Grant (PROTEUS) from the European Research Council under the European Union’s Horizon 2020 research and innovation programme (grant no. 835297). Funding from the Vienna Science and Technology Fund (no. LS18-111), Austrian Science Fund (nos. I4162 and P35841-B) and St. Anna Kinderkrebsforschung supported S.T.-M. We thank D. Nathanson for provision of clinical data. We thank A. Desai for usage of equipment and experimental advice.
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
j.t.l., v.b., B.W.和P.S.M.构思了这个项目。J.T.L J.C.R。,C.Y.C L.X, j.t., K.L.H, K.E.Y,问:,M.L.E、表面张力和M.B.执行这个实验。y.p., w.h., V.B.和B.W.进行了计算建模。J.T.L J.C.R。,C.Y.C Y.P, L.X, j.t., K.L.H, K.E.Y, Q.S.和采用分析了数据,指导下白雪,王秋森,Z.L, W.H。H.Y.C, V.B, A.G.H, B.W.和P.S.M. J.T.L, W.H。V.B B.W.和P.S.M.手稿反馈所有的作者写道。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
P.S.M.是无边生物公司的联合创始人。他拥有股权,并担任科学顾问委员会主席,为此他获得了报酬。V.B.是Boundless Bio的联合创始人、顾问、科学顾问委员会成员,并拥有股权。和Abterra生物科学公司。这一安排的条款已根据其利益冲突政策进行了审查和批准。H.Y.C.是Accent Therapeutics、Boundless Bio、Cartography Biosciences、Orbital Therapeutics的联合创始人,也是10X Genomics、Arsenal Biosciences和Spring Discovery的顾问。完成这项工作后,J.T.L.受雇于Boundless Bio。C.S.感谢阿斯利康、勃林格-殷格翰、Bristol Myers Squibb、辉瑞、Roche-Ventana、Invitae(以前是ArcherDX,在最小残留疾病测序技术方面的合作)和Ono Pharmaceutical的资助。他是阿斯利康咨询委员会成员和阿斯利康美人鱼1和2临床试验的首席研究员,也是NHS-Galleri试验的首席研究员。他曾为安进、阿斯利康、辉瑞、诺华、GSK、MSD、Bristol Myers Squibb、Illumina、Genentech、Roche- ventana、GRAIL、Medicxi、Metabomed、Bicycle Therapeutics、罗氏创新中心上海和Sarah Cannon研究所提供咨询。 C.S. had stock options in ApoGen Biotechnologies and GRAIL until June 2021, has currently stock options in Epic Bioscience and Bicycle Therapeutics, and has stock options and is a cofounder of Achilles Therapeutics. C.S. holds patents relating to assay technology to detect tumor recurrence (PCT/GB2017/053289), targeting neoantigens (PCT/EP2016/059401), identifying patent response to immune checkpoint blockade (PCT/EP2016/071471), determining human leukocyte antigen loss of heterozygosity (PCT/GB2018/052004), predicting survival rates of patients with cancer (PCT/GB2020/050221), identifying patients who respond to cancer treatment (PCT/GB2018/051912), a US patent relating to detecting tumor mutations (PCT/US2017/28013), methods for lung cancer detection (US20190106751A1) and both European and US patents related to identifying insertion/deletion mutation targets (PCT/GB2018/051892). The other authors declare no competing interests.
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然遗传学gydF4y2Ba感谢Andrew Futreal和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展数据gydF4y2Ba
扩展数据图1利用数学建模、计算机模拟、细胞系和患者数据开发和测试ecDNA行为规则的策略概述。gydF4y2Ba
a,用于开发和测试ecDNA的实验行为、患者行为和理论行为的策略概述。gydF4y2Ba
扩展数据图2跨癌症类型和扩增癌基因的癌细胞系中的ecDNA分离。gydF4y2Ba
a,图中用于量化分离动力学的细胞系的代表性中期FISH图像。gydF4y2Ba1gydF4y2Ba比例尺为10μm。b,图中分析的子细胞相同。gydF4y2Ba1 cgydF4y2Ba通过量化每个子细胞中FISH信号的像素强度,作为ecDNA数的代理来分析。理论预测(虚线)与观察结果(直方图)之间的一致性由KS检验显示。c,分位数-分位数图,比较测量的ecDNA分离分数和模拟的随机ecDNA分离的分布。gydF4y2Ba
扩展数据图3通过将Tet-O阵列插入PC3细胞的ecDNA来追踪ecDNA的活细胞。gydF4y2Ba
a, PC3亲本细胞系和PC3- teto细胞系的代表性图像显示两者均有广泛的MYC扩增。PC3-TetO在多个ecDNA体上也表现出明显的TetO FISH信号。b, 96位TetO重复序列的PCR扩增。c,从PC3-TetO细胞分离的DNA中PCR扩增96个mer TetO重复序列,确认插入。d, PC3-TetO细胞PCR扩增产物的Sanger测序。左、右连接均在插入部位进行同源重组修复。gydF4y2Ba
扩展数据图4细胞系中ecDNA拷贝数分布。gydF4y2Ba
a,在ecDNA随机分离后,比较细胞系群体与模拟群体中ecDNA拷贝数分布的分位数-分位数图。gydF4y2Ba
图5患者肿瘤中的ecDNA异质性。gydF4y2Ba
a, MYCN扩增的成神经细胞瘤(NB)患者肿瘤组织间期FISH评估的ecDNA拷贝数直方图。b,比较来自患者组织FISH的测量ecDNA数量和来自模拟肿瘤生长的模拟ecDNA数量的ecDNA拷贝数的分位数-分位数图。gydF4y2Ba
扩展数据图6 ecDNA动态响应治疗。gydF4y2Ba
a,中性选择和阳性选择下ecDNA拷贝数、频率和拷贝数趋势的示意图。b,模拟显示ecDNA在来自一个ecDNA阳性选择或中性选择的ecDNA+细胞的群体中流行。正选择s = 3;中性s = 1。c,定量患者和细胞系样本中的ecDNA频率,根据扩增的癌基因分组,并根据所示的不同选择水平进行比较。d,在CRISPR切割col320 - dm基因组区域(ecDNA上或外)后,在第6天和第10天定量ecDNA数量。显示明确的证据,选择ecDNA的拷贝数严重下降时,目标和指示,拷贝数开始返回到初始水平。注意ecDNA_MYC在第6天的生长受到严重限制,只能识别6个中期并成像。来自两面t检验的p值。gydF4y2Ba
扩展数据图7 ecDNA动态响应治疗。gydF4y2Ba
a,等基因GBM39细胞系中期扩散FISH的代表性图像。b,随机ecDNA遗传(GBM39-EC)或规范染色体遗传(GBM39-HSR)下Shannon多样性的随机模拟。每种条件进行100次随机模拟。箱形图显示的线位于中位数,方框范围为25至75百分位,晶须延伸至最极端值。c,流式细胞术分析EGFR蛋白在等基因GBM39-EC和GBM39-HSR细胞系中的表达,显示出与拷贝数相似的异质性模式。X-CV量化了两个样本的%变异系数。我们用FSC-A/SSC-A定位主要细胞群,用FSC-H/FSC-W对单细胞进行门控。我们使用负控制样本(仅次要)来调整Alexa-Fluor488通道的电压。d,用阿贝西库或帕博西库治疗60天的TR14细胞的代表性图像。绿色为CDK4 FISH信号,红色为CEN12对照FISH探针。 e, Quantification of experiment described in d shows significant shift in CDK4 ecDNA copy number distribution under both drug conditions. f, Quantification of EGFR ecDNA in GBM39-EC cells after short-term treatment with erlotinib shows rapid change in ecDNA copy number distribution. Lines indicate medians. P values calculated using Mann-Whitney tests. * p ≤ 0.05; **** p ≤ 0.0001. Scale bars 10 µm.
扩展数据图8 ecDNA动力学与耐药形成相关。gydF4y2Ba
a,使用palbociclib或abemaciclib治疗长期耐药的TR14细胞群体,表现出对治疗的耐药性。b,使用帕博西布或阿贝西布治疗对治疗显示耐药性的长期耐药TR14细胞群体。c,通过qPCR验证CDK4增加的ecDNA拷贝数。(a-c)数据以3次技术重复的平均值+/−标准差表示。d, TR14细胞在产生耐药或不产生耐药后,用palbociclib或abemaciclib重新挑战(DMSO),结晶紫染色。e,在CDK4抑制剂治疗60天的人群中,d显示耐药性的定量。数据以3次技术重复的平均值+/−标准偏差表示。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充图1-14。gydF4y2Ba
补充视频1gydF4y2Ba
延时视频显示ecDNA的不平等分离。gydF4y2Ba
补充表3gydF4y2Ba
PCR和CRISPR寡核苷酸序列gydF4y2Ba
源数据gydF4y2Ba
图1 .来源数据gydF4y2Ba
未加工的凝胶。gydF4y2Ba
图2 .来源数据gydF4y2Ba
未加工的凝胶。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
兰格,j.t.,罗斯,j.c.,陈志勇gydF4y2Baet al。gydF4y2Ba人类癌症中染色体外DNA的进化动力学。gydF4y2BaNat麝猫gydF4y2Ba54gydF4y2Ba, 1527-1533(2022)。https://doi.org/10.1038/s41588-022-01177-xgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41588-022-01177-xgydF4y2Ba