摘要
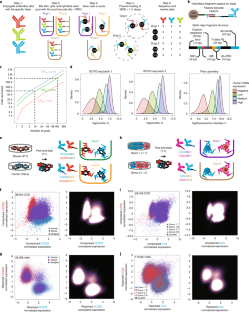
用于标记细胞表面分子的dna条形码抗体的开发使使用基于液滴的单细胞测序(dsc-seq)能够同时分析数千个细胞的蛋白质丰度。与流式和大规模细胞术相比,当前基于dsc-seq的工作流程每细胞成本高,阻碍了它们在临床应用和大规模合并筛查中的应用。在这里,我们介绍了SCITO-seq,一种使用夹板寡核苷酸(oligos)的工作流程,使dna条形码抗体的组合索引dsc-seq超过105每反应使用商业微流体的细胞。通过将样本条形码编码到夹板寡核苷酸中,我们证明了复用SCITO-seq可以产生与大规模细胞术相当的细胞组成和表面蛋白表达的可重复估计。我们进一步展示了两种改进的夹板寡核苷酸设计,扩展了SCITO-seq,以实现与商业dna条形码抗体的兼容性,并同时表达来自同一细胞的转录组和表面蛋白。这些结果表明,SCITO-seq是一种灵活的、超高通量的单细胞蛋白质测序和多模态分析平台。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
在诊断应用中使用dna条形码抗体拆分池测序定量单细胞上的蛋白质丰度
科学报告开放获取2022年1月18日
访问选项
订阅《自然》+
立即在线访问《自然》和其他55种《自然》杂志
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
99.00美元
每期只要8.25美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。
数据可用性
本项目产生的单细胞测序数据已存入基因表达Omnibus,并附有登录代码GSE147808.经处理后的数据亦可于网站(https://github.com/yelabucsf/SCITO-seq)和教程。
代码的可用性
所有用于模拟和生成图形的代码可在以下网站找到(https://github.com/yelabucsf/SCITO-seq,https://doi.org/10.5281/zenodo.4988182).另一个与工程项目有关的计算费用网站,亦可浏览https://yelabtools.herokuapp.com/scSeqCostCalc/scito.html).
参考文献
马科斯科,e.z.等。使用纳米升液滴对单个细胞进行高度平行的全基因组表达分析。细胞161, 1202-1214(2015)。
克莱因等人。用于单细胞转录组学的液滴条形码应用于胚胎干细胞。细胞161, 1187-1201(2015)。
布恩罗斯特罗,J. D.等。单细胞染色质可及性揭示了调控变异的原理。自然523, 486-490(2015)。
斯托克修斯等人。单细胞中同时测定表位和转录组。Nat方法。14, 865-868(2017)。
Shahi, P., Kim, S. C., Haliburton, J. R., Gartner, Z. J. & Abate, A. R. Abseq:用液滴微流控条形码进行超高通量单细胞蛋白质分析。科学。代表。7, 44447(2017)。
Gerlach, J. P.等。来自固定单细胞的胞内(磷酸化)蛋白和转录组的联合定量。科学。代表。9, 1469(2019)。
彼得森,v.m.等人。单细胞蛋白质和转录物的多重定量。生物科技Nat。》。35, 936-939(2017)。
班杜拉,d.r.等。海量细胞术:基于感应耦合等离子飞行时间质谱的实时单细胞多靶点免疫分析技术。分析的化学。81, 6813-6822(2009)。
斯皮策,M. H.诺兰,G. P.海量细胞术:单细胞,多种特征。细胞165, 780-791(2016)。
斯旺森,E.等。同时使用TEA-seq对转录本、表位和染色质可及性进行三模态单细胞测量。eLife10, e63632(2021)。
米米图,e.p.。et al。可扩展的,单细胞染色质可达性,基因表达和蛋白质水平的多模式分析。生物科技Nat。》。(2021)https://doi.org/10.1038/s41587-021-00927-2
Kang, h.m.等。利用自然遗传变异的多液滴单细胞rna测序。生物科技Nat。》。36, 89-94(2018)。
McGinnis, C. S.等。MULTI-seq:使用脂质标记指标进行单细胞RNA测序的样本多路复用。Nat方法。16, 619-626(2019)。
斯托克修斯等人。使用条形码抗体的细胞哈希使单细胞基因组学的多路复用和双态检测成为可能。基因组医学杂志。19, 224(2018)。
达林格,P.等人。用单细胞转录组读出的混合CRISPR筛选。Nat方法。14, 297-301(2017)。
Mimitou, e.p.等。单细胞中蛋白质、转录组、克隆型和CRISPR扰动的多重检测。Nat方法。16, 409-412(2019)。
玛格丽特等。增殖细胞和静止细胞中裂变酵母转录组和蛋白质组的定量分析。细胞151, 671-683(2012)。
郑国勇,等。大规模并行数字单细胞转录谱分析。Commun Nat。8, 14049(2017)。
O 'Huallachain等人。通过分裂池合成超高通量单细胞分析蛋白质和rna。Commun。医学杂志。3., 213(2020)。
曹,J.等。多细胞生物的综合单细胞转录谱分析。科学357, 661-667(2017)。
曹,J.等。数千个单细胞中染色质可及性和基因表达的联合分析。科学361, 1380-1385(2018)。
曹,J.等。哺乳动物器官发生的单细胞转录景观。自然566, 496-502(2019)。
罗森博格等人。用分裂池条形码对发育中的小鼠大脑和脊髓进行单细胞分析。科学360, 176-182(2018)。
拉里奥,C. A.等。基于液滴的大规模单细胞染色质可及性的组合索引。生物科技Nat。》。37, 916-924(2019)。
达林格,P.等人。超高通量单细胞RNA测序与组合流控标度扰动筛选。Nat方法。18, 635-642(2021)。
希顿,H.等。Souporcell:通过基因型对单细胞RNA-seq数据进行无参考基因型的稳健聚类。Nat方法。17, 615-620(2020)。
Gehring, J., Hwee Park, J., Chen, S., Thomson, M. & Pachter, L.用DNA寡核苷酸标记细胞蛋白的高多重单细胞RNA-seq。生物科技Nat。》。38, 35-38(2020)。
Ferrer-Font, L.等。利用谱流式细胞术进行高维免疫分型分析的面板设计和优化。咕咕叫。Protoc。Cytom。92, e70(2020)。
柯林,M. & Bigley, V.人类树突细胞亚群:更新。免疫学154, 3-20(2018)。
Zunder, E. R.等。采用双滤波方案和单细胞反褶积算法的钯质标签细胞条形码。Protoc Nat。10, 316-333(2015)。
特拉格,V. A.沃特曼,L. &范埃克,N. J.从鲁汶到莱顿:保证良好连接的社区。科学。代表。9, 5233(2019)。
确认
C.J.Y, Y.S.S.和M.H.S.是陈·扎克伯格生物中心调查员,C.J.Y和M.H.S.是PICI的成员。美国国立卫生研究院(NIH)拨款号R01AR071522, R01AI136972和R01HG011239进一步支持了C.J.Y.。E.D.C.由加州大学旧金山分校先进技术中心PBBR拨款支持。这项工作由美国国立卫生研究院资助no。DP5 OD023056和UCSF PBBR给M.H.S.和NIH的资助。S10 1S10OD018040,使本研究中使用的Helios细胞仪得以采购。我们感谢PFCC (RRID:SCR018206)的部分资助。NIH P30 DK063720和NIH S10仪器批准号。S10 1 s10od021822-01。这项研究也得到了美国国立卫生研究院的资助。 R35-GM134922.
作者信息
作者及隶属关系
贡献
C.J.Y, B.H.和D.S.L.构想了这个实验。B.H.和D.S.L.设计并进行了实验。B.Z.Y.和K.L.N.好心地提供了用于商业相容性实验的抗体。w.t. D.S.L C.J.Y B.H。,。,A.O。G.C.H, A.W。,屈服应力,廖曜生,E.D.C. M.H.S.分析结果。所有作者都审阅了手稿。
相应的作者
道德声明
相互竞争的利益
C.J.Y.是Related Sciences和ImmunAI的SAB成员并持有股权,Maze Therapeutics的顾问并持有股权,以及Trex Bio的顾问。陈振宇获得了陈-扎克伯格倡议、陈-扎克伯格生物中心和基因泰克的研究支持。
额外的信息
同行评审信息自然方法感谢匿名审稿人对本工作的同行评议所作的贡献。Arunima Singh是本文的主要编辑,并与编辑团队的其他成员合作管理其编辑过程和同行评审。
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展数据
图1 SCITO-seq的仿真和成本分析。
一个,碰撞率(y轴,记为CRS)作为加载单元数和池数(用不同颜色表示)的函数。这里的模拟次数如下:当cells_loaded < = 1e4时maxSim = 1000,当1e4 < cells_loaded < = 1e5时maxSim = 100,当1e5 < cells_loaded时maxSim = 10。预期的(实线)和模拟的(虚线)碰撞率基于10万液滴的泊松统计量,模拟时将含有细胞的液滴数量建模为0.6 × 10万。当加载的单元格数量不大(例如小于10,000)时,碰撞数量会有明显的差异,因此使用多次模拟运行来估计虚线中显示的碰撞率。b,液滴数量(y轴)不包含细胞(蓝色),正好一个细胞(绿色)或大于一个细胞(红色)作为加载细胞数量(x轴)的函数。单态是指液滴中包含一个细胞,多态包含多个细胞(>=2),而空态液滴中不包含细胞。c,不同细胞加载数(cells_loaded)基于泊松分布的每液滴细胞数的分布(y轴,计数)(x轴)。d,(左)液滴碰撞率,描述至少发生一次条形码碰撞的液滴比例。(右)条形码碰撞率,估计给定液滴中有碰撞的批次(池)的比例。碰撞率的计算使用泊松点过程(实线)或封闭形式的解(虚线;见的方法)。来自封闭形式解的估计健壮地和几乎相同地概括了模拟,并可用于计算实验的碰撞率。e,总成本估算(紫色)包括文库准备(绿色)、抗体准备(红色)和测序成本(蓝色),假设40个reads/Ab/cell和30个抗体组用于不同数量的SCITO-seq池。
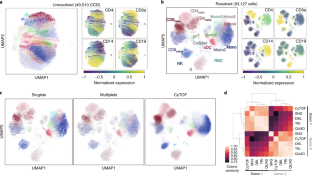
扩展数据图2物种混合QC分析(人/小鼠)。
一个,(上)人类和小鼠细胞的转录组UMAP,通过转录本对齐来区分。(较低)小鼠和人细胞ADT染色叠加在转录组UMAP上进行100k加载实验。合并每个抗体的池条形码(即CD29_h_merged-1 = CD29_h_barcode-1 + ....)+CD29_h_barcode-5,其中后一个数字表示池号)。物种分类在转录组上由> 95%的截止值确定,该截止值基于特定于任何物种的归一化计数。未达到阈值的细胞被归类为多胞胎。重叠归一化ADT计数显示人与小鼠抗体染色。b,当加载100k细胞时,在批次中显示物种多胞胎(双正轴上显示)的散点图。使用单个批处理条形码和Multiplets注释的单元格类型的解析(对于两个池条形码都为正数,例如CD29h_barcode1和CD29h_barcode2为正数或CD29m_barcode1和CD29m_barcode2)。c,物种混合20k(左)和100k(右)加载实验的散点图,由池着色,显示池特定的染色水平。坐标轴上的分解hCD29或mCD29是指分解成单细胞后的归一化抗体计数。如果液滴中含有来自池1和池2的hCD29的混合物,液滴被分解为两个具有池归一化计数的细胞。d, 2 × 10的SCITO-seq归一化计数散点图4物种混合负荷以确定跨池或池内背景水平。e,基于2 × 10的直接偶联hCD29和mCD29抗体归一化计数散点图4使用直接共轭法显示跨池或池内背景水平的物种混合负荷。Hg19、mm10和Multiplet根据各自的转录组比对定义细胞群。直接共轭为SCITO-seq系统中的噪声提供了基线。
图3物种混合QC分析(人B/T细胞)。
一个, 2 × 10 T/B细胞实验的UMAP投影(左)5负载按转录组测定的细胞类型着色(高度可变基因差异的截止值为0.9)。双联(多胞胎)表示T和B的混合,用绿色表示。另外两个面板显示了合并(合并所有5个池条形码)和标准化SCITO-seq计数的特定染色。b, 200k T/B实验的散点图,按池加载彩色。坐标轴上的分解hCD4或hCD20是指分解成单细胞后的归一化抗体计数。例如,如果液滴是池1到池5的hCD20的混合物,则解析计数应该是特定池的归一化计数中的任意一个(对于池1图例,已解析轴由归一化池1计数表示)。c,在2 × 10的估计(观察)和预期(模拟)之间,估计(x轴)与预期(y轴)的多胞胎(包含1个细胞到5个细胞的液滴频率)5加载实验(左)。五个点代表液滴中的细胞数量(从单个细胞到五个细胞)。加载浓度为2 × 10时,抗体池条形码之间共出现的预期(x轴)与观察(y轴)频率(右)5细胞。根据单线阵中条形码的频率计算出预期频率。同时给出了相关值和p值。使用R包mixtools (v1.0.4)计算观察到的抗体池条形码的共现情况,实现normalmixEM函数,默认参数(epsilon = 1e-08, maxit = 1000)。d,在200k负载实验中,从每个供体含有单池条形码(S)和多池条形码(M)的液滴中分解的细胞中,每个抗体的归一化UMI计数的分布。抗体在池多胞胎中的分布显示了预期的混合比例(供体1 (D1)为5:1,供体2 (D2)为1:3),并与池单胞胎中的相应分布重叠。
扩展数据图4人体供体PBMC QC分析(28plex)。
一个,池特异性抗体28-plex实验脊图。将每个池内60个抗体归一化表达值相加进行阈值。一个单独的图包含批量特定的归一化表达式值,用于演示预期特异性的信号到噪声分布。b,代表标记为100k PBMC加载实验的UMAP投影。使用RNA表达的UMAP投影比较(左),拆分前(中)和拆分后(右)合并抗体计数。标尺表示标准化合并ADT计数(左和中)和解析ADT计数(右)。
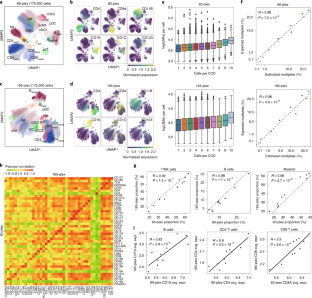
图5人体供体PBMC QC分析
一个,基于UMAP投影的RNA表达为200k PBMC负载的代表性标记。由于RNA分子没有组合索引,这些UMAP与基于归一化ADT计数的解析UMAP形成鲜明对比,我们可以看到所有簇的明显区别。bUMAP ADT投影的200k加载PBMC数据由不同的池着色(颜色编号0到9)。两个池供体在10个不同的池中进行混合,以调查所有染色井的批量效应,发现没有明显的批量效应。c, ADT UMAP簇覆盖在ADT UMAP(左)上,ADT UMAP簇覆盖在转录组UMAP上。d,(上)CD4/8和CD45RA/RO在ADT UMAP上的蛋白表达。(下)CD4/8和CD45RA/RO转录组UMAP蛋白表达。
图6人体供体PBMC QC分析
一个UMAP (x轴,y轴,UMAP1和UMAP2维度),具有代表性的PBMC标记,基于CyTOF实验,使用与SCITO-seq相同的供体和抗体面板。刻度显示arcsinh(双曲反正弦)转换的归一化值。b,对于100k加载数据(SNG:单线态,DBL:双线态,TRI:三线态),SCITO-seq与每个供体的CyTOF (D1:上,D2:中)的比较。还显示了成对相关热图(底部)(类似于图。二维在主图中)。在捐赠者内部,各莱顿聚类的比例高度相关(捐赠者内部的余弦相似度为1:0.95,捐赠者内部的余弦相似度为2:0.94)。
扩展数据图7可伸缩性实验和QC分析(60和165 plex)。
一个、采用Totalseq-C兼容系统的夹板低聚体设计。将夹板寡核苷酸(FBC RC(反向补体)+ Well + Ab BC (5 + 5,10 bp) + Read2 + Totalseq-C Barcode RC)杂交到Totalseq-C寡核苷酸偶联抗体的条形码区域(蓝色区域周围的右虚线)。将寡聚夹板(1 μM)与抗体孵育15 min进行杂交(流程与常规SCITO-seq相同)。井和抗体条形码序列用橙色和蓝色编码。b, 60-plex实验脊图显示池特异性抗体的特异性。每个抗体池60个抗体计数和的归一化表达值。单个图包含批量特定的归一化表达式值,以显示预期特异性的信噪比分布。c, 165-plex实验脊图显示池特异性抗体的特异性。每个抗体池中所有165个抗体的归一化表达值。单个图包含批量特定的归一化表达式值,以显示预期特异性的信号对噪声分布。d, 165-plex实验的Barplots显示,在所有10个池(前1-5批,后6-10批)中,一个示例同型对照ADT计数(大鼠IgG)的UMI计数较低。计算表达ADT小于2umi或大于2umi的细胞百分比(y轴)。批次间的背景噪声显示~92%的细胞中小于2umi计数。e,样本CD8a与同型对照Ab(大鼠IgG)的叠加密度直方图,以评估165 plex数据中所有10个池(顶部1-5批,底部6-10批)的“噪声”水平。x轴为log1p(原始计数)转换值,y轴为密度。f,样本抗体聚集在所有10个池(CD3, CD11c, CD45, CD127, CD8a, CD4, CD19)与同型对照Ab(大鼠IgG)的叠加密度直方图,以评估165-plex数据中的“噪声”水平。x轴为log1p(原始计数)转换值,y轴为密度。
扩展数据图8可伸缩性实验和QC分析2(60和165 plex)
一个, 60 plex(上)和165 plex(下)实验的umap显示cDC1、cDC2和pDC标记的正常表达。CD141和CD370用于cDC1, CD1c用于cDC2, CD123和CD303用于pDC标记。b,样本多路复用SCITO-seq的示意图,其中不同的样本用不同的池条形码(红,蓝,紫)散列。含有不同个体(两种不同颜色)细胞的液滴可以分解成单独的细胞。c, 60-plex实验中CD4抗体所有组合归一化表达值的成对相关图(使用GGally R包中的ggpair函数)。d, TSC 165-plex实验中CD4抗体各组合归一化表达值的成对相关图。
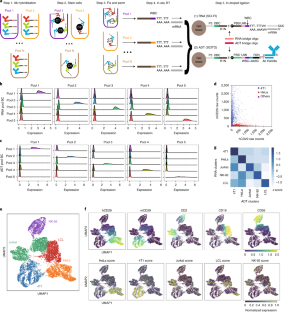
扩展数据图9商品性实验和QC分析的发展。
一个,使用ATAC-kit分析SCITO-seq的概念验证实验。用12种代表性表面标记物(CD4, 8a, 14, 16, 45, 45RA, 45RO, 19, 20, 56, 11c和HLA-DR)在5个不同的5x10负载池中染色pbmc的UMAP4细胞(nCM:非传统单核细胞,cMono:常规单核细胞)。B,利用10x Genomics ATAC-seq试剂盒,在交配实验中GEM连接步骤示意图。使用scifi-RNA-seq工作流程在GEM反应过程中捕获RNA和ADT的详细序列结构。更详细的RNA工作流程可以在scifi-RNA-seq论文的补充图2中找到。10x_round2是指16 bp的液滴条形码,round1条形码是指井眼条形码(11 bp)中使用的原位逆转录反应。在反转录反应(MMLV变体)的末尾添加未模板化的‘CCC’。抗体条形码(Ab BC 10-bp)和抗体柄(Ab柄20-bp,直接偶联到蓝色抗体)序列是特定于每个抗体的。Read2n表示Read2 Nextera序列。与桥式寡核苷酸1(用于捕获原位RT mRNA分子)相比,桥式寡核苷酸2有额外的10个bp(红色和蓝色序列之间的AACGTATCGA)。ddC(双脱氧C)和InvdT(倒置dT)用于防止延伸。箭头表示GEM反应中的连接位点。c,采用规范化RNA计数的UMAP进行降维,并在UMAP空间上进行相应细胞系特异性ADT标记表达。d,采用规范化RNA计数的UMAP降维,并在UMAP空间上进行相应的单RNA标记表达。
图10 SCITO-seq流动验证实验
一个,为了减少次级寡核苷酸的非特异性染色,我们以1 μM(右)和100 μM(左)滴定寡核苷酸。将寡核苷酸偶联抗体与Cy5偶联的反向互补寡核苷酸杂交15分钟后,将LCLs和原代单核细胞的混合物用杂交材料和CD13-BV421染色30分钟,洗涤两次,并在LSRII上分析。通过Violet-F通道(x轴)捕获CD13 BV421抗体,在Red-C通道捕获Cy5标记的次级寡核苷酸,以检查背景染色水平(Q6门控群体是指非同源次级寡核苷酸在原代单核细胞群体中的溢出)。b为了确定1 μl 1 μM反向互补寡核苷酸是否能饱和1 μg抗体,我们首先将1 μl结合CD3的寡核苷酸与1 μl结合Cy5的反向互补寡核苷酸杂交。在此之后,再加入1 μl 1 μM反向互补FAM共轭寡聚物15分钟,然后清洗和分析。红色群体为用序贯杂交抗体染色的细胞。蓝色群体代表用偶联CD3抗体染色,但没有Cy5或FAM寡核苷酸杂交序列的细胞。c,在CD8a表达的样本进行分类之前,对淋巴细胞进行单态和活细胞的门控(在YG C-A通道中捕获PI信号)。红色- c代表CD8a-APC,蓝色- b代表同型对照- af488。
权利和权限
关于本文
引用本文
黄斌,李德寿,田木伟。et al。SCITO-seq:单细胞组合索引细胞术测序。Nat方法18, 903-911(2021)。https://doi.org/10.1038/s41592-021-01222-3
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41592-021-01222-3
这篇文章被引用
在诊断应用中使用dna条形码抗体拆分池测序定量单细胞上的蛋白质丰度
科学报告(2022)
通过组合索引优化单核转录谱
自然的协议(2022)
单细胞原位蛋白质分析的先进技术
【科学中国】化学(2022)
