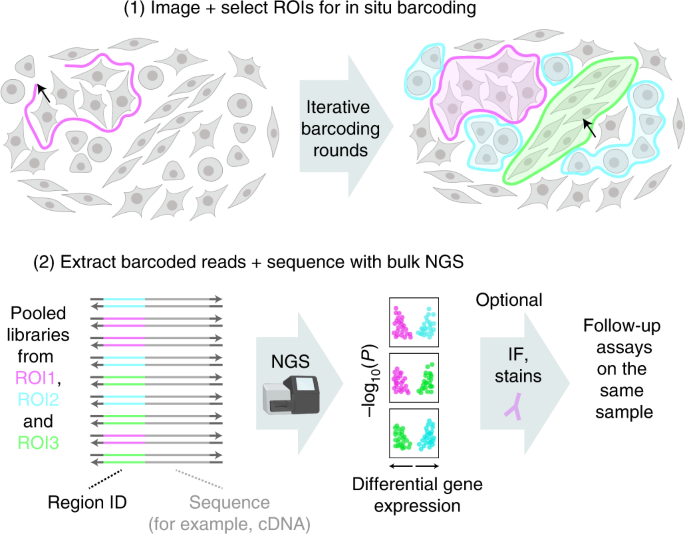
摘要gydF4y2Ba
我们提出了Light-Seq,一种在固定细胞和组织中使用光定向DNA条形码对完整生物样本进行多重空间索引的方法,然后进行迁位测序。Light-Seq结合了空间定向,快速光交联DNA条形码到互补DNA原位与一步DNA拼接反应,创建池,空间索引测序文库。这种光定向条形码能够在完整的固定组织样本中对多个细胞群进行原位选择,用于基于位置、形态或蛋白质染色的全转录组测序,而无需细胞分离。将Light-Seq应用于小鼠视网膜切片,我们从三个细胞层中恢复了数千个差异富集的转录本,并发现了一种非常罕见的神经元亚型,多巴胺能无分泌细胞的生物标志物,每个切片只有4到8个单个细胞。Light-Seq提供了一个可访问的工作流程,将原位成像和蛋白质染色与相同细胞的下一代测序相结合,使样品完整,以便测序后进行进一步分析。gydF4y2Ba
主要gydF4y2Ba
为了全面了解驱动生物功能和病理的细胞状态,通过下一代测序(NGS)将光学表征和基于成像的固定细胞和组织筛选与组学水平分析相结合将是理想的。gydF4y2Ba
最近的应用集中在整合成像与转录组测量相同的单个细胞主要依赖于物理解离和细胞分类使用光流体或微流体成像之前,之后或期间gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.另一种方法依赖于荧光蛋白在选定的活细胞中的光转换,以便稍后进行分选gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba或光激活信使RNA捕获部分,然后物理吸取目标活细胞进行转录组分析gydF4y2Ba8gydF4y2Ba.这些方法通常需要细胞的实时成像和提取有限数量的视觉特征,然后再对分离细胞进行池化或单细胞分选以进行测序。在这些情况下,空间信息或单元格的原始位置通常丢失或不相关。gydF4y2Ba
为了保存空间信息和形态,特别是在组织中,最近的空间转录组学方法旨在原位分析细胞gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba.利用各种模式的DNA条形码,空间组学方法旨在空间索引二维捕获表面(Slide-SeqgydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba, HDSTgydF4y2Ba13gydF4y2Ba, Seq-ScopegydF4y2Ba14gydF4y2Ba, Stereo-SeqgydF4y2Ba15gydF4y2Ba, Sci-SpacegydF4y2Ba16gydF4y2Ba)或生物分子。在后一种情况下,条形码序列可以通过组合条形码探针的原位杂交(ISH),或通过逆转录(RT)或结扎(例如,在微流控将条形码递送到DBIT-Seq中定义的样品位置后)被合并到生物分子上gydF4y2Ba17gydF4y2Ba).然后可以通过迭代成像(例如SeqFISH)在原位读取这些条形码gydF4y2Ba18gydF4y2Ba或MERFISHgydF4y2Ba19gydF4y2Ba, HybISSgydF4y2Ba20.gydF4y2Ba),通过原位测序(FISSEQ .gydF4y2Ba21gydF4y2Ba, CARTANAgydF4y2Ba22gydF4y2Ba)或从样本中提取后进行非原位测序(DBIT-SeqgydF4y2Ba17gydF4y2Ba, IGSgydF4y2Ba23gydF4y2Ba).替代方法使用空间限制(1)迭代光切割和从目标感兴趣区域(ROIs) (DSP)收集ISH探针条形码gydF4y2Ba24gydF4y2Ba)或(2)通过选择性固定化收集靶细胞本身gydF4y2Ba25gydF4y2Ba,吸gydF4y2Ba26gydF4y2Ba或者激光捕获显微解剖gydF4y2Ba27gydF4y2Ba用于随后的条形码和测序。gydF4y2Ba
现有的空间分析方法目前依赖于一种或几种昂贵的仪器设备(耗资10万至100万美元的封闭盒子系统,高端定制显微镜或荧光分选机),复杂的条形码阵列或条形码序列的多轮光学反卷积,以及具有严格样品格式限制的定制微流体系统或阵列。许多这些方法也使用靶向ISH探针,而不是整个转录组测序或部分或完全破坏样本。这就产生了对可访问和可扩展的可视化选择方法的高度需求,这些方法可以直接将多维和高分辨率的细胞表型(包括形态、蛋白质标记、空间组织)与不同样本类型的转录组谱联系起来。在这一方向上,最近的方法采用了紫外线(UV)-笼化方法来实现rna (PIC)的空间条形码gydF4y2Ba28gydF4y2Ba)或整个细胞(通过dna条形码抗体或脂质,如ZipSeqgydF4y2Ba29gydF4y2Ba),但连接到测序输出需要销毁样本。gydF4y2Ba
在这里,我们提出了一种不同的范式,称为Light-Seq,用于在不破坏样品的情况下,在所需的roi中对离地NGS的目标分子进行光定向原位空间条形码(图2)。gydF4y2Ba1gydF4y2Ba).我们通过两项创新来实现这一目标:(1)建立在我们之前光控制核苷酸快速交联的工作基础上gydF4y2Ba30.gydF4y2Ba,我们利用超快交联化学gydF4y2Ba31gydF4y2Ba和平行光刻gydF4y2Ba32gydF4y2Ba用于将预先设计的条形码序列光控无酶共价附着在原位生物分子上。(2)开发交叉结合成反应,将DNA条形码整合到生物分子上进行迁位测序。我们将这种条形码策略与非破坏性工作流程相结合,使固定样本中所选细胞的成像和整个转录组测序成为可能,并有可能重新访问样本进行进一步的分析,如蛋白质染色。我们测试并证明了Light-Seq在混合细胞培养和小鼠视网膜组织切片上的适用性,并利用罕见细胞转录组学方法,在其中我们识别了小鼠视网膜中非常罕见的多巴胺能无分泌细胞(dac)的生物标志物。gydF4y2Ba
结果gydF4y2Ba
Light-Seq概述和条形码化学gydF4y2Ba
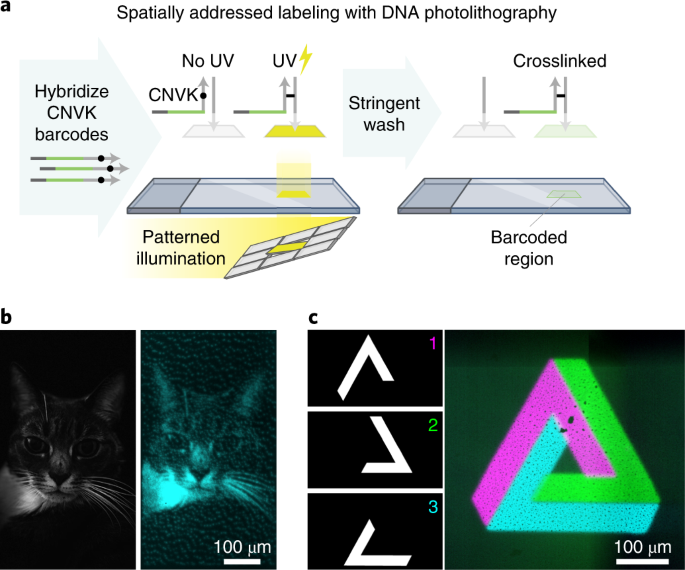
Light-Seq采用光控DNA条形码附着策略,以实现成像样本中roi的自定义索引。为了实现这一能力,我们使用了包含超快光交联剂3-氰基咔唑核苷(CNVK)的条形码链。gydF4y2Ba31gydF4y2Ba.杂交的CNVK在短的紫外光照下可以形成链间交联(图。gydF4y2Ba2gydF4y2Ba).我们的一般策略是将含有cnvk的条形码与互补的对接序列杂交,然后将紫外线照射到一个ROI,仅在该区域将条形码链进行光交联,然后将非交联的条形码链冲洗掉。这个过程可以迭代地执行,用正交的条形码链标记多个roi。gydF4y2Ba
一个gydF4y2Ba,玻璃载玻片上的光定向条形码附件示意图。生物素化单链DNA寡聚物通过生物素-链霉亲和素结合固定在玻璃表面。在互补域包含CNVK部分的荧光条形码链与这些固定的低聚物杂交。在所述视场中与roi对应的目标像素以平行方式使用DMD以光交联掩模模式中的条形码进行紫外线照射。未交联的链被严格的清洗去除,这揭示了编码的条形码模式在荧光。gydF4y2BabgydF4y2Ba自定义模式(右)通过使用猫的照片(左)来创建二进制掩模,并将含有cnvk的荧光条形码链光交联到功能化的玻片上。gydF4y2BacgydF4y2Ba,迭代光交联使用三个掩模(左),定义三个roi,将三个正交的条形码链连接到dna涂层的玻片上,形成彭罗斯三角形(右)。gydF4y2Ba
为了建立light - seq平台,我们优化了交联曝光时间和光强,发现1-10 s产生了高效的交联,与之前的体外实验结果相似gydF4y2Ba33gydF4y2Ba.为了以并行方式创建自定义掩模,我们使用了数字微镜设备(DMD)gydF4y2Ba34gydF4y2Ba附在标准的广角成像装置上。使用×10物镜,我们的DMD设置中的一个镜子可以产生一个<2微米的实际分辨率,这是基于在点数组上估计全宽度的半最大值(扩展数据图。gydF4y2Ba1 a, bgydF4y2Ba).我们首先在体外用固定DNA链涂覆的玻璃表面上验证了条形码化学。通过添加荧光标记的条形码链,并使用定制的掩模(例如,猫的掩模)将它们交联到表面,我们能够使用单个条形码链创建模式(图2)。gydF4y2Ba2 bgydF4y2Ba)或使用连续的条形码轮与独特的股图案在同一幻灯片上的多个区域,如三色彭罗斯三角形(图。gydF4y2Ba2摄氏度gydF4y2Ba).虽然我们主要使用带有DMD的365纳米发光二极管(LED)外显照明进行UV交联步骤,但我们也使用405纳米激光演示了亚细胞空间标记gydF4y2Ba30.gydF4y2Ba在共聚焦点扫描显微镜上(扩展数据图。gydF4y2Ba1 c ggydF4y2Ba).激光扫描系统提供更高的分辨率和对比度,但比DMD和LED照明慢。gydF4y2Ba
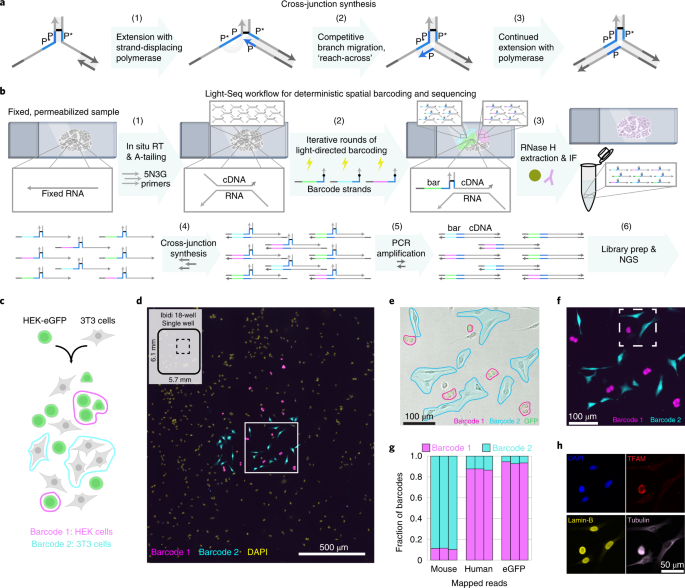
接下来,要通过NGS读取目标dna的序列及其相应的交联条形码序列,必须在不丢失条形码标识或条形码序列的情况下对交联碱基进行寻址。为此,我们开发了一种交叉结合成反应,将条形码DNA序列和条形码复制到一个新的DNA单链中,而不需要交联(图2)。gydF4y2Ba3gydF4y2Ba).为此,我们使用类似于我们之前开发的引物交换反应(PER)的策略gydF4y2Ba35gydF4y2Ba.我们使用带有一条链置换聚合酶的引物,该聚合酶复制一条新链,直到它在交联点停止。我们设计了交联周围的序列,使其具有相同的结构域,这样扩展的引物就可以通过分支迁移到达结,并在相反的链上进行模板化gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba两个相同领域之间的竞争。这种交叉结反应的单链DNA产物可以用标准的NGS管道进行扩增和读出。gydF4y2Ba
一个gydF4y2Ba交叉结合成反应的设计。首先,引物将新链延伸至挡板(步骤1)。接下来,延伸的引物P域通过分支迁移与对面模板上相同的P域竞争,类似于我们之前开发的引物交换反应(PER)。gydF4y2Ba35gydF4y2Ba(步骤2)。一旦被取代,合成的P结构域(蓝色)引物可以结合形成一个三向结,然后继续延伸(步骤3)。P结构域通常是7 nt,如果Bst聚合酶a -tails,它可能变成8 nt。gydF4y2BabgydF4y2BaLight-Seq原位转录组测序工作流程:(1)RT使用随机引物进行,引物包含一个5 '条形码停靠位点,然后是3 ' cDNA末端的a尾。(2)在每个ROI中,一个唯一的cnvk修饰的DNA条形码链被uv交联到5 ' cDNA dock位点上。(3)使用RNase H提取条形码cdna,该酶在RNA - dna杂交体中切割RNA。(4)交叉结合成反应将条形码和cDNA序列复制成一条单链,用于(5)PCR扩增和(6)测序文库制备和NGS。gydF4y2BacgydF4y2Ba,细胞混合试验:将表达egfp的HEK293与小鼠3T3细胞共培养固定,分别用条形码1和2标记cdna。gydF4y2BadgydF4y2Ba,将~25个3T3细胞(青色)和~25个HEK细胞(洋红色)的子集条形码编码在一个18孔室盖的整个孔中(原理图上虚线的矩形区域标记了该面板中显示的缝合图像相对于整个孔的面积的大小),每个包含约4500个细胞(gydF4y2BangydF4y2Ba= 3个技术重复,代表性图像所示)。所有细胞条形码后用DAPI染色(黄色)。gydF4y2BaegydF4y2Ba, Brightfield和GFP荧光覆盖了用于条形码1和2标记的roi(视场为面板的放大倍数)gydF4y2BadgydF4y2Ba,白色方块)。gydF4y2BafgydF4y2Ba,面板的荧光图像gydF4y2BaegydF4y2Ba经过光交联条形码1(洋红色)和2(青色)。gydF4y2BaggydF4y2Ba,在合并的人和小鼠参考基因组中,部分reads映射到人、小鼠或eGFP序列,分别用Barcode 1或2标记(gydF4y2BangydF4y2Ba= 3个技术重复)。gydF4y2BahgydF4y2Ba,条形码序列提取后,相同的细胞(白色方块从面板gydF4y2BafgydF4y2Ba)用IF染色检测Lamin-B(黄色)、tubulin(紫色)和TFAM(红色,人表位特异性)。gydF4y2Ba
利用Light-Seq进行全转录组测序gydF4y2Ba
为了使用Light-Seq进行整个转录组测序,我们首先进行原位RTgydF4y2Ba39gydF4y2Ba在固定的和渗透的细胞或组织切片上合成互补DNA (cDNA)(图。gydF4y2Ba3 bgydF4y2Ba为了标记rna而不考虑多聚腺苷酸,我们使用了一个简并引物,在3 '端有5个N和3个G碱基gydF4y2Ba40gydF4y2Ba在5 '端有一个条形码对接点。RT后,生成的cdna的3 '端a尾,形成一个3 '柄,用于原位引物结合。然后将含有CNVK和唯一分子标识符(UMI)的条形码链杂交到所有cdna上的5 '对接位点。然后,我们将紫外线直接照射到ROI,使该区域的条形码链光交联,然后洗掉非交联的条形码链。这一过程可以迭代执行,用正交条形码链标记多个roi(图2)。gydF4y2Ba3 bgydF4y2Ba,步骤2)。gydF4y2Ba
在所有的roi被条形码后,用温和的RNase H处理从样本中收集条形码cdna,并准备测序(图2)。gydF4y2Ba3 bgydF4y2Ba,步骤3)。然后,我们应用我们的交叉结合成反应,将cdna和条形码序列通过一个酶催化反应步骤缝合到一个单独的读出链上(图3)。gydF4y2Ba3 bgydF4y2Ba这种将空间条形码直接附着到转录组序列上的方法可以在PCR扩增和NGS文库制备后直接过渡到读出序列的迁位测序(图4)。gydF4y2Ba3 bgydF4y2Ba,步骤5-6)。在制定方案时,我们做出了几个关键的设计选择,以最大限度地减少先前观察到的原位RT反应的潜在伪影gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,我们将在补充附注中详细讨论gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
为了验证基于表型谱选择和条形码细胞的能力,我们进行了一个细胞混合实验,将小鼠3T3细胞与稳定表达eGFP的人HEK细胞混合(图2)。gydF4y2Ba3 cgydF4y2Ba).我们通过基于eGFP表达和细胞形态手动选择具有不同条形码序列的每种细胞类型(为了可视化目的,与不同的荧光团耦合)(补充图)。gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba2gydF4y2Ba).在同一孔中总共约4500个细胞中,我们对~25个人类细胞和~25个小鼠细胞进行了条形码编码,并通过荧光扫描确认了目标细胞类型的条形码是正确的(图2)。gydF4y2Ba3 d-fgydF4y2Ba).测序读数被映射到合并的人类和小鼠基因组,并进一步分析独特的地图。我们能够通过交叉结合成来验证条形码集成,通过匹配读取到我们预期的序列输出(补充图。gydF4y2Ba3得了gydF4y2Ba).我们观察到小鼠和人类地图与各自条形码序列的良好辨别率(小鼠读数为89.1±0.7%,人类读数为87.3±0.7%,gydF4y2BangydF4y2Ba= 3个技术重复,平均值±s.d),值得注意的是,93±0.5%的eGFP读取正确归因于人类特异性条形码(图。gydF4y2Ba3 ggydF4y2Ba).序列提取后,我们进行了多重免疫荧光(IF),以验证样品的完整性,用于二次检测(图2)。gydF4y2Ba3 hgydF4y2Ba).gydF4y2Ba
接下来,我们估计了Light-Seq可以捕获的转录本的丰度。因为Light-Seq的条形码区域可以由用户任意设置,所以我们选择将Light-Seq可以捕获的转录本数量归一化,即每个“单位区域”的UMI序列数量,大约是Slide-Seq中珠子的大小gydF4y2Ba11gydF4y2Ba或者DBIT-Seq编码的方块gydF4y2Ba17gydF4y2Ba,我们定义为10 × 10µmgydF4y2Ba2gydF4y2Ba.我们通过分割ROIs的显微图像,估计每个条形码受紫外线照射的总细胞或组织面积,从而获得单位面积的UMIs数量(扩展数据图)。gydF4y2Ba2gydF4y2Ba),并计算100µm的平均UMI计数(正确条形码和唯一映射读数)gydF4y2Ba2gydF4y2Ba条形码区域。在每个重复约3000万reads的测序深度下,只有约1/2的样本被扩增用于文库制备和测序,我们观察到HEK和3T3细胞平均每单位面积分别为1959±453和1170±207 UMIs(补充表)gydF4y2Ba1gydF4y2Ba,平均值±s.d)。我们注意到~ 3000万的读取深度是不饱和的(扩展数据图。gydF4y2Ba2 bgydF4y2Ba),来自一半样本的单个约2亿次读取数据集分别为HEK和3T3细胞产生3328和2029个单位面积的UMIs(补充表gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
归一化基因表达水平(以对数表示)gydF4y2Ba2gydF4y2Ba-transformed transcripts per kilobase per million reads (TPM)在技术重复之间表现出良好的相关性(Pearson相关系数> 0.8)。表达量最高的200个基因在技术重复间高度相关(Pearson相关系数> 0.9;扩展数据图。gydF4y2Ba2 c, dgydF4y2Ba),包括各种蛋白质编码基因,以及短转移rna和长非编码rna(补充表gydF4y2Ba2gydF4y2Ba以及源数据),说明了条形码策略可以针对的转录本的范围。gydF4y2Ba
使用Light-Seq,我们能够成功地恢复目标细胞的物种特异性转录组,尽管只选择了整个孔中1-2%的细胞。我们假设,在Light-Seq背景中,由一小部分物种特异性转录本所反映的条形码对应于其他物种(图。gydF4y2Ba3 ggydF4y2Ba),可能来自三个潜在的来源:(1)光交联前rna或cdna的扩散,(2)严格清洗期间未交联条形码链的不完全去除,以及(3)光散射诱导的roi外条形码。为了缓解(1)和(2),我们在接下来的所有实验中都在条形码杂交步骤中添加了阻塞剂和拥挤剂,这大大改善了信噪比(gydF4y2Ba方法gydF4y2Ba及补充说明gydF4y2Ba2gydF4y2Ba).为了减轻光散射的影响,我们在汇合点下方播种了细胞。对于高密度标记(例如,在组织中),我们建议优化光学设置和样品制备,并轻微侵蚀ROI边界(~ 1-3 μ m)以考虑散射(gydF4y2Ba方法gydF4y2Ba和扩展数据图。gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
组织切片的Light-Seq空间测序gydF4y2Ba
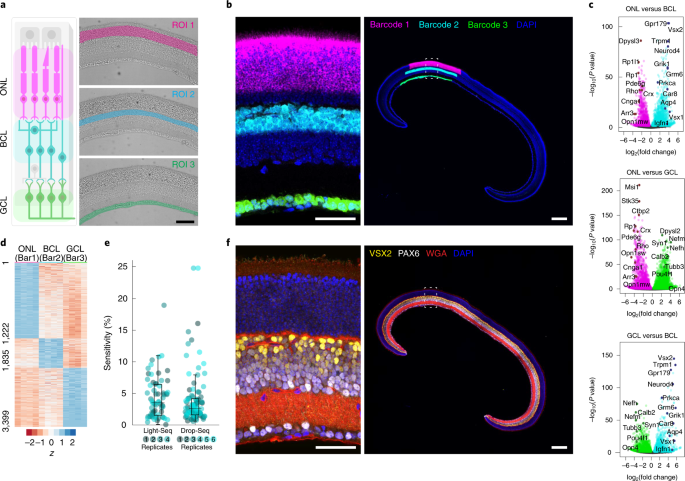
组织样本中特定细胞群的RNA测序仍然具有挑战性,特别是当靶细胞很少或难以分离时。因此,我们在小鼠视网膜的固定切片上测试了Light-Seq,以基于原位鉴定从感兴趣的细胞群中捕获生物标志物。首先,在固定的18µm视网膜冷冻切片中进行原位RT,合成用于空间条形码的cdna。然后,我们手动选择了视网膜的三个细胞层,每个细胞层都有独特的功能和细胞组成:外核层(ONL),包含杆状和锥状光感受器;双极细胞层(BCL),包括双极细胞、水平细胞和Müller胶质细胞;和神经节细胞层(GCL),包括视网膜神经节细胞和移位的无分泌细胞(ACs)(图。gydF4y2Ba4gydF4y2Ba).在将荧光标记的条形码光交联到每一层内的所有cdna后,使用共聚焦显微镜对目标roi成像,以验证层特异性标记(图2)。gydF4y2Ba4 bgydF4y2Ba和扩展数据图。gydF4y2Ba3得了gydF4y2Ba).然后按照上述方法提取条形码cdna,将其合并并制备NGS,保持样品完整。gydF4y2Ba
一个gydF4y2Ba小鼠视网膜的三个区域被唯一编码:ONL具有条形码1,BCL具有条形码2和GCL具有条形码3。gydF4y2BabgydF4y2Ba,条形码后,在目标细胞层中检测到荧光标记的条形码链:ONL(洋红色,条形码1;1112±199个细胞,gydF4y2BangydF4y2Ba= 4节),BCL(青色,条形码2;298±29个细胞,gydF4y2BangydF4y2Ba= 4节)和GCL(绿色,条形码3;91±14个细胞,gydF4y2BangydF4y2Ba= 4节)。gydF4y2BacgydF4y2Ba, ONL与BCL(上)、ONL与GCL(中)、BCL与GCL(下)之间差异表达基因的火山图,并标记了选定标记。的gydF4y2BaxgydF4y2Ba而且gydF4y2BaygydF4y2Ba坐标轴显示日志gydF4y2Ba2gydF4y2Ba(折叠变化)和对数gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba分别值)。gydF4y2BadgydF4y2Ba,热点图gydF4y2BazgydF4y2Ba-仅在一层富集的差异表达基因的得分(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05;见源数据;多重假设检验的双侧Wald检验和Benjamini-Hochberg调整)。gydF4y2BaegydF4y2Ba, Light-Seq估计灵敏度箱线图(gydF4y2BangydF4y2Ba= 4个重复,16个基因)和Drop-SeqgydF4y2Ba46gydF4y2Ba(gydF4y2BangydF4y2Ba= 6个重复,16个基因)与smFISH数据进行比较gydF4y2Ba50gydF4y2Ba.灵敏度定义为(smFISH期望的转录本数)/(Light-Seq或Drop-Seq观察到的读取数)。中线标记中位数,边缘表示第25和第75百分位。胡须扩展到包含所有不被认为是异常值的数据(MATLAB中的默认阈值)gydF4y2Ba箱线图gydF4y2Ba函数;最大须长1.5 ×四分位差)。网点颜色对应复制数。gydF4y2BafgydF4y2Ba提取条形码cdna后,在同一组织切片上对VSX2和PAX6蛋白进行DAPI、WGA染色和IF染色。比例尺长度为100 μmgydF4y2Ba一个gydF4y2Ba, 50µm(左)和200µm(右)gydF4y2BabgydF4y2Ba而且gydF4y2BafgydF4y2Ba.gydF4y2Ba
由于视网膜每一层的细胞组成已经确定gydF4y2Ba44gydF4y2Ba,基于与每个条形码相关的细胞类型特异性标记的水平,我们验证了空间条形码的特异性。我们使用DESeq2(参考。gydF4y2Ba45gydF4y2Ba)对外显子映射的条形码测序reads进行差异表达分析,以寻找层特异性标记。根据条形码区域的大小,我们标记了估计的1112±199,298±29和91±14(平均值±s.d.,gydF4y2BangydF4y2Ba分别为ONL、BCL和GCL层= 4个技术重复)细胞。不同视网膜层的UMI产量不同,取决于每层的细胞和RNA含量,范围从~ 1200到5000 UMI / 10 × 10µmgydF4y2Ba2gydF4y2Ba单位面积(扩展数据图。gydF4y2Ba3 dgydF4y2Ba及补充表gydF4y2Ba3 gydF4y2Ba).正如预期的那样,我们观察到具有较大细胞质体积的细胞与单位面积较高的UMIs相关(扩展数据图)。gydF4y2Ba3 dgydF4y2Ba).gydF4y2Ba
技术复制显示了在整个序列处理管道中一致的读过滤(扩展数据图。gydF4y2Ba4 a、bgydF4y2Ba),并基于各层的主成分分析进行了良好的相关性(扩展数据图。gydF4y2Ba4摄氏度gydF4y2Ba),我们发现了>3,400个基因在对条形码群体之间具有显著的表达差异(ONL和BCL的3,430个基因;BCL与GCL分别为3,434宗;ONL vs GCL 6165,gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05;无花果。gydF4y2Ba4 c, dgydF4y2Ba以及伴随的源数据),包括ONL中杆状和锥状光感受器的许多已知标记,BCL中的双极细胞和Müller胶质细胞以及GCL中的视网膜神经节细胞。gydF4y2Ba
为了进一步对Light-Seq数据进行基准测试,我们使用已发表的ONL和BCL细胞的单细胞Drop-Seq数据模拟视网膜层的伪体RNA测序gydF4y2Ba46gydF4y2Ba(gydF4y2Ba方法gydF4y2Ba).我们发现ONL和BCL之间富集的基因有很强的相关性(在两种检测中均显著富集的基因中有98.6%富集在同一层;扩展数据图。gydF4y2Ba4 dgydF4y2Ba).对于所有bcl富集基因,Light-Seq和Drop-Seq reads的比值对较长的基因显示出较高的相关性和相当的敏感性,而对较短的基因显示出较低的相关性和敏感性(扩展数据图)。gydF4y2Ba4比gydF4y2Ba),与Light-Seq的内部启动策略和Drop-Seq的polya靶向转录本捕获相一致。gydF4y2Ba
有趣的是,尽管在技术重复中检测到的基因总数相似(24,460对24,904),但Light-Seq数据层间的显著差异基因比Drop-Seq数据层(3,430对1,524)要多,这与先前的研究一致,这些研究表明,与单细胞测序相比,靶向的大量转录组测量可以为发现中至低表达生物标志物提供更好的统计数据gydF4y2Ba47gydF4y2Ba.gydF4y2Ba
为了评估Light-Seq检测mrna的敏感性,我们将Light-Seq和Drop-Seq直接与单分子FISH (smFISHgydF4y2Ba48gydF4y2Ba)数据。我们之前的工作使用了定量多路复用的SABER-FISHgydF4y2Ba49gydF4y2Ba共同检测年龄匹配的小鼠视网膜中双极神经元亚型的16种mRNA标记物gydF4y2Ba50gydF4y2Ba.从这些数据中,我们估计了池双极细胞群中每个细胞的平均转录本数量。将其乘以条形码Light-Seq BCL (ROI2)和伪bulk Drop-Seq数据中的细胞数量,我们估计了测序群体中每个基因的可检测转录本的预期数量。相对于smFISH,我们发现Light-Seq的灵敏度为4.29±3.39% (mean±s.d.,gydF4y2BangydF4y2Ba= 16个基因,4个重复),Drop-Seq的敏感性为3.97±4.38% (mean±s.d,gydF4y2BangydF4y2Ba= 16个基因,6个重复)(图gydF4y2Ba4 egydF4y2Ba和扩展数据图。gydF4y2Ba4 h-jgydF4y2Ba).gydF4y2Ba
在Light-Seq之后,用DAPI、小麦胚芽凝集素(WGA)和靶向PAX6和VSX2蛋白的抗体对相同的切片进行染色,表明在提取条形码cdna进行测序后,细胞DNA、细胞膜上的寡糖和蛋白质仍然可以检测到(图2)。gydF4y2Ba4 fgydF4y2Ba和扩展数据图。gydF4y2Ba3 cgydF4y2Ba).gydF4y2Ba
由于RT步骤中的内部启动,Light-Seq显示了跨基因体的读覆盖(扩展数据图)。gydF4y2Ba5gydF4y2Ba)和在不同长度的转录本上一致的每千碱基每百万映射读(RPKM)(扩展数据图。gydF4y2Ba5 b, cgydF4y2Ba).这种策略,结合原位膜渗透,使Light-Seq能够捕获各种各样的RNA物种,包括非聚腺苷酸和线粒体转录本(见图来源数据)。gydF4y2Ba4gydF4y2Ba).当包含内含子序列用于转录组映射时,约21-26%的reads映射到内含子,这表明核RNA的捕获比例与使用内部RNA启动的类似方法一致(补充说明)gydF4y2Ba3 gydF4y2Ba)gydF4y2Ba51gydF4y2Ba这表明我们的工作流程可以同时检测细胞核和细胞质rna。gydF4y2Ba
罕见的细胞转录组与Light-SeqgydF4y2Ba
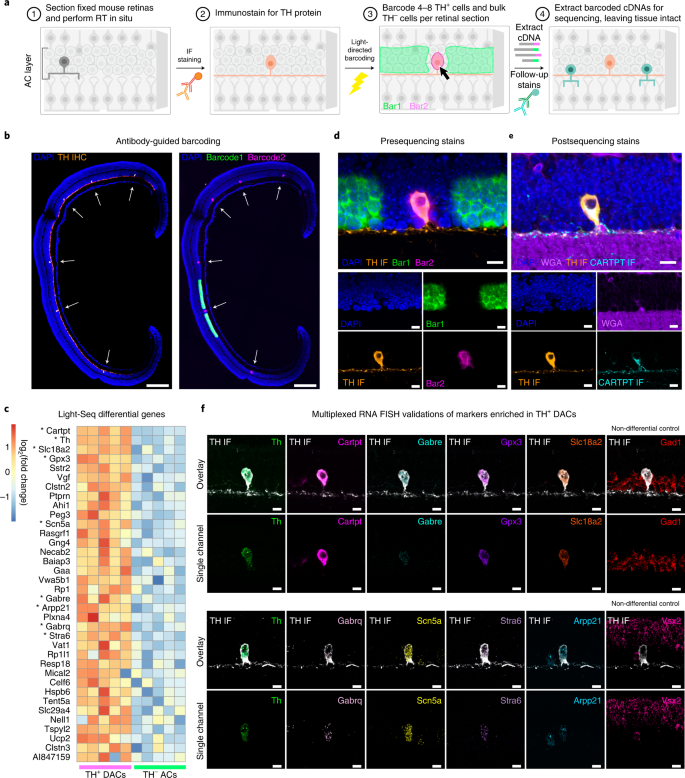
对于只有少量感兴趣的细胞存在和/或空间环境对其识别至关重要的应用,捕获转录组仍然是一个主要的挑战。为了测试Light-Seq在罕见细胞成像、条形码和测序方面的实用性,我们瞄准了小鼠视网膜中极其罕见且难以分离的dac。DACs占视网膜细胞的比例≤0.01%gydF4y2Ba52gydF4y2Ba并散布在不同类型的神经元中。之前的工作旨在用单细胞RNA-seq分析dacgydF4y2Ba53gydF4y2Ba或解离后微阵列捕获gydF4y2Ba54gydF4y2Ba取得了有限的成功。为了找到DACs的亚型特异性生物标志物,我们固定并切片小鼠视网膜,并首先进行原位RT(图2)。gydF4y2Ba5gydF4y2Ba,步骤1),然后进行IF检测已知的DACs标记物tyrosine羟化酶(TH),定位到DACs进行条形码(步骤2),然后在TH IF信号的引导下进行两轮条形码(图2)。gydF4y2Ba5 bgydF4y2Ba和扩展数据图。gydF4y2Ba6gydF4y2Ba),对TH内的cdna进行条形码gydF4y2Ba−gydF4y2Ba带有fitc标记的条形码链(每个切片大约500个细胞)和所有TH的细胞体的dacgydF4y2Ba+gydF4y2Ba带有cy3标记条形码链的dac(每节4-8个细胞),gydF4y2BangydF4y2Ba= 5个切片重复)(步骤3)。最后,从组织中移出条形码cdna,准备测序,样品保持完整,用于测序后染色(步骤4)。gydF4y2Ba
一个gydF4y2Ba,在罕见TH上执行Light-Seq的工作流gydF4y2Ba+gydF4y2BaAC亚型,dac:(1)小鼠视网膜固定、冷冻、冷冻切片。(2)原位RT后,切片用针对TH蛋白的抗体染色,标记dac(橙色)。(3) TH条码gydF4y2Ba−gydF4y2Ba具有fitc条形码链(Bar1)和TH的acgydF4y2Ba+gydF4y2Ba在抗体染色的引导下,用cy3条形码链(Bar2)进行两轮光定向条形码。(4)条形码后,将dna移位进行测序,使样品完整,以便在同一细胞上进行进一步染色。gydF4y2BabgydF4y2Ba,代表图像(gydF4y2BangydF4y2Ba= 5个重复),条形码前用抗th抗体(橙色)和DAPI(蓝色)染色。每次复制只有4到8个TH个体gydF4y2Ba+gydF4y2Ba鉴定出dac,并用Bar2(洋红色)对其细胞体进行条形码,共占每切片所有细胞的0.01-0.02%,约300 THgydF4y2Ba−gydF4y2Baac用Bar1编码(绿色)。比例尺为200微米。gydF4y2BacgydF4y2Ba差异表达分析显示,有36个转录本富集于dac (gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05;多重假设检验的双侧Wald检验及Benjamini-Hochberg调整;带对数的基因gydF4y2Ba2gydF4y2Ba(折叠变化)> 1显示;参见源数据)gydF4y2BangydF4y2Ba= 5个技术重复。*选择标记基因进行进一步验证(loggydF4y2Ba2gydF4y2Ba(折叠变化)> 3和gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05)。gydF4y2BadgydF4y2Ba荧光标记条形码(Bar1, Bar2)揭示了条形码cdna相对于TH IF的位置。比例尺为10µm (gydF4y2BangydF4y2Ba= 5个重复,每个重复4-8个THgydF4y2Ba+gydF4y2Ba每节单元格)。gydF4y2BaegydF4y2Ba, cdna移位和测序后,除了原始的TH IF和DAPI标记外,还对相同的完整切片进行了膜标记(WGA)和已知的经IF标记的DACs标记(CARTPT,青色)染色。gydF4y2BafgydF4y2Ba,带日志的标记gydF4y2Ba2gydF4y2Ba(折叠变化)> 3和gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba在新样本中使用TH IF和RNA-FISH验证< 0.05。Nondifferential控制,gydF4y2BaGad1gydF4y2Ba而且gydF4y2BaVsx2gydF4y2Ba,也被检测到在TH中显示FISH标记gydF4y2Ba−gydF4y2Baac和其他视网膜细胞。上行为TH IF覆盖RNA检测,下行为单个RNA- fish通道。比例尺为10微米。代表图像gydF4y2BangydF4y2Ba=每个标记重复3-4个切片。gydF4y2Ba
光定向条形码允许精确标记单个THgydF4y2Ba+gydF4y2Ba致密组织环境中的dac(关于光学系统的注释,请参见gydF4y2Ba方法gydF4y2Ba及补充说明gydF4y2Ba4gydF4y2Ba).虽然一些光散射可以诱导出roi外交联,这种影响可以通过绘制比预期roi略小的掩模来缓解:用于条形码THgydF4y2Ba+gydF4y2Ba在细胞边界内1-3µm处绘制DACs、掩膜(扩展数据图。gydF4y2Ba6 a - cgydF4y2Ba).使用基于激光的点扫描显微镜较慢,但提供更高的条形码分辨率,并可用于DMD(扩展数据图。gydF4y2Ba1 egydF4y2Ba).gydF4y2Ba
测序深度为每个重复350 - 500万个reads(一个MiSeq池用于5个成功扩增的片段重复;扩展数据图。gydF4y2Ba6 dgydF4y2Ba)每10 × 10-µm产生6000 - 10,000个UMIsgydF4y2Ba2gydF4y2BaTH单位面积gydF4y2Ba+gydF4y2BaDACs,平均每个单元有7800个UMIs(补充表gydF4y2Ba4gydF4y2Ba).我们再次观察到良好的基因体覆盖率和重复间一致的RPKM(扩展数据图)。gydF4y2Ba7得了gydF4y2Ba).差异表达分析显示有36个基因在TH中显著富集gydF4y2Ba+gydF4y2Ba人口(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05, loggydF4y2Ba2gydF4y2Ba(fold enrichment) > 1),包括已知标记TH (gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba= 1.32 × 10gydF4y2Ba−24gydF4y2Ba)及CARTPT (gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba= 4.98 × 10gydF4y2Ba−65gydF4y2Ba)gydF4y2Ba54gydF4y2Ba(无花果。gydF4y2Ba5度gydF4y2Ba以及附带的源数据)。要验证顶级生物标志物(gydF4y2BaCartptgydF4y2Ba)测序后,我们重新访问了存储的样本,并对相同的细胞进行了CARTPT IF,揭示了特定的标记(图。gydF4y2Ba5 d, egydF4y2Ba).gydF4y2Ba
为了进一步验证,在新的视网膜切片中进行RNA-FISH,以检测顶级差异表达基因(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05和loggydF4y2Ba2gydF4y2Ba(折叠富集)> 3)。在所有情况下,RNA-FISH均证实了Light-Seq结果,显示TH中标记物富集gydF4y2Ba+gydF4y2Ba相对于邻近TH的dacgydF4y2Ba−gydF4y2BaACs(无花果。gydF4y2Ba5 fgydF4y2Ba).作为对照,我们检测gydF4y2BaGad1gydF4y2BamRNA,在TH和TH中均有已知表达gydF4y2Ba+gydF4y2Badac和THgydF4y2Ba−gydF4y2BaACs,gydF4y2BaVsx2gydF4y2BamRNA,在两种人群中都不表达。重要的是,FISH数据表明一些标记具有高表达水平(gydF4y2BaCartptgydF4y2Ba,gydF4y2BaSlc18a2gydF4y2Ba),而其他人的表达则较为温和(gydF4y2BaGabregydF4y2Ba,gydF4y2BaGabrqgydF4y2Ba,gydF4y2BaStra6gydF4y2Ba),证明Light-Seq可以准确检测中等表达的rna。gydF4y2Ba
一些Light-Seq富集标记物先前已在小鼠TH中报道过gydF4y2Ba+gydF4y2Badac:gydF4y2BaCartptgydF4y2Ba54gydF4y2Ba,gydF4y2BaSlc18a2gydF4y2Ba(gydF4y2BaVMAT2gydF4y2Ba)gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba和γ -氨基丁酸(GABA)受体亚基gydF4y2Ba加布尔和加布尔gydF4y2Ba57gydF4y2Ba.其他成绩单,包括gydF4y2BaStra6gydF4y2Ba,gydF4y2BaGpx3gydF4y2Ba,gydF4y2BaArpp21gydF4y2Ba,gydF4y2BaScn5agydF4y2Ba据我们所知,这里发现的大多数生物标志物还没有关于这种亚型的报道。gydF4y2Ba
讨论gydF4y2Ba
在这里,我们提出了light - seq,一种使用光将可排序的空间指数附加到完整样品中的生物分子上的方法。通过直接整合两个强大的域,显微镜和NGS, Light-Seq能够将靶细胞的形态和空间参数与其转录组谱连接起来。捕捉形态、组织背景、转录组和蛋白表达在同一细胞提供了一个更全面的测量细胞的状态和它们的相互作用。gydF4y2Ba
我们证明了Light-Seq可以用于固定组织切片内4 - 1000 +细胞群体的全转录组分析,其灵敏度与现有方法相似。Light-Seq生产的UMI产量为1000 - 10,000 / 10 × 10-µmgydF4y2Ba2gydF4y2Ba单位面积取决于目标细胞类型,与DBIT-Seq相当(~5,000 UMIs)gydF4y2Ba17gydF4y2Ba和Slide-SeqV2(500-1,000个ui)gydF4y2Ba12gydF4y2Ba相同大小的组织面积(补充表格)gydF4y2Ba3 gydF4y2Ba而且gydF4y2Ba4gydF4y2Ba).与smFISH测量相比,单个基因的Light-Seq转录检测灵敏度在1% - 10%之间,平均值为4.29±3.39%,与同一组基因的单细胞RNA测序灵敏度相似(图2)。gydF4y2Ba4 egydF4y2Ba和扩展数据图。gydF4y2Ba4 h-jgydF4y2Ba).即使我们保守的管道只包括唯一映射到基因组和外显子特征的reads, Light-Seq的灵敏度也符合现有的空间测序方法(0.005-15.5%(参考文献)。gydF4y2Ba17gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba),如上文所述gydF4y2Ba9gydF4y2Ba).我们注意到,灵敏度和单位面积的UMIs测量通常受到特定细胞和组织类型、基因测定和序列处理管道的高度影响,这使得跨技术和应用的直接比较不完善。gydF4y2Ba
我们希望通过原位RT和条形码的优化,如蛋白酶处理、抗原提取或改变固定/渗透条件,Light-Seq的灵敏度可以进一步提高gydF4y2Ba21gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,使用有针对性的ISH探针gydF4y2Ba61gydF4y2Ba以及靶向核糖体RNA损耗。这些改进,结合定制掩模的灵活性,最终可以以更高的效率对单细胞或亚细胞隔间进行分析。我们将继续更新应用Light-Seq的详细协议gydF4y2Balightseq.iogydF4y2Ba,包括对不同类型组织的优化建议。gydF4y2Ba
尽管使用不同的RNA检测方法,我们观察到来自相同细胞类型的单细胞Drop-Seq和Light-Seq数据中差异富集基因之间有很强的一致性,例如,在两个数据集中,98.6%的显著差异基因富集于相同的视网膜细胞群(扩展数据图)。gydF4y2Ba4 dgydF4y2Ba).Drop-Seq数据来源于许多视网膜中的分离细胞,而Light-Seq需要的细胞输入要少得多(仅从4个18µm切片中选择细胞),并且不受分离时细胞损失的影响,也不会产生选择偏差。gydF4y2Ba
许多现有的空间转录组学方法既复杂又昂贵,不管生物学问题的兴趣。使用标准的NGS, Light-Seq绕过了许多与原位测序和FISH方法相关的挑战,这些方法受到组织自身荧光和图像反卷积的限制。Light-Seq的测序输出不仅可以检测RNA的存在,还可以检测精确的序列信息,如单核苷酸多态性和剪接异构体,与大多数ISH方法相比具有主要优势(补充说明)gydF4y2Ba3 gydF4y2Ba).这种方法对于追踪癌症中的突变和克隆性或解决组织样本中的微生物物种特别有用。每个切片上的RT、a -tail、三轮条形码、位移、交叉结合成和PCR的成本仅为约34.50美元(补充表gydF4y2Ba5gydF4y2Ba)。光交联可用于任何标准光学成像装置,可以将UV光聚焦在指定区域(例如,带有365纳米UV LED和DMD附件的显微镜,或共聚焦扫描上405纳米激光,甚至用于三维应用的光片显微镜)。gydF4y2Ba
DSP平台通过使用迭代的紫外裂解和释放条形码的微毛细管收集,为空间定位提供了类似的灵活性gydF4y2Ba24gydF4y2Ba但该平台需要昂贵的设备和靶向杂交的mrna条形码,目前提供的灵敏度较低,至少需要20-300个细胞进行测序。另一种方法,PIC,同样利用了光定向ROI目标,并显示出更高的灵敏度与单细胞条形码,但是单一的和破坏性的样本。这些方法包括样品制备和UMI恢复方案的差异,这可能有助于考虑Light-Seq的未来变化。gydF4y2Ba
通过在空间上限制条形码到目标cdna,并选择性地放大它们以生成文库,Light-Seq允许测序读取集中在感兴趣的细胞上。因此,与表面捕获或基于微流控通道的方法不同,测序深度和成本可以灵活优化,特别是针对极少数细胞的实验。这种优势在我们罕见的细胞实验中是显而易见的(图。gydF4y2Ba5gydF4y2Ba),其中我们对所有复制使用了一个MiSeq池运行(2000万次读取)。通过实验分离成像和测序也使工作流程高度灵活,并且光定向条形码允许使用相同的试剂和实验策略来处理不同规模的roi(从亚细胞结构到大型超细胞区域)。gydF4y2Ba
我们演示了Light-Seq如何为研究非常罕见的细胞群提供简单和可定制的工作流程。我们发现并验证了先前未知的TH生物标志物gydF4y2Ba+gydF4y2Badac,在之前的尝试中没有被捕捉到转录分析这个种群gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba.我们的数据证实了之前已知的标记,例如gydF4y2BaThgydF4y2Ba,gydF4y2BaCartptgydF4y2Ba而且gydF4y2BaSlc18a2gydF4y2Ba,也为理解多巴胺能视网膜神经元的生物学提供了新的线索。其中的几个,例如gydF4y2BaArpp21gydF4y2Ba(ref。gydF4y2Ba62gydF4y2Ba),gydF4y2BaVgffgydF4y2Ba63gydF4y2Ba而且gydF4y2BaGpx3gydF4y2Ba(ref。gydF4y2Ba64gydF4y2Ba),被认为在多巴胺能神经元或神经系统其他地方的相关疾病中发挥作用,但许多仍未被研究。其中一个新颖的标志是gydF4y2BaStra6gydF4y2Ba编码一种跨膜维生素a转运蛋白,双向运输视黄醇。有趣的是,该基因在视网膜色素上皮细胞中被额外表达,并与视网膜疾病有关,但TH的作用gydF4y2Ba+gydF4y2Ba疾病表型中的dac仍未被探索gydF4y2Ba65gydF4y2Ba,gydF4y2Ba66gydF4y2Ba.抗河豚毒素电压门控钠通道的发现,gydF4y2BaScn5agydF4y2Ba,是令人惊讶和有趣的。gydF4y2BaScn5agydF4y2Ba编码心脏主要钠通道,gydF4y2BaNav1.5gydF4y2Ba,在神经元群体中很少被检测到gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba.在这里,我们报道gydF4y2BaScn5agydF4y2Ba在THgydF4y2Ba+gydF4y2Ba但其在这些细胞中的功能仍有待进一步研究。gydF4y2Ba
Light-Seq的非破坏性特性使测序后的样品完整无损,增强了来自同一细胞的多组学测量。我们还设想Light-Seq可以用于基于地标的转录组学(如APEX-Seq)gydF4y2Ba69gydF4y2Ba),无需基因干预和样本破坏。虽然Light-Seq的灵敏度、空间分辨率和易用性综合起来优于现有技术,但与其他空间方法相比,目前可寻址区域的数量有限。我们期望增加多路复用将是高度可行的,因为其他已发表的方法(例如,SABERgydF4y2Ba49gydF4y2Ba,gydF4y2Ba70gydF4y2Ba, CycIFgydF4y2Ba71gydF4y2Ba或法典gydF4y2Ba72gydF4y2Ba)通过重复标记、成像和去杂交/漂白进行更长的(多达60个)连续循环,并在更多的条形码轮中为良好的组织保存提供了强有力的前期证据。在未来的应用中,扩展到组合条形码构建和其他生物分子(例如,蛋白质组,表观基因组)的标记,可以支持高通量条形码和数百到数千个细胞或区域的筛选,成本增加极小,对进一步开发具有极大的兴趣。gydF4y2Ba
方法gydF4y2Ba
低聚糖的设计和制备gydF4y2Ba
含cnvk的条形码序列用NUPACK进行筛选gydF4y2Ba73gydF4y2Ba具有最小的二级结构,并使用BLAST与小鼠和人类基因组进行了对比gydF4y2Ba74gydF4y2Ba和咩咩的叫声gydF4y2Ba75gydF4y2Ba在线工具。对于原位转录组条形码,一个RT引物以5个n和3个g结尾gydF4y2Ba40gydF4y2Ba设计为包含与条形码序列中的共享区域互补的对接序列(补充图。gydF4y2Ba3 gydF4y2Ba).条形码寡核苷酸从Gene Link订购,所有剩余的寡核苷酸从Integrated DNA Technologies订购。100 μM的低聚糖储存在−20°C, 10 μM的工作液储存在IDTE (Integrated DNA Technologies cat。不。11-01-02-02)是为最常用的低聚物制备的。所有序列、纯化和供应商信息均列在补充表中gydF4y2Ba5gydF4y2Ba而且gydF4y2Ba6gydF4y2Ba.gydF4y2Ba
细胞培养gydF4y2Ba
从GenTarget购买了稳定的HEK293-GFP细胞系(SC001),在CMV启动子下组成性地表达eGFP,并在高糖D-MEM中培养,使用GlutaMax补充10%胎牛血清,0.1 mM MEM非必需氨基酸,1% pens - strep (Thermo cat。不。15140122)和10µg mlgydF4y2Ba−1gydF4y2Bablasticidin(热猫。不。J67216)。从ATCC (CRL-1658)购买小鼠3T3细胞系,在D-MEM中与GlutaMax (Thermo cat。no.10569-010)中添加10%犊牛血清(ATCC 30-2030)和1% Pen-Strep。在细胞混合实验中,细胞在D-MEM中接种过夜,谷氨酰胺补充10%犊牛血清、1% Pen-Strep和0.1 mM MEM非必需氨基酸(Thermo cat。没有11140 - 050)。gydF4y2Ba
组织gydF4y2Ba
所有动物实验均按照哈佛大学动物保护与使用委员会批准的IS00001679协议进行。实验采用出生后第18天野生型CD1 IGS小鼠(Charles River, Strain Code 022)的组织。gydF4y2Ba
条码设置gydF4y2Ba
条形码在倒置尼康Eclipse Ti-E显微镜上进行,并附有Mightex Polygon 400 DP DMD。一个Mightex bls系列高功率液体光导耦合LED光源,365 nm, 50 w发射器,在10%的功率下使用10 s,通过CFI计划fluor 10×物镜每个选定的ROI每个条形码轮(图中除外)。gydF4y2Ba2 bgydF4y2Ba其中10%的功率应用5秒)。对于玻璃表面或细胞培养交联实验,重点设置为gydF4y2BazgydF4y2Ba-与玻璃罩上表面对应的位置。用于TH的视网膜层实验和条形码gydF4y2Ba−gydF4y2Baac,聚焦在玻璃表面上方10µm处。对于THgydF4y2Ba+gydF4y2BaDAC群体,为每个细胞找到th抗体染色的焦平面,在5µm以上的平面上进行条形码,以细胞中间为目标。光交联是在单个上完成的gydF4y2BazgydF4y2Ba飞机。细胞混合和视网膜实验的掩膜使用贝塞尔ROI工具手工绘制,并使用尼康Elements (v.4.51) Polygon 400模块用户界面设置为刺激区域。请参阅我们的协议gydF4y2Balightseq.iogydF4y2Ba及补充说明gydF4y2Ba4gydF4y2Ba详细的条形码细节,包括校准和优化光学设置。gydF4y2Ba
视网膜组织条形码和免疫荧光gydF4y2Ba
从出生后第18天开始,用1× PBS (Invitrogen AM9625,超纯水稀释,Invitrogen猫)解剖小鼠神经视网膜。不。10977),并立即在1× PBS与4%多聚甲醛(从16%溶液中稀释,Thermo Scientific cat)室温下固定25分钟。不。28908)和0.25% Triton X-100 (Sigma Aldrich T8787)。然后,视网膜在1× PBS中洗涤3 × 5分钟,在1× PBS中7%蔗糖中洗涤一次10分钟(40 μ m过滤灭菌),然后将视网膜嵌入OCT (Tissue-Tek 4583)和30%蔗糖在1× PBS中1:1的溶液(v:v)中,在冷冻模具中冷冻,随后在−80°C保存。进行冷冻切片,将18µm的切片切到poly上gydF4y2BalgydF4y2Ba-赖氨酸涂层18孔ibidi室载玻片(ibidi定制订单猫。不。81814, #1.5聚合物)。视网膜层的比较(图。gydF4y2Ba4gydF4y2Ba),将同一动物的四个不同视网膜切片切入不同孔中,制备四个技术重复。为了促进组织的粘附,在冷冻切片前,在腔室载玻片上涂上一层额外的聚gydF4y2BadgydF4y2Ba-赖氨酸(PDL) (Sigma P6407)在0.3 mg ml溶解gydF4y2Ba−1gydF4y2Ba2×硼酸缓冲液(稀释于Thermo Scientific PI28341水中,alialix并保存在−20°C)。用无菌PDL溶液在4℃下覆盖腔室2小时,去除溶液并完全干燥,用超纯水清洗一次即可完成PDL涂层。冷冻切片后,将视网膜短暂干燥(~10分钟),并在0.1% Tween-20 (vol/vol, Sigma Aldrich cat)的1× PBS中洗涤三次。不。P9416-50ML) (PBST)。gydF4y2Ba
然后将缓冲液替换为由300 μ M dNTPs (NEB N0447S)、0.5% Triton X-100 (vol/vol)、6 mM RNaseOUT (Invitrogen cat。不。10777019), 1µM RT引物和8 UµlgydF4y2Ba−1gydF4y2BaMaxima RT H减酶(Thermo Scientific猫。不。FEREP0753)在1× RT缓冲液中,并在平顶热循环器(Mastercycler Nexus Flat, Eppendorf cat)上孵育。不。6335000020)使用以下程序:在22°C下进行30分钟,然后进行12个周期的斜坡程序:8°C 30秒,15°C 30秒,25°C 30秒,30°C 1分钟,37°C 1分钟和42°C 2分钟。在42°C孵育30分钟后,样品在4°C临时保存。RT后,样品在含60%去离子化甲酰胺的PBST中洗涤(vol/vol, Thermo Scientific cat。不。AM9342)浸泡3 × 5分钟,在PBST中加入1 M NaCl (Invitrogen AM9760G)浸泡2 × 2分钟,在PBST中浸泡2 × 2分钟。样品保存在PBST中,直到缓冲液与由1× ThermoPol buffer (NEB cat。 no. B9004S), 1 mM dATP (NEB N0446S), 25 µM ddATP (Sigma GE27-2051-01) and 1,000 U ml−1gydF4y2Ba末端转移酶(NEB M0315L)在37°C孵育45分钟。a尾后,样品在PBST中洗涤3 × 1 min,在PBST中4℃保存过夜。第二天,在条形码前,去除PBST,加入1 M NaCl的新鲜PBST。用于条形码杂交,由2 mg ml的PBST组成的条形码溶液gydF4y2Ba−1gydF4y2Ba剪切的三文鱼精子DNA (Invitrogen AM9680), 10%硫酸右旋糖酐(wt/vol, Sigma Aldrich cat)。不。S4030), 250 nM Barcode 1股(GATE.D12.B1)和500 mM NaCl作用30分钟。然后用PBST和1 M NaCl洗涤样品(3 × 1 min)。最后一次清洗缓冲液替换为新鲜PBST和1 M NaCl,然后进行条形码处理。然后将载玻片转移到显微镜下进行光定向条形码。在明场图像中视觉识别所需区域,并将手绘掩模设置为光刺激区域gydF4y2Ba条码设置gydF4y2Ba部分)。在感兴趣的区域进行光刺激后,将腔室从显微镜中取出进行清洗。然后用含60%去离子甲酰胺(vol/vol)的PBST洗涤8次(两次缓冲液交换4个循环,中间孵养5分钟),再用含1 M NaCl的PBST洗涤(2 × 2 min)。最后一次清洗缓冲液替换为新鲜PBST和1 M NaCl,用于下一轮条形码。然后在显微镜上手动找到相同的组织区域,并使用条形码2 (GATE.D12.B2)和3 (GATE.D12.B3)进行另外两轮条形码。在最后一轮编码和清洗后,用PBST替换缓冲区。gydF4y2Ba
条形码视网膜用DAPI染色30分钟(0.5µg mlgydF4y2Ba−1gydF4y2Ba在PBST)和成像(图。gydF4y2Ba4gydF4y2Ba和扩展数据图。gydF4y2Ba3 gydF4y2Ba).成像后,用67.5µl由1× ThermoPol缓冲液和250 U ml组成的置换混合物处理样品gydF4y2Ba−1gydF4y2BaRNase H (NEB猫;不。M0297L), 37℃孵育45分钟。为了最大限度地提高回收率,首先在20 nM溶液中上下移液,在低保留率的移液器尖端涂上底漆,然后在收集之前,使用相同的空尖端在井内上下移液几次。然后将每个洗脱液转移到含有1.6µl 1µM交叉结合成引物(GATC.20T)的管中,混合并在PCR机(Eppendorf Mastercycler Nexus Gradient)中在75°C下热灭活20分钟。然后,10.9 μ l交叉结合成混合物含有1.15 × ThermoPol缓冲液,734 μ M dNTPs和5,872 U mlgydF4y2Ba−1gydF4y2BaBST LF聚合酶(NEB cat。不。将M0275L)加入洗脱液中。样品在37°C的PCR机中孵育30分钟,然后在80°C热失活20分钟。gydF4y2Ba
提取后,冷冻切片在PBST中洗涤两次,并保存在新鲜PBST中4°C,直到进一步分析。视网膜用组织阻断液(1× PBS, 0.3% Triton X-100和5%正常驴血清(Jackson免疫研究猫)孵育。不。017-000-121, RRID:AB_2337258)和过滤器-使用40 μ m注射器过滤器消毒)在室温下1小时,然后进行抗体染色。在组织阻断液中制备一抗:绵羊抗chx10 (Exalpha X1180P, RRID:AB_2314191,稀释1:500)和兔抗pax6 (Abcam cat。不。ab195045, RRID:AB_2750924,稀释1:300)。一抗4℃孵育过夜,用PBST冲洗5 × 5 min。二抗(驴抗羊- alexa647,杰克逊免疫研究猫。不。 713-605-147, RRID:AB_2340751; donkey anti-rabbit-Cy3, Jackson ImmunoResearch cat. no. 711-165-152, RRID:AB_2307443) were incubated overnight at 4 °C in Blocking Solution (both antibodies diluted 1:500 from 50% glycerol stock) and washed 5 × 5 min with PBST. Wheat germ agglutinin (WGA, Biotium cat. no. 29022-1) diluted 1:100 in PBST from a 1 mg ml−1gydF4y2Ba在室温下应用原液1小时,然后用PBST中的DAPI(0.5µg mlgydF4y2Ba−1gydF4y2Ba).gydF4y2Ba
交叉结合成后,对提取的序列进行定量,然后按如下方法进行批量扩增。首先,将5µl体积的反应以1:1的比例与由Sybr Green I (1×, Invitrogen S7563)、Kapa HiFi Buffer (2×,来自Roche KK2502)、正向和反向引物(每个600 nM, GATE和GATC序列)组成的PCR混合物组合;参见补充表gydF4y2Ba6gydF4y2Ba), dNTPs(600µM, from Roche KK2502)和Kapa HiFi热启动聚合酶(0.04 UµlgydF4y2Ba−1gydF4y2Ba,来自Roche KK2502)。样品在定量PCR仪(Biorad CFX Connect Real-Time System)上按以下程序扩增:98°C 3 min,然后98°C 20 s, 60°C 30 s, 72°C 2 min,循环30次。然后,样品在72°C下孵育5分钟,并进行熔体曲线测量。在定量的基础上,使用所有剩余的反应材料在相同的条件下进行体扩增20个循环。gydF4y2Ba
分别制备了四个技术重复的提取材料,然后将样本集中起来进行测序,如下所示(gydF4y2Ba文库制备和测序gydF4y2Ba).占切片总组织面积的7-11%(补充表gydF4y2Ba3 gydF4y2Ba)根据条形码区域在整个视网膜组织面积上的大小,估计是用条形码标记的。使用掩膜面积测量条形码面积,在斐济用徒手面积工具测量视网膜组织总面积gydF4y2Ba76gydF4y2Ba.gydF4y2Ba
条码THgydF4y2Ba+gydF4y2BadacgydF4y2Ba
视网膜按照上述方法制备并切片,重复6个切片。对图中的视网膜层进行如上所述的Light-Seq。gydF4y2Ba4gydF4y2Ba原位RT和a -tail后,条形码前,切片用抗th抗体(Millipore cat。不。AB152, RRID:AB_390204)室温放置1小时。抗体在由1× PBS、0.1% Tween-20和1%分子级BSA (GeminiBio, 700-106 P)制成的阻断液中1:500稀释,并使用20 μ m注射器过滤器过滤灭菌。用PBST冲洗样品5 × 5 min。驴抗兔- alexa647二抗(Jackson免疫研究猫。不。711-605-152, RRID:AB_2492288)以1:250稀释的比例加入相同的堵塞液中,室温下浸泡30 min,然后用PBST冲洗5 × 3 min。gydF4y2Ba
然后按照上面描述的方式进行条形码编码,只做了一些小改动,如下所示。第一轮针对的是fitc条形码链(条形码3,GATE.D12。B3序列,标注为图中条码1。gydF4y2Ba5gydF4y2Ba)至THgydF4y2Ba−gydF4y2BaACs,第二轮针对Cy3-barcode链(Barcode 2, GATE.D12.B2)到THgydF4y2Ba+gydF4y2Badac(见补充表)gydF4y2Ba6gydF4y2Ba对于条形码序列)。的gydF4y2BazgydF4y2Ba用于TH条形码的焦平面gydF4y2Ba+gydF4y2BaDACs由抗th抗体信号引导,并在距离井面5µm的地方进行额外调整。在含有60%甲酰胺的PBST中进行定向条形码后,严格的清洗次数增加了一倍(4个5分钟的培养周期,其中每个周期包括4次缓冲交换,而不是视网膜层的两次缓冲交换)。所有THgydF4y2Ba+gydF4y2Ba同一部分的dac在多个视野中单独处理,并在人工选择后与条形码2交联。在制备的6个切片重复中,5个在提取序列的PCR扩增过程中产生了足够的测序材料。对于所有重复,TH的数量gydF4y2Ba+gydF4y2BaDACs在4到8之间,因此THgydF4y2Ba+gydF4y2BaDAC转录组来自每个技术复制的4到8个细胞池。gydF4y2Ba
TH测序后免疫染色gydF4y2Ba+ /−gydF4y2BaACsgydF4y2Ba
测序后,我们重新访问了来自两个原始切片的相同条形码细胞,以验证顶部命中(最低)的富集表达gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba),gydF4y2BaCartptgydF4y2Ba在蛋白质水平上。样品在4°C的PBST中保存>10天,同时进行测序和分析。为了染色,样品与山羊抗cartt抗体(赛默飞世尔科学猫)孵育。不。PA5-47170, RRID:AB_2607700), 1:20在封闭液中室温浸泡1.5小时,然后用PBST冲洗5 × 5分钟。然后,驴抗山羊alexa488二抗(杰克逊免疫研究猫。不。705-545-003, RRID:AB_2340428)以1:250的比例加入相同的堵塞液中,室温下静置30 min,然后用PBST冲洗5 × 3 min。wga -罗丹明(矢量实验室猫。不。 RL-1022-5, 1:100 from 1 mg ml−1gydF4y2Ba二抗混合液中加入DAPI标记细胞膜和细胞核。gydF4y2Ba
TH的RNA-FISH验证gydF4y2Ba+gydF4y2BaDAC标记gydF4y2Ba
对于排名前十的标记(与gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05, loggydF4y2Ba2gydF4y2Ba(fold change) > 3),我们使用RNA-FISH进行验证。如前所述,我们使用系列SABER-FISH检测新的25µm视网膜切片中的标记物gydF4y2Ba49gydF4y2Ba.FISH验证是在用于Light-Seq实验的源动物的一窝小鼠的四个新切片上进行的。SABER-FISH探针使用PaintSHOP工具的“RNA探针设计”功能设计(gydF4y2Bahttps://oligo.shinyapps.io/paintshop/gydF4y2Ba)gydF4y2Ba77gydF4y2Ba.对于每个基因,所有探针都附加一个共同的基因特异性的SABER-FISH引物序列,用于同一细胞中多个基因的正交检测。参见补充表gydF4y2Ba7gydF4y2Ba针对每个基因的探针序列(及其附加引物)。先前描述的探测集用于gydF4y2BaGad1.26gydF4y2Ba而且gydF4y2BaVsx2.25gydF4y2Ba(ref。gydF4y2Ba49gydF4y2Ba).如前所述,进行了SABER-FISH探针制备和RNA检测(由Kishi等人,2019年,补充方案的“用户友好协议:视网膜组织切片RNA- fish”部分)gydF4y2Ba49gydF4y2Ba).的gydF4y2BaSstr2gydF4y2Ba.25 SABER-FISH探针组在探针合成反应期间未能延伸(在应用于组织样本之前),因此被排除在验证实验之外。gydF4y2Ba
探针分为两组进行多路检测,进行三轮荧光检测,每个样品中共捕获6个rna(代表性图像如图中行所示)。gydF4y2Ba5 fgydF4y2Ba).如前所述,每一轮使用ATTO565和Alexa647荧光寡核苷酸检测两个不同的基因gydF4y2Ba49gydF4y2Ba(序列见补充表gydF4y2Ba7gydF4y2Ba).TH抗体染色是在第一轮荧光检测后进行的,如上一节所述,因此在所有轮连续成像中都存在。WGA-405S (Biotium cat。不。在1:100的二抗孵育中加入29022-1)进行膜染色。gydF4y2Ba
培养细胞的条形码和免疫荧光gydF4y2Ba
Eighteen-well保利-gydF4y2BalgydF4y2Ba赖氨酸涂层的伊比迪室(伊比迪定制订购猫。不。81814)用无菌PDL (Sigma Aldrich cat。不。P6407) 0.3 mg mlgydF4y2Ba−1gydF4y2Ba4°C过夜。然后,干燥1小时,用超纯水(Invitrogen cat。不。10977)并在播种前再次干燥。然后将每孔约4000个HEK293和约5000个NIH/3T3细胞播种到培养箱中(37°C, 5% CO)gydF4y2Ba2gydF4y2Ba一夜之间)。样品用DPBS(预温至37°C, Gibco cat)轻轻清洗。不。14190-144)和固定在4%甲醛(wt/vol,热猫。不。28908)在1× PBS (Invitrogen cat。不。AM9625)室温10分钟。然后用1× PBS洗涤2次,0.25% Triton X-100 (vol/vol)渗透10 min。然后用1× PBS冲洗样品两次,原位RT步骤按照上述视网膜样品的相同方案进行。 Following RT, samples were washed in PBST and 60% deionized formamide for 3 × 2 min, in PBST with 1 M NaCl for 2 × 2 min and in PBST for 2 × 2 min. Samples were kept in PBST at 4 °C (as a pausing point in the protocol). To proceed further, the PBST buffer was exchanged with the A-tailing master mix (as described above for tissues) and incubated at 37 °C for 30 min.
a尾后,样品在PBST中洗涤3 × 1 min,在PBST中加入1 M NaCl,直至条形码。条形码杂交时,将含有250 nM barcode 1链(GATE.D12.B1)的PBST和500 mM NaCl的条形码溶液施加于样品上15 min,多余的链用PBST和1 M NaCl洗涤3 × 1 min。最后一次清洗缓冲液替换为新鲜PBST加1 M NaCl,将载玻片转移到显微镜下进行定向条形码。选定的HEK细胞然后使用手绘掩模光交联(见gydF4y2Ba条码设置gydF4y2Ba有关详情)。然后用60%去离子甲酰胺(vol/vol)的PBST洗涤样品8次(4个循环,两次缓冲交换,中间孵养2分钟),然后用PBST和1 M NaCl洗涤2 × 2分钟。在下一轮条形码2 (GATE.D12)之前,最后一次清洗缓冲液被带有1 M NaCl的新鲜PBST取代。B2, 3T3细胞),以相同的条码培养和清洗方案进行,除了所有清洗后,孔内液体更换为PBST。参见补充说明gydF4y2Ba2gydF4y2Ba有关减少非特定背景的建议协议更新,请参见gydF4y2Balightseq.iogydF4y2Ba获取最新建议的协议。gydF4y2Ba
条形码后,用67.5µl由1× ThermoPol缓冲液和250 U ml组成的置换混合物处理样品gydF4y2Ba−1gydF4y2BaRNase H (NEB M0297L),在37°C孵育30分钟。对视网膜样本进行cDNA收集、RNase H热失活和交叉结合成。gydF4y2Ba
提取cdna后,细胞在PBST中洗涤两次,然后保存在新鲜的PBST中4°C,直到进一步分析。对于细胞混合实验,在一个阱上进行多路中频。所有抗体的浓度都是1万gydF4y2BaggydF4y2Ba在4°C下浸泡10分钟后使用。用含有山羊抗lamin - b (sc-6216, RRID:AB_648156)、小鼠抗tfam (MA5-16148, RRID:AB_11157422)和大鼠抗α微管蛋白(MA1-80017, RRID:AB_2210201)的一抗混合抗体孵育细胞。不。001-000-162) 1小时。在1× PBS中洗涤2 × 1 min后,将抗小鼠alexa647 (Jackson ImmunoResearch 715-605-150, RRID:AB_2340862)、抗山羊cy3 (VWR 102649-368)和抗大鼠Alexa488 (Jackson ImmunoResearch 712-545-150, RRID:AB_2340683)二抗在含0.3% Triton X-100和5% BSA的1× PBS中孵育1小时。细胞在1× PBS 2 × 1 min中洗涤,然后用4µg ml培养gydF4y2Ba−1gydF4y2BaDAPI (Invitrogen猫)。不。D1306)在1× PBS中浸泡5min,然后用1× PBS冲洗2 × 1min,然后在新鲜的1× PBS中成像。gydF4y2Ba
交叉连接合成后,提取的序列按照上面视网膜组织条形码和免疫荧光部分定量。然后在相同的条件下进行体扩增24个循环,但从大约一半(40 μ l)的起始材料开始。保留另一半样本材料用于测试其他PCR试剂盒(本工作中图中所示的所有结果均使用Kapa HiFi试剂盒获得,我们选择Kapa HiFi试剂盒作为我们的标准扩增方法)。gydF4y2Ba
在细胞混合实验中,平均而言,在三个技术重复中,每个孔从约4500个细胞群体中收集每种类型的约25个细胞。从每个重复中分别提取并汇总测序,如下所示。总细胞数由人工计数估计约130个细胞在1毫米gydF4y2Ba2gydF4y2Ba单井面积和外推到总表面积。gydF4y2Ba
文库制备和测序gydF4y2Ba
细胞混合和视网膜组织实验的文库制备和测序相同。PCR后,样品保存在−20°C,然后用1.2×比例的Ampure XP Beads (Beckman A63881)纯化,并用水洗脱。接下来,用Nextera XT库制备试剂盒(Illumina cat。不。FC-131-1096),但i5端使用自定义引物。使用标准制造商协议和试剂(TD缓冲液,ATM, NT缓冲液,NPM PCR主混合物)在2 ng样品上进行Tagmentation,在20µl反应中,包含10µl TD缓冲液和5µl ATM,在55°C下持续5分钟,用5µl NT缓冲液停止反应,并在冰上保存。每个试管,6.5µl水,1.75µl Nextera i7底漆,1.75µl定制i5底漆(GATE*。附表中P5*gydF4y2Ba6gydF4y2Ba),加入15µl的NPM PCR master mix。反应在72°C孵育3分钟,95°C孵育15秒,然后进行12个循环:95°C孵育15秒,55°C孵育15秒,72°C孵育40秒。72°C最后孵育1分钟后,样品在10°C保存。用0.9× Ampure XP Beads纯化反应,用水洗脱。样品保存在−20°C,直到测序。gydF4y2Ba
测序由GeneWiz在Illumina HiSeq机器上进行,或由哈佛医学院生物聚合物设施在Illumina NovaSeq机器上进行,使用定制的Read 1和i5引物和30% Phi-X spike-in。对于细胞混合数据,所有复制序列都集中在HiSeq 4000流池的一个通道上,尽管一个复制也在其自己的通道上进行了测序。视网膜层实验(图;gydF4y2Ba4gydF4y2Ba),所有复制序列汇集在一起,并在NovaSeq 6000流池的两个通道中进行测序。视网膜无分泌实验(图;gydF4y2Ba5gydF4y2Ba),所有的重复聚合在一起,用一次Illumina MiSeq测序。gydF4y2Ba
体外表面条形码技术gydF4y2Ba
一个八孔ibidi ibiTreat室(ibidi猫。不。80826)被bsa -生物素和链霉亲和素功能化,用于体外表面条形码测试。bsa -生物素(Sigma A8549)和链霉亲和素(Invitrogen S-888)溶液稀释至1 mg mlgydF4y2Ba−1gydF4y2Ba0.5 mg mlgydF4y2Ba−1gydF4y2Ba分别在1 × PBS中。然后,将200 μ l bsa -生物素溶液移液到空的ibidi孔中,在室温下孵育5分钟。然后将bsa -生物素溶液吸出井外,用1 × PBS洗涤两次。将链霉亲和素溶液加入孔中,同样在室温下孵育5min,然后进行三次1 × PBS洗涤。gydF4y2Ba
功能化表面然后与生物素化链孵育(补充表gydF4y2Ba6gydF4y2Ba), 1 μM在1 × PBS中浸泡5 min,然后用1 × PBS冲洗3次。杂交溶液由200 nM CNVK条形码链在1 × PBS和1 M NaCl中组成。gydF4y2Ba
图中猫照片和彭罗斯三角形的掩模。gydF4y2Ba2 b, cgydF4y2Ba生成二进制.tif文件,然后上传并映射到DMD芯片上。猫的二值图像是使用Adobe Photoshop (v.2021)扩散抖动函数在个人相机拍摄的照片上生成的。彭罗斯三角形各个部分的掩膜是在Adobe illustrator (v.2021)中手工绘制的,并保存为8位灰度.tif文件。gydF4y2Ba
条形码链的杂交按顺序与每个对应于唯一条形码链和荧光团的掩模进行(猫掩模交联作为单轮进行(使用条形码2链)并进行交联)。每个条形码轮在杂交溶液中引入条形码链,然后在室温下孵育5分钟,并进行紫外光交联,如中所述gydF4y2Ba条码设置gydF4y2Ba上面的部分。然后,用40%甲酰胺在1 × PBS中严格清洗2 × 2分钟,去除非交联链。然后在1 × PBS和1 M NaCl中洗涤2 × 1分钟去除甲酰胺,为下一轮条形码准备室。gydF4y2Ba
成像装置gydF4y2Ba
在对照实验中监测RT、条形码和cDNA提取后的信号,我们使用ImageXpress Micro-4系统(Molecular Devices)扫描样本,该系统配备了定制的5毫米液体光导Gen III光谱led光引擎(固态377/ 54,438 / 29,475 / 28,511 / 16,555 / 28,576 / 23,635 / 22,730 /40),Semrock滤波器(零像素移位滤波器立方体:用于DAPI和750/Cy7 LED-Da/Fi/Tr/Cy5。Cy/5x-A五重奏乐队;用于488/GFP LED-FITC-A单波段;用于565/Cy3 LED-TRITC-A单波段;647/Cy5 LED-Cy5-A单波段)和由MetaXpress软件控制的Andor Zyla 4.2相机(v.6.5.3.427)。平铺以10%的重叠进行。gydF4y2Ba
全电动尼康Ti-2倒置显微镜用于成像荧光视网膜样本。该共聚焦显微镜配备了横河CSU-W1单旋转盘(50 μ m针孔尺寸)和尼康线性编码电动平台(Mad City Labs 500 μ m范围Nano-Drive Z压电插入)和Andor Zyla 4.2 plus (6.5 mm光电二极管尺寸)sCMOS相机,使用尼康Apo λS LWD 40×/1.1 DICN2水浸物镜,蔡司Immersol W 2010。在Toptica icrome MLE激光组合器中,分别使用直接调制固体激光器对405 nm、488 nm、550 nm和640 nm的样品进行照明,获得荧光。405 nm二极管100 mw(在光纤尖端)激光线、488 nm二极管100 mw激光线、561 nm 100 mw二极管泵浦固体激光线和640 nm二极管70 mw激光线。所有通道均使用硬涂层Semrock Di01-T405/488/568/647多带通二向色镜。使用16位双增益(高动态范围相机模式)捕获图像。采集时使用Nikon Elements AR 5.02软件。gydF4y2BaZgydF4y2Ba-堆叠使用压电z装置(轴向运动时快门关闭)获得。数据保存导出为ND2文件。gydF4y2Ba
扩展数据图。gydF4y2Ba1 c ggydF4y2Ba,使用徕卡SP5 X MP倒置激光扫描共聚焦显微镜使用“FRAP”模块创建自定义扫描区域。通过HCX PL APO CS ×63.0 1.20水物镜进行点扫描,并将405 nm二极管激光设置为最高功率。扫描速度设置为最慢的10hz,并在roi上扫描两次。gydF4y2Ba
图像处理与分析gydF4y2Ba
对于来自细胞混合实验的图像,使用ASHLAR对每一轮的多通道图像进行注册和拼接gydF4y2Ba78gydF4y2Ba(v.1.12.0)。Brightfield图像用于细胞混合和视网膜图像进行手动对比以获得最佳可见性。手绘细胞混合和视网膜实验的roi保存为二进制。tif文件,并使用Adobe Illustrator的图像跟踪功能转换为矢量图像,然后覆盖到明场图像上。用于细胞混合的多通道荧光图像根据以下方法进行缩放和叠加:每个图像通道的像素值通过大津阈值分离为前景像素和背景像素。然后对每张图像进行从0到1的线性归一化,将最大像素值1设置为前景像素值的第95百分位。然后将图像进行伪彩色,并使用自定义Python脚本混合成覆盖图像,该脚本相当于Adobe Photoshop的屏幕混合功能。cat和Penrose三角荧光图像手动对比以获得最佳的可见性。图中缝合的叠加图像。gydF4y2Ba3 dgydF4y2Ba是在OMERO中准备的gydF4y2Ba79gydF4y2Ba(v.5.4.6.21)。gydF4y2Ba
图中视网膜图像。gydF4y2Ba4 b, fgydF4y2Ba都是从单一的gydF4y2BaZgydF4y2Ba-平面,从斐济的复合多通道图像中提取gydF4y2Ba76gydF4y2Ba(v.2.0.0-rc-69/1.52n)。最小和最大强度级别是手动选择的,以线性缩放像素强度以获得最佳显示,并使用gydF4y2Ba图像gydF4y2Ba→gydF4y2Ba调整gydF4y2Ba→gydF4y2Ba亮度/对比度gydF4y2Ba窗口在ImageJ。扩展数据图中的单通道图像。gydF4y2Ba3 b, cgydF4y2Ba使用“自动”设置gydF4y2Ba图像gydF4y2Ba→gydF4y2Ba调整gydF4y2Ba→gydF4y2Ba亮度/对比度gydF4y2Ba,将最小强度值设为零。gydF4y2Ba
图形在Adobe Illustrator (v.2021和2022)中组装。gydF4y2Ba
测序数据处理及差异基因表达分析gydF4y2Ba
在哈佛医学院O2集群(Kernel 3.10.0)上使用Python(3.7.5)、PyTables(3.6.1)、samtools(1.9和1.12)、pysam(0.17.0)、numpy(1.21.4)、pandas(1.3.4)、Biopython(1.79)和scikit-bio(0.5.6)对测序数据进行解析和映射。看到gydF4y2Ba代码的可用性gydF4y2Ba语句用于包含所有代码和虚拟环境参数的GitHub库。序列分析的管道概述在扩展数据图中。gydF4y2Ba4gydF4y2Ba,并在扩展数据图中描述了每个复制的读分布的细目。gydF4y2Ba4 bgydF4y2Ba.使用UMI-tools (v.1.1.1)包从Read 1 (R1)读取中提取条形码、UMI和cDNA映射序列(最多40 nt)gydF4y2Ba80gydF4y2Ba.然后将序列映射到适当的基因组(人类v38或小鼠vM27)或使用STAR校准器合并的基因组gydF4y2Ba81gydF4y2Ba(v.2.7.9a),在所有情况下都丢弃multimap对齐。gydF4y2Ba
映射后,featuretsgydF4y2Ba82gydF4y2Ba使用工具将基因组映射分配给基因,然后使用UMI-tools dedup命令重复数据删除(每个基因)。对于小鼠转录作图,读取对应于两个基因(gydF4y2BaENSMUSG00000119584.1gydF4y2Ba而且gydF4y2BaENSMUSG00000064337.1gydF4y2Ba(分别为核糖体RNA和线粒体rRNA)被丢弃,因为映射序列的数量接近或超过可能的UMIs的数量,并导致重复数据删除过程停滞。对于人与细胞混合实验,未映射的reads随后用stripe Smith Waterman算法与eGFP转录本序列进行比较gydF4y2Ba83gydF4y2Ba如果他们至少有40分并且UMI是唯一的,就被认为是eGFP读数。对于小鼠和人类细胞混合实验,基于映射到合并的人类和小鼠基因组来计算判别值,并且基于分别将人类条形码读数映射到人类基因组和小鼠条形码读数映射到小鼠基因组来估计UMI计数。gydF4y2Ba
在基因分配和UMI重复数据删除之后,通过仅使用自定义Python脚本精确匹配的DNA条形码序列来解析reads。细胞混合实验的归一化表达水平计算为loggydF4y2Ba2gydF4y2Ba-转换TPM(日志gydF4y2Ba2gydF4y2Ba(tpm + 1))。视网膜组织实验用DESeq2包在R中进行基因富集分析gydF4y2Ba45gydF4y2Ba,gydF4y2Ba84gydF4y2Ba.调整后的基因被认为是富集的gydF4y2BaPgydF4y2Ba值(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba(Benjamini-Hochberg法)均小于0.05。无花果。gydF4y2Ba4 dgydF4y2Ba,每一层的标记是相对于其他两层富集的一组基因,在R 3.6.1中用pheatmap函数绘制(v.1.0.12)。无花果。gydF4y2Ba4摄氏度gydF4y2Ba,调整后的基因gydF4y2BaPgydF4y2Ba> 0.05用黑色表示(在图的底部),而在成对比较中显著富集的基因则根据其层富集程度进行着色(例如,品红,在ONL中富集,条形码1)。gydF4y2Ba
与现有的Drop-Seq数据进行比较gydF4y2Ba46gydF4y2Ba扩展数据图gydF4y2Ba4比gydF4y2Ba,伪体RNA测序数据基于单细胞计数建模,通过汇集来自已知定位于目标层内的每种细胞类型的所有细胞的计数。来自Drop-Seq数据集的单个细胞被分类为GitHub markdown中概述的细胞类型(在gydF4y2Bahttps://github.com/broadinstitute/BipolarCell2016gydF4y2Ba).将杆状和锥状感光细胞聚集在一起构建伪onl,将所有双极亚型簇和Müller胶质细胞聚集在一起构建伪bcl。每个细胞的转录本数(补充表gydF4y2Ba3 gydF4y2Ba)通过除以每一层的细胞池数量来估计。gydF4y2Ba
对于Light-Seq,每个区域的条形码单元数是根据条形码区域的面积来估计的(扩展数据图。gydF4y2Ba3 gydF4y2Ba)和人工计数条形码区域三维共聚焦图像中dapi染色核的数量。对于每一层,在条形码区域的子集(~ 5000 μm)内统计dapi染色核的数量gydF4y2Ba2gydF4y2Ba),并根据整个区域的精确大小,通过线性缩放来估计总数。由于BCL中包含双极细胞和Müller胶质细胞,BCL中双极细胞的数量估计为层内dapi染色核总数的72%gydF4y2Ba85gydF4y2Ba.在无花果。gydF4y2Ba4摄氏度gydF4y2Ba,比较两种测定方法中在ONL或BCL层中富集的标记物。gydF4y2Ba4 dgydF4y2Ba,相对于其他两层富集的标记根据它们的DESeq2绘制gydF4y2BazgydF4y2Ba分数gydF4y2Ba45gydF4y2Ba.在扩展数据图。gydF4y2Ba4 fgydF4y2Ba,绘制了两种测定中BCL富集的标记物。gydF4y2Ba
用于测序数据处理、映射和分析的脚本已经发布到GitHub,以及用于伪批量比较的细胞计数估计和运行代码的逐步说明。gydF4y2Ba
灵敏度估计gydF4y2Ba
为了评估Light-Seq的敏感性,我们比较了BCL Light-Seq数据中捕获的一组16个双极细胞标记的测序读取计数与已发表的smFISH数据gydF4y2Ba50gydF4y2Ba.在之前的文章中,我们使用smFISH每细胞点计数来估计检测到的总转录本的预期数量,这是基于Light-Seq BCL区域捕获的双极细胞数量和BCL内已知的平均每细胞转录本计数。为了估计在Light-Seq BCL区域内捕获的双极细胞的数量,我们用dapi染色的条形码图像覆盖ROI轮廓。首先,整个区域的弧长,圆弧gydF4y2Ba总计gydF4y2Ba,是在斐济使用gydF4y2Ba分段线gydF4y2Ba工具gydF4y2Ba分析gydF4y2Ba→gydF4y2Ba测量gydF4y2Ba.然后,一小部分弧长,弧gydF4y2Ba小gydF4y2Ba,用同样的方法测量(~200 μm),并计算电弧ROI内dapi染色的核数gydF4y2Ba小gydF4y2Ba区(细胞gydF4y2Ba小gydF4y2Ba).通过缩放来预测ROI中的核总数:(no。= (arc .gydF4y2Ba总计gydF4y2Ba×细胞gydF4y2Ba小gydF4y2Ba) /弧gydF4y2Ba小gydF4y2Ba(补充表gydF4y2Ba3 gydF4y2Ba).gydF4y2Ba
根据BCL中双极细胞数量的估计,我们然后使用smFISH数据对16个标记基因进行单细胞表达计数,发表在West et al. 2022(参考文献)。gydF4y2Ba50gydF4y2Ba)来预测在测量大小的双极细胞群中应该存在多少转录本。由于发表的数据是所有双极亚型的16个标记基因中的每一个的单细胞转录本(smFISH点)计数(按其测量的比例),我们选择从smFISH数据和Drop-Seq数据中排除所有BC1B型细胞,因为它们的细胞体位于AC层内,而不会在Light-Seq BCL中(如以前的工作所示,特别是Shekhar等人2016年的图3中所示(参考文献)。gydF4y2Ba46gydF4y2Ba))。为了将BC1B从West et al. 2022的smFISH单细胞基因表达矩阵中排除。gydF4y2Ba50gydF4y2Ba),我们删除了' Retina1.csv '中的所有行gydF4y2Bahttps://github.com/ewest11/Bipolar-Serial-SABER-FISH-AnalysisgydF4y2Ba),“Subtype”= 2。对于Drop-Seq数据,同样去除属于BC1B簇的细胞。gydF4y2Ba
利用所有其他双相亚型的剩余基因表达计数,我们对所有细胞进行了平均,以获得BCL的“每细胞计数的平均转录本”。然后用我们估计的细胞数(列在补充表中)对平均值进行缩放gydF4y2Ba3 gydF4y2Ba)以获得每个Light-Seq BCL复制和每个Drop-Seq复制的预期转录本数量。灵敏度用MATLAB 2018a绘制gydF4y2Ba箱线图gydF4y2Ba函数,使用默认设置。gydF4y2Ba
我们注意到,在West et al. 2022(引用。gydF4y2Ba50gydF4y2Ba)注射EdU和BrdU进行细胞出生年代测定,分离Drop-Seq细胞进行测序,这应该被视为潜在的方差来源。gydF4y2Ba
嵌合读分析gydF4y2Ba
R1和R2读取从TH开始的每个复制gydF4y2Ba+gydF4y2BaDAC实验分别进行了映射和重复数据删除。然后合并重复数据删除的R1和R2读文件,按读名称排序,并遍历R1和R2都映射到转录本的对。映射到同一转录本与不同转录本的配对数量在补充说明中报告gydF4y2Ba1gydF4y2Ba.在本分析中,Python v.3.10.4在macOS Monterey (v.12.2.1)的MacBook Pro(2021)上使用。gydF4y2Ba
基因内区分析gydF4y2Ba
为了分析内含子读取,feature的-t基因标记gydF4y2Ba82gydF4y2Ba被用来映射基因而不仅仅是外显子。然后,RSeQCgydF4y2Ba86gydF4y2Ba(v.4.0.0) read_distribution.py程序用于分析每个复制从UTR外显子、CDS外显子和10kb区域上游和下游读取的数量。详情及计数可参阅补充说明gydF4y2Ba3 gydF4y2Ba.在本分析中,Python v.3.10.4在macOS Monterey (v.12.2.1)的MacBook Pro(2021)上使用。gydF4y2Ba
基因长度偏倚分析gydF4y2Ba
由featurets报告的基因长度gydF4y2Ba82gydF4y2Ba(v.2.0.1)用于对每个复制的每个条形码的不同长度的转录本的读取计数进行分析。生成直方图以显示不同转录本长度的转录本计数分布。然后生成盒状图来显示不同长度范围内的转录本计数,并基于小鼠胚胎干细胞全长比较选择箱子gydF4y2Ba87gydF4y2Ba在R (v.4.1.3)。对于相同的箱子,RPKM值的计算方法是将读取计数除以转录本的长度(以千基为单位),然后再除以一个比例因子,该比例因子是由条件中的读取数除以100万计算出来的。gydF4y2Ba
基因体覆盖分析gydF4y2Ba
以BAM文件格式包含对齐的重复数据删除读取的文件被输入到RSeQCgydF4y2Ba86gydF4y2BageneBody_coverage.py程序生成基因体覆盖图。RseQC的参考BED文件输入是通过使用UCSC基因组浏览器托管的转换实用程序将综合基因注释GFF3文件(vM27)转换为12列BED文件而生成的gydF4y2Ba88gydF4y2Ba(特别是gff3ToGenePred和genePredToBed (v.1.04.00))。这些数据在IGV中可视化(v.2.12.3)gydF4y2Ba89gydF4y2Ba.gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
条形码实验的详细协议可在协议上在线访问。IO平台,以及最新的协议和资源可以在gydF4y2Balightseq.iogydF4y2Ba.这些方案包括:(1)原位逆转录和a尾,(2)原位空间条形码,(3)条形码cDNA序列的置换和提取,(4)交叉结合成,(5)PCR扩增和(6)通过tagmentation制备文库。原始测序数据可在NCBI的基因表达Omnibus中在线获得,并可通过GEO系列登录号访问gydF4y2BaGSE208650gydF4y2Ba.扩展数据图2的源数据中提供了人鼠细胞混合实验复制的基因映射和计数。图4的源数据提供了视网膜组织实验中各层之间富集的差异富集基因的完整列表。在TH之间富集的差异富集基因的完整列表gydF4y2Ba+gydF4y2Ba和THgydF4y2Ba−gydF4y2Ba罕见视网膜AC组织实验中的细胞见图5的源数据。所有的SABER-FISH探针序列在补充表中提供gydF4y2Ba7gydF4y2Ba.如欲索取资料,请致函j.y.k.、s.k.s.、P.Y.或C.L.C.gydF4y2Ba
代码的可用性gydF4y2Ba
代码可在GitHubgydF4y2Bahttps://github.com/Harvard-MolSys-Lab/Light-Seq-Nature-Methods-2022gydF4y2Ba.这包括图像分析的代码,序列分析和差异基因表达分析和绘图。gydF4y2Ba
参考文献gydF4y2Ba
Altemose, N. et al. μDamID:一种用于单细胞中蛋白质- dna相互作用的联合成像和测序的微流体方法。gydF4y2Ba细胞系统。gydF4y2Ba11gydF4y2Ba, 354 - 366。e9(2020)。gydF4y2Ba
张建强等。高通量单细胞连接光学和基因表达谱。gydF4y2Ba基因组医学杂志。gydF4y2Ba21gydF4y2Ba, 49(2020)。gydF4y2Ba
尼塔,N.等。智能图像激活细胞分类。gydF4y2Ba细胞gydF4y2Ba175gydF4y2Ba, 266 - 276。e13(2018).
Schraivogel, D.等。高速荧光成像细胞分选。gydF4y2Ba科学gydF4y2Ba375gydF4y2Ba, 315-320(2022)。gydF4y2Ba
Hasle, N.等人。高通量,基于显微镜的分类解剖细胞异质性。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba16gydF4y2Ba, e9442(2020)。gydF4y2Ba
你,L.等等。功能性单细胞选择和动态变化癌细胞的注释分析。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/2021.10.12.464054gydF4y2Ba(2021)。gydF4y2Ba
Medaglia, C.等人。结合光激活报告子和scRNA-seq的免疫壁龛空间重建gydF4y2Ba科学gydF4y2Ba358gydF4y2Ba, 1622-1626(2017)。gydF4y2Ba
洛瓦特,D.等。活组织中空间定义的单细胞的体内转录组分析。gydF4y2BaNat方法。gydF4y2Ba11gydF4y2Ba, 190-196(2014)。gydF4y2Ba
Asp, M., Bergenstråhle, J. & Lundeberg, J.空间分辨转录组-组织探索的下一代工具。gydF4y2BaBioessaysgydF4y2Ba42gydF4y2Ba, e1900221(2020)。gydF4y2Ba
Moses, L. & Pachter, L.空间转录组学博物馆。gydF4y2BaNat方法。gydF4y2Ba19gydF4y2Ba, 534-546(2022)。gydF4y2Ba
罗德里克斯,S. G.等。Slide-seq:一种在高空间分辨率下测量全基因组表达的可扩展技术。gydF4y2Ba科学gydF4y2Ba363gydF4y2Ba, 1463-1467(2019)。gydF4y2Ba
斯蒂克尔斯,r.r.等人。Slide-seqV2在近细胞分辨率下高度敏感的空间转录组学。gydF4y2Ba生物科技Nat。》。gydF4y2Ba39gydF4y2Ba, 313-319(2021)。gydF4y2Ba
Vickovic, S.等人。用于原位组织分析的高清空间转录组学。gydF4y2BaNat方法。gydF4y2Ba16gydF4y2Ba, 987-990(2019)。gydF4y2Ba
曹,c。et al。使用Seq-Scope对空间转录组进行显微镜检查。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 3559 - 3572。e22(2021)。gydF4y2Ba
Chen, A.等。使用DNA纳米球模式阵列的小鼠器官发生的时空转录组图谱。gydF4y2Ba细胞gydF4y2Ba185gydF4y2Ba, 1777 - 1792。e21(2022)。gydF4y2Ba
Srivatsan, S. R.等。胚胎尺度,单细胞空间转录组学。gydF4y2Ba科学gydF4y2Ba373gydF4y2Ba, 111-117(2021)。gydF4y2Ba
刘,Y.等。在组织中通过确定性条形码进行高空间分辨率的多组学测序。gydF4y2Ba细胞gydF4y2Ba183gydF4y2Ba, 1665 - 1681。e18(2020)。gydF4y2Ba
Eng,学术界。L.等。RNA seqFISH在组织中的转录组尺度超分辨成像。gydF4y2Ba自然gydF4y2Ba568gydF4y2Ba, 235-239(2019)。gydF4y2Ba
莫菲特,J. R.等。高通量单细胞基因表达谱与多重误差稳健荧光原位杂交。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, 11046-11051(2016)。gydF4y2Ba
Gyllborg等人。基于杂交的原位测序(HybISS)用于人类和小鼠脑组织的空间分辨转录组。gydF4y2Ba核酸测定。gydF4y2Ba48gydF4y2Ba, e112(2020)。gydF4y2Ba
李,J. H.等。高度多重亚细胞RNA原位测序。gydF4y2Ba科学gydF4y2Ba343gydF4y2Ba, 1360-1363(2014)。gydF4y2Ba
Ke, R.等。保存组织和细胞中RNA分析的原位测序。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 857-860(2013)。gydF4y2Ba
佩恩,a.c.等人。原位基因组测序可以在完整的生物样本中解析DNA序列和结构。gydF4y2Ba科学gydF4y2Ba371gydF4y2Ba, eaay3446(2021)。gydF4y2Ba
梅里特,C. R.等。固定组织中蛋白质和RNA的多重数字空间分析。gydF4y2Ba生物科技Nat。》。gydF4y2Ba38gydF4y2Ba, 586-599(2020)。gydF4y2Ba
简,M.-P。,Werley, C. A., Farhi, S. L. & Cohen, A. E. Photostick: a method for selective isolation of target cells from culture.化学。科学。gydF4y2Ba6gydF4y2Ba, 1701-1705(2015)。gydF4y2Ba
尼尔马尔,A. J.等。原发性黑色素瘤在单细胞分辨率下的进展和免疫编辑的空间格局。gydF4y2Ba癌症。gydF4y2Ba12gydF4y2Ba, 1518-1541(2022)。gydF4y2Ba
奇维塔等人。激光捕获显微解剖和RNA-seq分析:高灵敏度方法解释人类胶质母细胞瘤FFPE存档组织的组织病理学异质性。gydF4y2Ba前面。肿瘤防治杂志。gydF4y2Ba9gydF4y2Ba, 482(2019)。gydF4y2Ba
本田,等人。光隔离化学高深度空间转录组分析。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 4416(2021)。gydF4y2Ba
胡,K. H.等。ZipSeq:用于实时绘制单细胞转录组的条形码。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 833-843(2020)。gydF4y2Ba
刘宁,戴敏,Saka, S. K.,殷平。超分辨率标签用Action-PAINT。gydF4y2BaNat,化学。gydF4y2Ba11gydF4y2Ba, 1001-1008(2019)。gydF4y2Ba
中村,S., Kawabata, H. & Fujimoto, K.用3-cyanovinylcarbazole进行超快光交联诱导DNA的双双侵袭。gydF4y2Ba化学。Commun。gydF4y2Ba53gydF4y2Ba, 7616-7619(2017)。gydF4y2Ba
金世辉,刘永耀,何泽尔,张晓霞,李廷辉。DNA超分辨率光刻技术。gydF4y2BaNano。gydF4y2Ba19gydF4y2Ba, 6035-6042(2019)。gydF4y2Ba
Vieregg, J. R. Nelson, H. M. Stoltz, B. M. & Pierce, N. A.用屏蔽共价探针选择性核酸捕获。gydF4y2Baj。化学。Soc。gydF4y2Ba135gydF4y2Ba, 9691-9699(2013)。gydF4y2Ba
辛格-加森等人。使用数字微镜阵列光定向寡核苷酸微阵列的无掩模制造。gydF4y2Ba生物科技Nat。》。gydF4y2Ba17gydF4y2Ba, 974-978(1999)。gydF4y2Ba
陈建平,陈建平,陈建平,陈建平,陈建平。单链DNA可编程自动合成方法的研究。gydF4y2BaNat,化学。gydF4y2Ba10gydF4y2Ba, 155-164(2018)。gydF4y2Ba
李,C. S.,戴维斯,R. W. &戴维森,N.用电子显微镜对末端重复的、环状排列的DNA进行物理研究gydF4y2Ba大肠杆菌gydF4y2Ba15.gydF4y2BaJ. Mol.生物学。gydF4y2Ba48gydF4y2Ba, 1-22(1970)。gydF4y2Ba
Yurke, B., Turberfield, A. J. Mills, A. P. Jr . Simmel, F. C. & Neumann, J. L. DNA燃料分子机器。gydF4y2Ba自然gydF4y2Ba406gydF4y2Ba, 605-608(2000)。gydF4y2Ba
潘玉廷,李志刚,谢晓平。DNA分支自发迁移的动力学研究。gydF4y2Ba国家科学院学报美国gydF4y2Ba91gydF4y2Ba, 2021-2025(1994)。gydF4y2Ba
Tecott, L. H., Barchas, J. D. & Eberwine, J. H.原位转录:在固定组织切片中特异性合成互补DNA。gydF4y2Ba科学gydF4y2Ba240gydF4y2Ba, 1661-1664(1988)。gydF4y2Ba
盛凯,曹玮,牛玉玉,邓琪,宗昌。基于MATQ-seq的单细胞转录组变异检测。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 267-270(2017)。gydF4y2Ba
张国强,张国强,张国强。逆转录酶模板转换与假替代转录本。gydF4y2Ba基因组学gydF4y2Ba88gydF4y2Ba, 127-131(2006)。gydF4y2Ba
朱艳燕,马立德,陈奇克,李锐,李培德。逆转录酶模板切换:一种构建全长cDNA文库的SMART方法。gydF4y2Ba生物学技术gydF4y2Ba30.gydF4y2Ba, 892-897(2001)。gydF4y2Ba
沃尔夫,M. G.等。基于Moloney小鼠白血病病毒(MMLV)的逆转录酶的非模板添加和模板切换同时发生并相互竞争。gydF4y2Ba生物。化学。gydF4y2Ba294gydF4y2Ba, 18220-18231(2019)。gydF4y2Ba
道林,j.e.。gydF4y2Ba视网膜:大脑可接近的部分,修订版gydF4y2Ba(哈佛大学出版社,2012)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
谢卡尔,K.等。视网膜双极神经元的单细胞转录组综合分类。gydF4y2Ba细胞gydF4y2Ba166gydF4y2Ba, 1308 - 1323。e30(2016)。gydF4y2Ba
丹宁格,J. K.等。用低输入RNA测序对成年海马神经干和祖细胞群体的差异表达基因进行可靠的转录谱分析和鉴定。gydF4y2Ba前面。摩尔。>。gydF4y2Ba15gydF4y2Ba, 810722(2022)。gydF4y2Ba
Raj, A., van den Bogaard, P., Rifkin, s.a ., van Oudenaarden, A. & Tyagi, S.使用多个单标记探针对单个mRNA分子进行成像。gydF4y2BaNat方法。gydF4y2Ba5gydF4y2Ba, 877-879(2008)。gydF4y2Ba
Kishi, J. Y.等。SABER扩增FISH:增强细胞和组织中RNA和DNA的多重成像。gydF4y2BaNat方法。gydF4y2Ba16gydF4y2Ba, 533-544(2019)。gydF4y2Ba
韦斯特,E. R.等。神经元亚型发生的时空模式提示视网膜多样性的分层发展。gydF4y2Ba细胞的代表。gydF4y2Ba38gydF4y2Ba, 110191(2022)。gydF4y2Ba
李,S.等。覆盖所有的基础:结合来自RNA-seq数据的内含子信号。gydF4y2BaNAR染色体组。Bioinform。gydF4y2Ba2gydF4y2Ba, lqaa073(2020)。gydF4y2Ba
Gustincich, S., Feigenspan, A., Wu, d.k ., Koopman, L. J. & Raviola, E.视网膜中多巴胺释放的控制:神经网络的转基因方法。gydF4y2Ba神经元gydF4y2Ba18gydF4y2Ba, 723-736(1997)。gydF4y2Ba
严,W.等。小鼠视网膜细胞图谱:超过60种无分泌细胞类型的分子鉴定。gydF4y2Baj . >。gydF4y2Ba40gydF4y2Ba, 5177-5195(2020)。gydF4y2Ba
Gustincich, S.等人。基因发现的基因标记单多巴胺能神经元的视网膜。gydF4y2Ba国家科学院学报美国gydF4y2Ba101gydF4y2Ba, 5069-5074(2004)。gydF4y2Ba
Puopolo, M. Hochstetler, S. E. Gustincich, S. Wightman, R. M. & Raviola, E.视网膜神经元突触外释放多巴胺:活动依赖和递质调制。gydF4y2Ba神经元gydF4y2Ba30.gydF4y2Ba, 211-225(2001)。gydF4y2Ba
威特科夫斯基,P.等人。大鼠视网膜多巴胺能神经元中酪氨酸羟化酶的活性依赖磷酸化。gydF4y2Baj . >。gydF4y2Ba24gydF4y2Ba, 4242-4249(2004)。gydF4y2Ba
Greferath, U.等人。GABAA受体亚基在大鼠视网膜中有差异分布:原位杂交和免疫组化。gydF4y2BaJ. Comp. Neurol。gydF4y2Ba353gydF4y2Ba, 553-571(1995)。gydF4y2Ba
Ståhl, P. L.等。空间转录组在组织切片中基因表达的可视化和分析。gydF4y2Ba科学gydF4y2Ba353gydF4y2Ba, 78-82(2016)。gydF4y2Ba
博尔姆,L. E.等。通过电泳捕获mRNA可扩展的原位单细胞分析。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/2022.01.12.476082gydF4y2Ba(2022)。gydF4y2Ba
李国强,李国强。RT原位PCR成功的基础。gydF4y2Ba前面。Biosci。gydF4y2Ba1gydF4y2Ba, c4-c15(1996)。gydF4y2Ba
马歇尔,J. L.等。HyPR-seq:通过DNA探针的杂交和测序对所选rna进行单细胞定量。gydF4y2Ba国家科学院学报美国gydF4y2Ba117gydF4y2Ba, 33404-33413(2020)。gydF4y2Ba
Ouimet, C. C., Hemmings, H. C. Jr & Greengard, P. ARPP-21,一种富含多巴胺神经支配的脑区域的环amp调节磷蛋白。2大鼠脑免疫细胞化学定位。gydF4y2Baj . >。gydF4y2Ba9gydF4y2Ba, 865-875(1989)。gydF4y2Ba
Quinn, J. P, Kandigian, S. E, Trombetta, B. A. Arnold, S. E. & Carlyle, B. C. VGF作为神经退行性和精神疾病的生物标志物和治疗靶点。gydF4y2Ba大脑Commun。gydF4y2Ba3 gydF4y2Ba, fcab261(2021)。gydF4y2Ba
蒋峰,吴庆强,孙松,毕刚,郭磊。帕金森病潜在诊断生物标志物的鉴定。gydF4y2BaFEBS Open BiogydF4y2Ba9gydF4y2Ba, 1460-1468(2019)。gydF4y2Ba
戈尔齐奥,C.等人。马修-伍德综合征是由视黄醇结合蛋白受体基因的截断突变引起的gydF4y2BaSTRA6gydF4y2Ba.gydF4y2Ba点。j .的嗡嗡声。麝猫。gydF4y2Ba80gydF4y2Ba, 1179-1187(2007)。gydF4y2Ba
帕苏托,F.等人。突变gydF4y2BaSTRA6gydF4y2Ba引起广泛的畸形包括无眼症、先天性心脏缺陷、膈疝、肺泡毛细血管发育不良、肺发育不全和智力迟钝。gydF4y2Ba点。j .的嗡嗡声。麝猫。gydF4y2Ba80gydF4y2Ba, 550-560(2007)。gydF4y2Ba
盖伦斯,m.e.等人。人心脏河豚毒素不敏感电压依赖钠通道的基本结构和功能表达。gydF4y2Ba国家科学院学报美国gydF4y2Ba89gydF4y2Ba, 554-558(1992)。gydF4y2Ba
韦尔曼,王尔德,a.a.m.和罗德,e.m.心脏钠通道基因gydF4y2BaSCN5AgydF4y2Ba及其基因产物NaV1.5:在生理和病理生理中的作用。gydF4y2Ba基因gydF4y2Ba573gydF4y2Ba, 177-187(2015)。gydF4y2Ba
法扎尔,F. M.等。APEX-Seq揭示亚细胞RNA定位图谱。gydF4y2Ba细胞gydF4y2Ba178gydF4y2Ba, 473 - 490。e26(2019).
Saka, S. K.等。免疫- saber能够在组织中实现高度复用和放大的蛋白质成像。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba, 1080-1090(2019)。gydF4y2Ba
林,J.-R。et al。使用t-CyCIF和传统光学显微镜对人体组织和肿瘤进行高度多重免疫荧光成像。gydF4y2BaeLifegydF4y2Ba7gydF4y2Ba, e31657(2018)。gydF4y2Ba
Schürch, C. M.等。协调的细胞社区在结直肠癌侵袭前沿协调抗肿瘤免疫。gydF4y2Ba细胞gydF4y2Ba182gydF4y2Ba, 1341 - 1359。e19(2020)。gydF4y2Ba
扎德,J. N.等。NUPACK:分析和设计核酸系统。gydF4y2Baj .第一版。化学。gydF4y2Ba32gydF4y2Ba, 170-173(2011)。gydF4y2Ba
阿特舒,S. F.,吉什,W.,米勒,W.,迈尔斯,E. W. &李普曼,D. J.基本的局部对齐搜索工具。gydF4y2BaJ. Mol.生物学。gydF4y2Ba215gydF4y2Ba, 403-410(1990)。gydF4y2Ba
Kent, W. J. blat -类似blast的对齐工具。gydF4y2Ba基因组Res。gydF4y2Ba12gydF4y2Ba, 656-664(2002)。gydF4y2Ba
Schindelin, J.等。斐济:生物图像分析的开源平台。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 676-682(2012)。gydF4y2Ba
赫什伯格,e.a.等人。PaintSHOP能够交互设计转录组和基因组规模的寡核苷酸FISH实验。gydF4y2BaNat方法。gydF4y2Ba18gydF4y2Ba, 937-944(2021)。gydF4y2Ba
Muhlich, J. L.等。使用ASHLAR拼接和注册高度多路的组织和肿瘤的整个幻灯片图像。gydF4y2Ba生物信息学gydF4y2Ba(2022)gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btac544gydF4y2Ba
Burel,人类。et al。利用OMERO发布和共享多维图像数据。gydF4y2BaMamm。基因组gydF4y2Ba26gydF4y2Ba, 441-447(2015)。gydF4y2Ba
Smith, T., Heger, A. & Sudbery, I. umi -工具:在唯一分子标识符中建模测序误差以提高量化精度。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 491-499(2017)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 923-930(2014)。gydF4y2Ba
赵敏,李,w - p。,Garrison, E. P. & Marth, G. T. SSW library: an SIMD Smith-Waterman C/C++ library for use in genomic applications.《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e82138(2013)。gydF4y2Ba
Amamoto, R.等。Probe-Seq能够通过基于rna的分离对来自异质组织的特定细胞类型进行转录分析。gydF4y2BaeLifegydF4y2Ba8gydF4y2Ba, e51452(2019)。gydF4y2Ba
田昌杰,史崔托,E. &马斯兰德,r.h.小鼠视网膜的主要细胞群。gydF4y2Baj . >。gydF4y2Ba18gydF4y2Ba, 8936-8946(1998)。gydF4y2Ba
王璐,王生,李伟。RSeQC: RNA-seq实验的质量控制。gydF4y2Ba生物信息学gydF4y2Ba28gydF4y2Ba, 2184-2185(2012)。gydF4y2Ba
Phipson, B., Zappia, L. & Oshlack, A.单细胞RNA测序方案中的基因长度和检测偏倚。gydF4y2BaF1000Res。gydF4y2Ba6gydF4y2Ba, 595(2017)。gydF4y2Ba
Karolchik, D, Hinrichs, A. S. & Kent, W. J. UCSC基因组浏览器。gydF4y2Ba咕咕叫。Protoc。嗡嗡声。麝猫。gydF4y2Ba第十八章gydF4y2Ba, Unit18.6(2011)。gydF4y2Ba
Thorvaldsdóttir, H., Robinson, J. T. & Mesirov, J. P.整合基因组学查看器(IGV):高性能基因组学数据可视化和探索。gydF4y2Ba简短。Bioinform。gydF4y2Ba14gydF4y2Ba, 178-192(2013)。gydF4y2Ba
确认gydF4y2Ba
作者感谢Wyss研究所的资助(通过他们的j.y.k.、n.l.、P.Y.和S.K.S.验证项目计划;分子机器人计划;美国国立卫生研究院(授权号UG3HL145600, UH3CA255133, DP1GM133052, R01GM124401, RF1MH124606和RF1MH128861给P.Y.),海军研究办公室(授权号为J.Y.K.)。N00014-18-1-2549至P.Y.),陈·扎克伯格倡议(授权号为。2019-02433 to P.Y.),欧洲分子生物学实验室(S.K.S.)和霍华德·休斯医学研究所(e.r.w., C.L.C.)。作者表彰了哈佛大学的MicRoN Core和P. Montero Llopis的成像设备和专业知识。我们感谢Kylie猫作为掩模生成的猫模型。我们感谢I. Goldaracena在细胞实验方面的帮助,感谢J. Immen在图形设计方面的帮助,感谢T. Ferrante, J. Rosenberg, J. McDonough和B. Bedell的讨论和反馈。若干图形面板(图。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba而且gydF4y2Ba3 cgydF4y2Ba)部分是由BioRender.com制作的。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
J.Y.K.构思并领导了这项研究,设计并执行了实验,编写了软件,分析了数据,并撰写了手稿。n.l构思了这项研究,设计并进行了实验,编写了软件,分析了数据,并撰写了手稿。E.R.W.设计并执行实验,编写软件,分析数据并撰写手稿。K.S.对实验设计、方法优化和数据分析做出了贡献。j.j.j进行了实验,并对方法的优化和手稿的撰写做出了贡献。M.S.提供了实验帮助。C.L.C.贡献了专业知识和监督、实验设计和数据分析。S.K.S.构思了这项研究,提供了科学和技术指导,设计并进行了实验,分析了结果并撰写了手稿。P.Y.构思并监督了这项研究,设计了实验并撰写了手稿。所有作者编辑并批准了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
j.y.k., n.l., P.Y.和S.K.S.是该方法专利申请的发明人。多位作者参与了这项技术的商业化,并与数字生物学公司合作(J.Y.K.和E.R.W.是联合创始人和员工;P.Y.是联合创始人、股东、董事和顾问;S.K.S.预计将成为咨询科学领域的联合创始人;n.l是一位咨询创始科学家;J.J.J.是一名雇员。)他也是Ultivue, Inc.的联合创始人、股东、董事和顾问。其余作者声明没有竞争利益。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然方法gydF4y2Ba感谢刘杨和其他匿名审稿人对这项工作的同行评审所做的贡献。主要处理编辑:Rita Strack,与gydF4y2Ba自然方法gydF4y2Ba团队。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展数据gydF4y2Ba
图1高分辨率光定向DNA条形码。gydF4y2Ba
(一)gydF4y2Ba点阵的荧光图像打印在玻璃载玻片与DNA对接序列功能化。通过荧光DNA条形码链的定向光交联将点打印到互补的对接位点(另见图)。gydF4y2Ba2gydF4y2Ba).选择5个点进行轮廓扫描,灰度值(洋红色虚线),像素对比度设置为450-800。gydF4y2Ba(b)gydF4y2Ba来自面板的线扫描gydF4y2Ba一个gydF4y2Ba(点状的彩色线)被平均成单行扫描(黑点加破折号)。平均线性扫描拟合为高斯曲线(蓝色)。单个点对应一个激活的DMD反射镜,估计可照亮0.76微米直径的区域。拟合的FWHM为~1.56µm。gydF4y2Ba(c)gydF4y2Ba405nm激光共聚焦显微镜下3T3细胞亚细胞标记(gydF4y2BangydF4y2Ba= 4单元格从一个单一的视野)。用于交联的掩模被缩放到荧光图像的大小,并手动覆盖(洋红色)以帮助可视化。在洋红色虚线之间的矩形区域上进行轮廓扫描。gydF4y2Ba(d)gydF4y2Ba面板上虚线框的强度剖面gydF4y2BacgydF4y2Ba,数据拟合为高斯曲线(绿色),测得FWHM为4.4µm。第二次指数衰减适用于剖面扫描的一半(蓝色),以计算84 - 16%的准则,信号从最大值的84下降到16%的距离。从指数拟合计算84-16%下降的距离为2.67µm。垂直虚线表示来自掩模的估计ROI边界。掩模的宽度估计为4微米。gydF4y2Ba(eg)gydF4y2Ba面板中的单一视场gydF4y2BacgydF4y2Ba在共聚焦扫描显微镜上成像。核信号(青色)与ROI选择(白线)重叠gydF4y2Ba(e)gydF4y2Ba,荧光Cy3条形码后严格清洗非交联链gydF4y2Ba(f)gydF4y2Ba,以低对比度显示核信号,使重叠的Cy3信号可视化gydF4y2Ba(g)gydF4y2Ba.gydF4y2Ba
图2 3T3和HEK细胞的细胞分割和读取计数。gydF4y2Ba
(一)gydF4y2Ba代表性分割结果来自小鼠(上一行)和人(下一行)条形码细胞的荧光信号(代表来自gydF4y2BangydF4y2Ba= 3个技术重复)。掩模用于计算条形码区域(另见gydF4y2Ba方法gydF4y2Ba及补充表gydF4y2Ba1gydF4y2Ba).gydF4y2Ba(b)gydF4y2Ba来自细胞混合实验的单个~2亿次读取数据集(品红)被映射到合并的基因组,并按不替换的读取部分进行次采样,并用UMI重复数据删除管道进行处理。来自5个模拟数据集的UMIs的平均数量显示(青色)。gydF4y2Ba(c - d)gydF4y2Ba归一化表达水平(loggydF4y2Ba2gydF4y2Ba(TPM + 1))。我们只考虑了所有重复中检测到的基因(loggydF4y2Ba2gydF4y2Ba(TPM + 1)截止值≥1)。突出显示的数据点(橙色)表示前200个基因,其余基因为蓝色。Pearson相关性为所有基因(黑色)和前200个基因(橙色)报道gydF4y2Ba(c)gydF4y2Ba人类细胞和gydF4y2Ba(d)gydF4y2Ba老鼠的细胞。对数直方图gydF4y2Ba2gydF4y2Ba每个重复的(TPM + 1)分布(不包括前200个基因)绘制在对角线上。完整的基因映射和计数列表如源数据表1所示。gydF4y2Ba
图3视网膜层条形码。gydF4y2Ba
(一)gydF4y2Ba布莱特菲尔德的小鼠视网膜冷冻切片图像,条形码区域覆盖。从左至右分别为外核层(ONL)、双极细胞层(BCL)、神经节细胞层(GCL)。比例尺为100 μ m。二值图像显示了所选的用于条形码掩模的roi。像素尺寸为1.6 μ m/像素。gydF4y2Ba(b)gydF4y2BaDAPI与荧光条形码1-3(分别用Cy5、Cy3和Fluorescein标记)条形码交联后的单z面旋盘共焦图像。gydF4y2Ba(c)gydF4y2Ba在恢复条形码cdna后,同一条形码细胞中PAX6和VSX2蛋白的DAPI、WGA和免疫荧光的单个z面图像用于测序。图片gydF4y2Ba(b)gydF4y2Ba而且gydF4y2Ba(c)gydF4y2Ba自动缩放显示(最小值设置为零)。比例尺为50微米。gydF4y2Ba(d)gydF4y2Ba每个条形码层内单z面旋盘条码荧光(白色)叠加DAPI(蓝色)共聚焦图像,显示不同细胞类型组成的每个细胞层在细胞大小、细胞质面积、RNA密度等方面的不同细胞形态。比例尺为10微米。每单位面积(10 μ m x 10 μ m)的UMI计数为每个条形码层列出。面板gydF4y2Ba模拟gydF4y2Ba是来自n = 4个技术重复的代表性图像。gydF4y2Ba
图4视网膜层实验的Light-Seq测序指标和灵敏度。gydF4y2Ba
(一)gydF4y2Ba序列处理管道。gydF4y2Ba(b)gydF4y2Ba太阳爆发图描述了在每个处理步骤中过滤的读取片段。gydF4y2Ba(c)gydF4y2BaLight-Seq重复的PCA图(每层n = 4个技术重复)。gydF4y2Ba(d)gydF4y2BaLight-Seq和Drop-Seq数据中ONL富集基因与BCL富集基因的相关矩阵gydF4y2Ba46gydF4y2Ba(pgydF4y2Ba邻接的gydF4y2Ba< 0.05,为双侧Wald检验,多重假设检验采用Benjamini-Hochberg调整)。gydF4y2Ba(e)gydF4y2Ba基于减法富集ONL和BCL之间每个细胞Light-Seq转录本的近似差异,以模拟ONL和BCL之间每个细胞Drop-Seq转录本的差异。在两种测定中,ONL或BCL中显著富集的基因被绘制出来(pgydF4y2Ba邻接的gydF4y2Ba< 0.05,为双侧Wald检验,多重假设检验采用Benjamini-Hochberg调整)。放大右边所示的图。gydF4y2Ba(f)gydF4y2Ba在Drop-Seq(模拟BCL)和Light-Seq BCL数据(条形码2)中,估计每个细胞在BCL中富集的基因的读数(pgydF4y2Ba邻接的gydF4y2Ba< 0.05)。gydF4y2Ba(g)gydF4y2Ba平均Drop-Seq的箱线图(gydF4y2BangydF4y2Ba= 6个样本重复)vs Light-Seq (gydF4y2BangydF4y2Ba= 4个片段重复)不同转录本长度的每个基因计数。Pearson R和中位数比率所示。中位数线和四分位数结合在盒子上,用胡须标记1.5×四分位数间距。gydF4y2Ba(h)gydF4y2Ba基于已发表的单细胞smFISH数据的16个BCL标记基因的Light-Seq相对于smFISH的敏感性gydF4y2Ba50gydF4y2Ba.基于BCL中的细胞数量,灵敏度计算为[# smFISH预期转录本]/[#观察到的Light-Seq reads]。点表示单个复制的灵敏度(gydF4y2BangydF4y2Ba= 4个重复)。误差条表示以平均值为中心的标准偏差。gydF4y2Ba(我)gydF4y2BaDrop-Seq相对于smFISH的敏感性。基于Shekhar等人,2016年混合双极簇中的细胞数量gydF4y2Ba46gydF4y2Ba,灵敏度计算为[smFISH预期转录本的数量]/[观察到的Drop-Seq读数](见gydF4y2Ba方法gydF4y2Ba).点反映单个复制/基因的敏感性(gydF4y2BangydF4y2Ba= 6个样本重复)。误差条反映了以平均值为中心的标准偏差。gydF4y2Ba(j)gydF4y2Ba每个基因的平均Light-Seq和Drop-Seq灵敏度之间的差异gydF4y2Ba(h)gydF4y2Ba而且gydF4y2Ba(我)gydF4y2Ba.误差条表示平均数之差的标准误差。为面板gydF4y2BahgydF4y2Ba-gydF4y2BajgydF4y2Ba,基因按基因长度递增排列和着色。gydF4y2Ba
扩展数据图5视网膜层的基因体覆盖和基因长度读取分布。gydF4y2Ba
(一)gydF4y2Ba转录本100元及以上(上)、1000元及以上(中)、10000元及以上(下)。gydF4y2Ba(c)gydF4y2Ba每千碱基转录读数,每百万映射读数(RPKM)(面板gydF4y2BabgydF4y2Ba)和读取计数(面板gydF4y2BacgydF4y2Ba)用于不同转录本长度的箱子(箱子基于Phipson等人,2017年)的所有条形码复制条件gydF4y2Ba87gydF4y2Ba).所有箱形图均以中位数线和四分位数为边框,并以胡须标记1.5×四分位数间距。gydF4y2BangydF4y2Ba= 4个技术重复。gydF4y2Ba
扩展数据图6 THgydF4y2Ba+gydF4y2Ba交流ROI选择和信号。gydF4y2Ba
(一)gydF4y2BaTH的第一个IFgydF4y2Ba+gydF4y2BaAC在40×物镜共聚焦显微镜上成像,条形码ROI覆盖(洋红色,左)。条形码区域的荧光图像(右)。像素值设置为0-400。gydF4y2Ba(b)gydF4y2Ba对荧光条形码信号上的50 × 5µm矩形(面板中的虚线框)进行轮廓扫描gydF4y2Ba一个gydF4y2Ba).虚线表示ROI边界。gydF4y2Ba(c)gydF4y2Ba选择单张TH图像gydF4y2Ba+gydF4y2BaIF染色的无分泌细胞(上),roi覆盖(洋红色,下)。ROI被稍微画在胞体内部,以解释在ROI边界处的光散射。比例尺为10微米。面板gydF4y2Ba(一)gydF4y2Ba而且gydF4y2Ba(c)gydF4y2Ba是来自gydF4y2BangydF4y2Ba= 5个技术重复。gydF4y2Ba(d)gydF4y2Ba从一个具有代表性的实验条件中选择一个370万读深的单次测序运行深度进行子采样,不进行替换,说明UMI按基因读的分数缩放gydF4y2BaThgydF4y2Ba,gydF4y2BaCartptgydF4y2Ba,以及UMIs总数。绘制了5个模拟(青色)的均值+ /-标准差,完整的数据集表示为单个点(洋红色)。gydF4y2Ba
扩展数据图7无分泌细胞实验的基因体覆盖和基因长度读取分布。gydF4y2Ba
(一个gydF4y2Ba)转录本100元及以上(左)、1000元及以上(中)、10000元及以上(右)的基因体覆盖率。gydF4y2Ba(c)gydF4y2Ba每千碱基转录数,每百万映射读数(RPKM) (ingydF4y2BabgydF4y2Ba)和读计数(ingydF4y2BacgydF4y2Ba)用于不同转录本长度的所有条形码复制条件。所有箱形图均以中位数线和四分位数为边框,并以胡须标记1.5×四分位数间距。gydF4y2BangydF4y2Ba= 5个技术重复。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
“补充资料”包含“补充图1-3”、“表1-6”、“附注1-4”及参考文献。gydF4y2Ba
补充表7gydF4y2Ba
用于RNA-FISH验证实验的SABER-FISH探针组列表。gydF4y2Ba
源数据gydF4y2Ba
图4 .来源数据gydF4y2Ba
视网膜组织实验中各层间差异富集基因的完整列表。gydF4y2Ba
图5 .来源数据gydF4y2Ba
在TH之间富集的差异富集基因的完整列表gydF4y2Ba+gydF4y2Ba和THgydF4y2Ba−gydF4y2Ba罕见的视网膜无分泌细胞组织实验。gydF4y2Ba
图2 .扩展数据gydF4y2Ba
人鼠细胞混合实验的基因映射和计数复制。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Kishi, j.y.,刘宁,West, E.R.gydF4y2Baet al。gydF4y2BaLight-Seq:固定细胞和组织中生物分子的光定向原位条形码,用于空间索引测序。gydF4y2BaNat方法gydF4y2Ba(2022)。https://doi.org/10.1038/s41592-022-01604-1gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41592-022-01604-1gydF4y2Ba