摘要
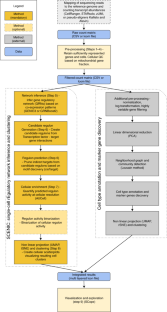
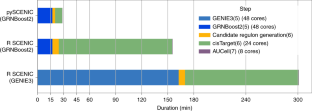
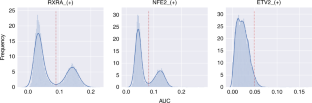
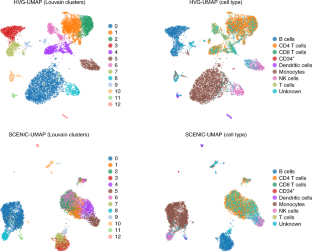
该协议解释了如何使用软件容器和Nextflow管道对单细胞rna测序数据执行快速SCENIC分析以及标准最佳实践步骤。SCENIC重建规则(即转录因子及其靶基因)评估这些发现的规则在单个细胞中的活性,并使用这些细胞活动模式来寻找有意义的细胞集群。在这里,我们提出了一个改进的版本的风景区有几个进步。在Python中重构和重新实现了SCENIC (pySCENIC),从而使速度提高了十倍,并已打包到容器中以方便使用。现在也有可能使用表观基因组跟踪数据库,以及基序,以完善规则。在这个方案中,我们解释了SCENIC的不同步骤:工作流程从描述所有细胞基因丰度的计数矩阵开始,由三个阶段组成。首先,使用逐目标回归方法(GRNBoost2)推断共表达式模块。接下来,使用顺式调控motif发现(cisTarget)从这些模块中修剪间接目标。最后,这些调控子的活性通过调控子靶基因(AUCell)的富集评分进行量化。基于这些规则的细胞活动模式,可以使用非线性投影方法来显示细胞的可视分组。 The results can be exported as a loom file and visualized in the SCope web application. This protocol is illustrated on two use cases: a peripheral blood mononuclear cell data set and a panel of single-cell RNA-sequencing cancer experiments. For a data set of 10,000 genes and 50,000 cells, the pipeline runs in <2 h.
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
一般转录因子TAF4通过Polycomb拮抗表观遗传沉默以维持肠道干细胞功能
细胞死亡与分化开放获取2023年1月13日
单细胞转录组学揭示了早期糖尿病肾病的机械敏性损伤信号通路
基因组医学开放获取2023年1月10日
单细胞主导的药物用于治疗阿尔茨海默病
科学报告开放获取2023年1月5日
访问选项
订阅《自然》+
立即在线访问《自然》和其他55种《自然》杂志
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
99.00美元
每期只要8.25美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。







数据可用性
在该协议中分析的所有数据都是公开的。PBMC 10k数据集可直接从10x Genomics公司网站下载:https://support.10xgenomics.com/single-cell-gene-expression/datasets/3.0.0/pbmc_10k_v3.以下数据集可从国家生物技术信息中心的GEO获得,并可通过GEO系列登录号访问:GSE60361(小鼠大脑数据集),GSE115978(人类皮肤黑色素瘤),以及GSE103322(人类HNSC)。非小细胞肺癌的数据集可以从ArrayExpress下载e - mtab - 6149而且e - mtab - 6653).其他元数据可作为来自生成这些数据集的原始出版物的补充信息文件。该协议中使用的案例研究的在线版本可在GitHub (https://github.com/aertslab/SCENICprotocol),包括Jupyter笔记本和Nextflow项目代码,以及相关的安装和使用说明。
代码的可用性
SCENIC作为Python包可在https://pypi.org/project/pyscenic/,其源代码可在GitHub (https://github.com/aertslab/pySCENIC).本手稿中的代码已经过同行评审。
参考文献
Satija, R., Farrell, J. A., Gennert, D., Schier, A. F. & Regev .单细胞基因表达数据的空间重建。生物科技Nat。》。33, 495-502(2015)。
伍尔夫,安杰尔,彭国强,冯杰。SCANPY:大规模单细胞基因表达数据分析。基因组医学杂志。19, 15(2018)。
Aibar, S.等。SCENIC:单细胞调控网络推断和聚类。Nat方法。14, 1083-1086(2017)。
Tommaso, P.等人。Nextflow支持可复制的计算工作流。生物科技Nat。》。35, 316-319(2017)。
Dobin, A.等人。STAR:超快通用RNA-seq对准器。生物信息学29, 15-21(2013)。
Bray, N. L, Pimentel, H., Melsted, P. & Pachter, L.近最优概率RNA-seq量化。生物科技Nat。》。34, 525-527(2016)。
parkh, S., Ziegenhain, C., Vieth, B., Enard, W. & Hellmann, I. zmis -一个快速灵活的管道,用于处理RNA测序数据与UMIs。Gigascience7, 1-9(2018)。
Srivastava, A., Malik, L., Smith, T., Sudbery, I. & Patro, R. Alevin从dscRNA-seq数据中有效地估计了准确的基因丰度。基因组医学杂志。20., 65(2019)。
Ilicic, T.等。从单细胞RNA-seq数据中分类低质量细胞。基因组医学杂志。17, 29(2016)。
Huynh-Thu, V., Irrthum, A., Wehenkel, L. & Geurts, P.基于树的方法从表达数据推断调节网络。《公共科学图书馆•综合》5, e12776(2010)。
莫尔曼,T.等。GRNBoost2和Arboreto:高效和可扩展的基因调控网络推理。生物信息学35, 2159-2161(2018)。
Gaiteri, C., Ding, Y., French, B., Tseng, G. & Sibille, E.超越模块和中心:基因共表达网络在研究复杂大脑疾病分子机制中的潜力。基因,大脑行为。13, 13-24(2014)。
Janky, R. et al. iRegulon:从一个基因列表到一个基因调控网络使用大型motif和轨道集合。公共科学图书馆第一版。医学杂志。10, e1003731(2014)。
Herrmann, C., de Sande, B., Potier, D. & Aerts, S. i-cisTarget:预测调控特征和顺式调控模块的整合基因组学方法。核酸测定。40, 1-44(2012)。
Imrichová, H., Hulselmans, G., Atak, Z., Potier, D. & Aerts, S. i-cisTarget 2015年更新:人类、小鼠和苍蝇的广义顺式调控富集分析。核酸测定。43, w57-w64(2015)。
贝克特,E.等。使用UMAP可视化单单元数据的降维。生物科技Nat。》。9, 26(2018)。
戴维,K.等人。衰老的单细胞转录组图谱果蝇大脑。细胞174, 1-38(2018)。
博蒂埃,D.等人。定位基因调控网络果蝇通过大规模转录组扰动和基序推断的眼睛发育。细胞的代表。9, 2290-2303(2014)。
Svensson, V., Vento-Tormo, R. & Teichmann, S. A.单细胞RNA-seq在过去十年中的指数缩放。Protoc Nat。13, 599-604(2018)。
桑吉尼提,G. & Huynh-Thu, V. A.。基因调控网络:方法和协议(施普林格,2019)。
库尔泽,g.m.,索查特,V. &鲍尔,m.w.奇点:计算移动性的科学容器。《公共科学图书馆•综合》12, e0177459(2017)。
Pratapa, A., Jalihal, A. P., Law, J. N., Bharadwaj, A. & Murali, T.从单细胞转录组数据进行基因调控网络推理的基准算法。Nat方法。17, 147-154(2020)。
兰伯特,s.a.等。人类转录因子。细胞172, 650-665(2018)。
菲尔斯,M. W.等。从单细胞组学数据绘制基因调控网络。简短。功能。基因组学17, 246-254(2018)。
de Smet, R. & Marchal, K.当前网络推理方法的优点和局限性。Nat. Rev.微生物。8, 717-729(2010)。
郭敏,王宏,波特,S. S., Whitsett, J. a . &徐勇。真挚的单细胞rna序列分析管道。公共科学图书馆第一版。医学杂志。11, e1004575(2015)。
Mohammadi, S., Ravindra, V., Gleich, d.f. & Grama, A.描述单细胞功能身份的几何方法。Commun Nat。9, 1516(2018)。
van Dijk, D.等。利用数据扩散从单细胞数据中恢复基因相互作用。细胞174, 716-729(2018)。
Deshpande, A., Chu, l.f。,Stewart, R. & Gitter, A. Network inference with Granger causality ensembles on single-cell transcriptomic data. Preprint athttps://www.biorxiv.org/content/10.1101/534834v1(2019)。
蔡泽尔等人。单细胞rna测序显示小鼠皮层和海马体中的细胞类型。科学347, 1138-1142(2015)。
陈x, Teichmann, S. A. & Meyer, K. B.从组织到细胞类型和返回:组织结构的单细胞基因表达分析。为基础。启生物医学。科学数据。1, 1-23(2018)。
Tirosh, I. & Suvà, M. L.通过单细胞表达谱破译人类肿瘤生物学。为基础。癌症生物学。3., 1-16 (2018)
奥巴迪亚,M.和Bhandoola .先天和适应性淋巴细胞谱系的转录调节。为基础。启Immunol。33, 1-36(2014)。
Laresgoiti, U.等。E2F2和CREB协同调节细胞周期基因的转录活性。核酸测定。41, 10185-10198(2013)。
诺克斯,J. J., Cosma, G. L., Betts, M. R. & McLane, L. M. T-bet和eomes在人免疫细胞外周的特征。前面。Immunol。5, 217(2014)。
林,y.c .等。一个全球转录因子网络,包括E2A, EBF1和Foxo1,调节B细胞的命运。Immunol Nat。11, 635(2010)。
Boller, S. & Grosschedl, R. b细胞分化的调节网络:早期b细胞因子1功能的重点观点。Immunol。牧师。261, 102-115(2014)。
Suo, S.等。揭示小鼠细胞图谱中细胞身份的关键调控因子。细胞的代表。25, 1436-1445(2018)。
香农等人。细胞景观:生物分子相互作用网络集成模型的软件环境。基因组Res。13, 2498-2504(2003)。
Jerby-Arnon, L.等。癌细胞程序促进T细胞排斥和抵抗检查点封锁。细胞175, 984-997(2018)。
Puram, S. V.等。头颈部癌症原发和转移性肿瘤生态系统的单细胞转录组分析。细胞171, 1611-1624(2017)。
Lambrechts, D.等。肺肿瘤微环境中基质细胞的表型成型。Nat,地中海。24, 1277-1289(2018)。
帕夫利迪斯,P. & Noble, W. S.小鼠大脑基因表达的品系和区域变异分析。基因组医学杂志。2,研究0042.1(2001)。
张,B. & Horvath, S.加权基因共表达网络分析的一般框架。Stat .。麝猫。摩尔。4,第17(2005)条。
Frith, m.c., Li, m.c. & Weng, Z. Cluster-Buster:在DNA序列中发现密集的基序簇。核酸测定。31, 3666-3668(2003)。
茨威格,a.s., Karolchik, D.,库恩,R. M.,豪斯勒,D. &肯特,J. W. UCSC基因组浏览器教程。基因组学92, 75-84(2008)。
Aerts, S.等人。通过基因组数据融合确定基因优先级。生物科技Nat。》。24, 537-544(2006)。
人类基因组中DNA元素的综合百科全书。自然489, 57-74(2012)。
维莱拉,A. J.等。集合比较基因树:完整的,重复意识的系统发育树在脊椎动物。基因组Res。19, 327-335(2009)。
古普塔,S., Stamatoyannopoulos, J. A. Bailey, T. L. & Noble, W.量化母题之间的相似性。基因组医学杂志。8, r24(2007)。
萨勃拉曼尼亚,A.等。基因集富集分析:解释全基因组表达谱的一种基于知识的方法。国家科学院学报美国102, 15545-15550(2005)。
Luecken, m.d. & Theis, f.j.单细胞rna序列分析的当前最佳实践:教程。摩尔。系统。医学杂志。15, e8746(2019)。
van der Maaten, L. & Hinton, G.使用t-SNE可视化数据。j·马赫。学习。Res。9, 2579-2605(2008)。
布朗德尔,V. D.,纪尧姆,J.-L。,Lambiotte, R. & Lefebvre, E. Fast unfolding of communities in large networks.J. Stat. Mech。2008, p10008(2008)。
Pliner, H. A., Shendure, J. & Trapnell, C.监督分类能够快速注释细胞图谱。Nat方法。16, 983-986(2019)。
确认
这项工作是由VLAIO (no。HBC.2017.1003至j.r.、y.s.和S. Aerts);由ERC整合商授予(no。724226_cis-CONTROL到S. Aerts);鲁汶大学(KU Leuven)批准。C14/18/092至S. Aerts)。计算工作在弗拉姆斯超级计算机中心进行。资助者在研究设计、数据收集和分析、发表决定或手稿准备中没有任何作用。
作者信息
作者及隶属关系
贡献
概念化:b.v.d.s., c.f., j.r., y.s., S. Aerts;方法:B.V.d.S,由K.D, M.D.W, G.H。s . Aibar R.S, w,阻容,Q.R,电视,D.D.M。j。r廖曜生,和美国Aerts;由软件:B.V.d.S, K.D, M.D.W, G.H。s . Aibar R.S, w,阻容,Q.R,电视,和D.D.M.;验证、资源和数据管理:B.V.d.S.和C.F.;写作-原稿:b.v.d.s., c.f., S. Aerts;写作评论和编辑:b.v.d.s., c.f., S. Aerts;可视化:b.v.d.s., c.f., S. Aerts;监督:S. Aerts。, y.s.和J.R.
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
相关链接
使用此协议的关键引用
戴维,K.等人。细胞174, 982-998 (2018):https://doi.org/10.1016/j.cell.2018.05.057
Lambrechts, D.等。地中海Nat。.24, 1277-1289 (2018):https://doi.org/10.1038/s41591-018-0096-5
Wouters, J.等人。预印在bioRxiv(2019):https://www.biorxiv.org/content/10.1101/715995v2
Aibar, S.等。Nat方法。14, 1083-1086 (2017):https://doi.org/10.1038/nmeth.4463
补充信息
权利和权限
关于本文
引用本文
范德·桑德,B.,弗莱林,C.,戴维,K.。et al。用于单细胞基因调控网络分析的可伸缩的SCENIC工作流程。Nat Protoc15, 2247-2276(2020)。https://doi.org/10.1038/s41596-020-0336-2
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41596-020-0336-2
这篇文章被引用
在单细胞水平上描述肿瘤微环境揭示了骨肉瘤中一种新的免疫逃避机制
骨的研究(2023)
一般转录因子TAF4通过Polycomb拮抗表观遗传沉默以维持肠道干细胞功能
细胞死亡与分化(2023)
单细胞主导的药物用于治疗阿尔茨海默病
科学报告(2023)
单细胞转录组学揭示了早期糖尿病肾病的机械敏性损伤信号通路
基因组医学(2023)
小型冷冻临床标本的多模式单细胞和全基因组测序
自然遗传学(2023)
