摘要
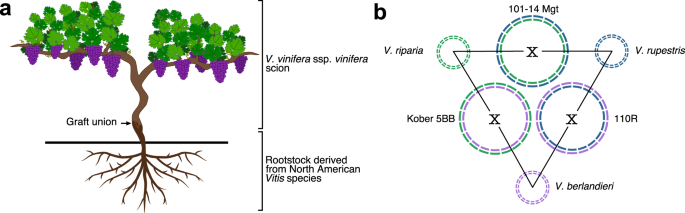
栽培葡萄通常嫁接在近亲上,以应对特定的生物和非生物胁迫条件。三个北美葡萄属物种诉锐利,诉rupestris,诉? ?,是栽培葡萄砧木的主要品种。在这里,我们报道了来自这些物种的三个广泛使用的砧木的二倍体染色体规模组装:Richter 110 (110R), Kober 5BB和101-14 Millardet et de Grasset(管理)。利用PacBio HiFi序列在平均覆盖53.1 x倍的情况下组装了三个杂种的基因组草图。使用HaploSync工具,我们为每个基因组重建了两组19个染色体尺度的假分子,平均单倍体基因组大小为494.5 Mbp。剩余单倍型开关使用共享单倍型信息进行解析。这三个参考基因组为研究葡萄适应生物和非生物胁迫的遗传基础,以及为砧木育种计划设计性状相关标记提供了宝贵的资源。
| 测量(s) | 基因组组装序列 |
| 技术类型(年代) | PacBio续作系统 |
| 样本特征-有机体 | 白葡萄x红葡萄•绿葡萄x绿葡萄•白葡萄x绿葡萄•绿葡萄x绿葡萄 |
| 样本特征-位置 | 加利福尼亚州 |
背景与总结
栽培葡萄(葡萄ssp。酿酒用葡萄)通常嫁接到源自北美的砧木上葡萄属物种(图。1).这种做法是在19世纪建立的,以应对葡萄根蚜虫对欧洲葡萄园的近乎毁灭(Daktulosphaira vitifoliae惠誉)1.葡萄根瘤蚜是在19世纪50年代通过北美的植物材料传入欧洲的2.大部分北美人葡萄属物种对根瘤蚜具有抗性,这可能是它们在原生环境中与这种昆虫共同进化的结果。葡萄属锐利而且葡萄属rupestris第一个野生葡萄品种被用作砧木是因为它们很容易从硬木扦插上生根,并且与产浆果的接穗具有良好的嫁接相容性吗3..然而,这两种植物不适合欧洲常见的钙质土壤。葡萄? ?另一种北美葡萄品种——葡萄根瘤蚜(phylloxera)被发现具有抗根瘤蚜和耐石灰性,尽管它的根很少来自休眠的插枝4.介绍耐石灰性诉? ?并通过杂交培育新砧木,提高其可生根性诉? ?用诉锐利或诉rupestris.今天,商业化的砧木主要是这三个葡萄品种的杂交5.其中,里克特110 (110R;诉? ?x诉rupestris)、高柏5BB (诉? ?x诉锐利)和101-14 Millardet et de Grasset(管理;诉锐利x诉rupestris)是世界上最常用的(图。1 b).除了对根瘤蚜的抗性外,葡萄砧木的选择还基于对生物(如线虫)和非生物胁迫(如干旱)的耐受性、对土壤理化性质的偏好以及它们赋予接穗的活力水平6.例如,101-14 Mgt虽然活力中等,但通常会引发营养生长早熟,而110R和Kober 5BB则具有较高的活力,并延迟植株成熟7.110R以其耐旱性而闻名,过多的土壤水分对其生长有负面影响6.相比之下,101-14 Mgt和Kober 5BB被认为不耐旱,在潮湿的土壤中生长良好6.三种砧木对线虫的耐受性也因线虫种类的不同而不同6,8.
除了商业价值外,砧木对研究葡萄适应生物和非生物胁迫的遗传基础也很有价值9.然而,迄今为止只有两个基因组诉锐利已经出版10,11而且没有任何常用砧木的参考基因组。本文介绍了110R、Kober 5BB和101-14 mgmt的染色体规模组装。使用高度精确的长读测序(HiFi, Pacific Biosciences)对基因组进行测序,并用Hifiasm进行组装12.然后使用HaploSync工具套件将每个二倍体草图基因组构建成两组假分子13,并为每个细胞分配单倍型葡萄属亲本基于来自同一物种的单倍型之间的序列相似性。这些基因组为研究砧木抗环境因子的遗传基础和设计标记以加速砧木育种提供了重要的资源。
方法
文库制备和测序
幼叶(1-2厘米宽)采集自110R (FPS 01)、Kober 5BB (FPS 06)和101-14 Mgt (FPS 01)的基础植物服务(加州大学戴维斯分校,戴维斯,CA),并立即冷冻并在液氮中研磨成粉末。高分子量基因组DNA从1克叶片组织中提取,如Chin所述等.14,使用Megaruptor将12µg高分子量gDNA剪切成15 ~ 20 kbp的大小分布®2 (Diagenode, Denville, NJ, USA)。每次加入时,使用SMRTbell®Express Template Prep Kit 2.0制备一个HiFi测序库,然后立即使用酶清理试剂盒(Pacific Biosciences, Menlo Park, CA, USA)进行处理。使用BluePippin (Sage Sciences, Beverly, MA, USA)和HiFi SMRTbell®模板收集长度超过15 kbp的文库大小选择。使用AMPure PB珠(Pacific Biosciences, Menlo Park, CA, USA)清洗大小选择的文库组分。分别使用Qubit™1X dsDNA HS检测试剂盒(Thermo Fisher, Waltham, MA, USA)和Femto Pulse系统(Agilent, Santa Clara, CA, USA)评估文库的浓度和最终大小分布。110R和Kober 5BB的HiFi文库在加州大学戴维斯分校(Davis, CA, USA)的DNA技术核心设施使用PacBio Sequel II系统(Pacific Biosciences, CA, USA)进行测序。101-14 Mgt的测序由Corteva Agriscience (Johnston, IA, USA)执行,作为Pacific Biosciences授予Noé Cochetel博士的奖励。每个基因组平均生成26.5±3.8 Gbp序列,对应于500 Mbp单倍体基因组的53.1±7.7 x倍覆盖(表2)1).
总RNA诉? ?9031年,诉rupestrisB38,诉锐利HP-1 (PI588271)叶片采用十六烷基三甲基溴化铵(CTAB)为基础的提取方案,如Blanco-Ulate所述等.15.RNA纯度用Nanodrop 2000分光光度计(Thermo Scientific, Hanover Park, IL, USA)进行评估,RNA完整性用电泳和Agilent 2100生物分析仪(Agilent Technologies, CA, USA)进行评估。RNA数量用Qubit 2.0荧光仪和大范围RNA试剂盒进行评估(Life Technologies, Carlsbad, CA, USA)。总RNA (300 ng, RNA完整性编号>8.0)用于文库构建。使用Illumina TruSeq RNA样品制备试剂盒v.2制备短读cDNA文库(Illumina, CA, USA)遵循Illumina™低吞吐量协议。使用高灵敏度芯片和Agilent 2100生物分析仪(Agilent Technologies, CA, USA)对文库的数量和质量进行评估。每个物种一个文库使用Illumina HiSeq 4000测序仪进行测序,测序程序为2x100bp (DNA技术核心设施,加州大学戴维斯分校,美国)。制备长读cDNA SMRTbell文库诉? ?而且诉锐利.第一链合成和cDNA扩增使用NEB Next Single Cell/Low Input cDNA合成和扩增模块(New England, Ipswich, MA, USA)完成。随后使用ProNex磁珠(Promega, WI, USA)按照Iso-Seq快速模板制备Sequel和Sequel II系统协议(Pacific Biosciences, Menlo Park, CA, USA)中的说明对cdna进行纯化。使用ProNex磁珠(86µL)筛选扩增的cDNA(≥2 kbp)。使用至少80 ng的大小选择的扩增cDNA制备cDNA SMRTbell文库。按照制造商的方案,使用SMRTbell Express Template Prep Kit 2.0 (Pacific Biosciences, Menlo Park, CA, USA)进行DNA损伤修复和SMRTbell连接。每个物种的一个SMRT细胞在PacBio Sequel I平台上测序(DNA技术核心设施,加州大学戴维斯分校,CA, USA)。
基因组组装和假分子构建
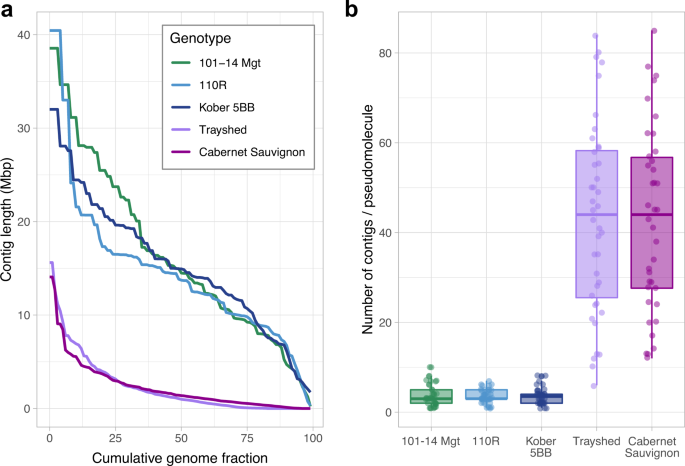
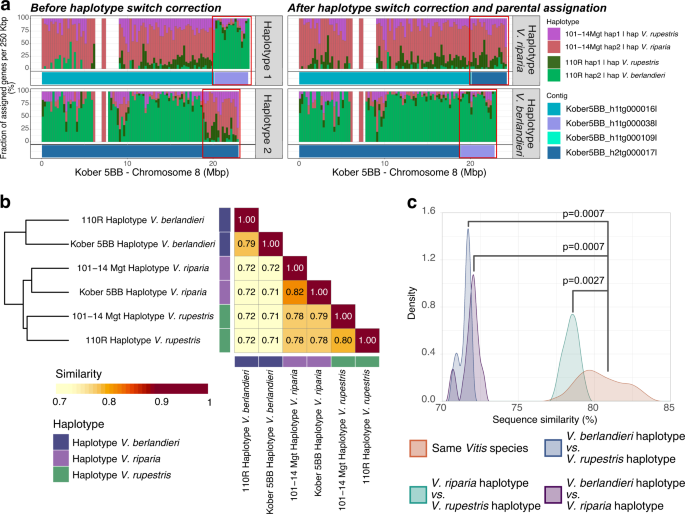
使用Hifiasm v.0.16.1-r374组装HiFi读取12.测试了几种装配参数的多种组合。总共生成了1,939个程序集。选择每个基因型碎片最少的组合。所选的草案组件由406±226个contigs组成,N50 = 14.3±0.6 Mbp(表2)1).与之前用PacBio CLR技术生成的其他葡萄基因组相比,PacBio HiFi读取器大大提高了草案装配的邻近性(PacBio CLR 1.2±0.3 Mbp,图。2).利用BUSCO V.5.1与Viridiplantae和Embryophyta ODB10数据集评估基因空间完整性16以及通过映射PN40024 (V1注释17)单拷贝基因,使用GMAP v.2019-09-12(考虑至少80%覆盖率和80%同源性的比对)。对于每个砧木,草图基因组组装经过质量控制,并使用HaploSync构建成染色体规模的二倍体假分子13和葡萄属邹教授开发的共识遗传图谱等.18.每个基因型使用一个周期的HaploFill。与最近发表的使用PacBio CLR技术测序的葡萄基因组相比,使用PacBio HiFi reads显著减少了草案装配的碎片化(图2)。2 b)13,14,19.较低的碎片化导致支架假分子所需的contigs数量减少了15倍(3.6±2.0 HiFi contigs/假分子)vs.43.0±20.6 CLR contigs/假分子)2 b).值得注意的是,在三个基因组中,从单个contig中重建了15个假分子。根据蛋白质编码序列的序列相似性鉴定单倍型开关。利用minimap2 v.2.17-r941对各砧木的基因位点序列进行比对20.参数" -x map-hifi "。具有最高覆盖率和身份的比对被用于分配常见的种亲缘关系,并沿假分子检测单倍型开关(图2)。3).手动校正单倍型开关后,进行第二周期HaploFill13是用同样的假分子做的吗葡萄属物种作为替代单倍型,以帮助缩小草稿序列的差距。
PacBio HiFi测序技术大大提高了葡萄属基因组组装草图。(一个) 101-14 Mgt、110R、Kober 5BB的初稿装配破碎度表示为contig NG(x)值的分布。Muscadidia rotundifolia简历。Trayshed和诉酿酒用葡萄简历。赤霞珠(Cabernet Sauvignon)采用CLR标准生产,作为比较。NG(x)值定义为当序列从最长到最短排序时,累积达到预期二倍体基因组长度(1 Gbp)的给定分数(x)所必需的最短contig的序列长度。使用PacBio HiFi reads (101-14 Mgt, 110R和Kober 5BB)生产的二倍体组装,与使用旧的长读测序技术组装的其他葡萄基因组相比,尽管采用了较低的X-Fold覆盖率(PacBio Sequel CLR reads为Trayshed 140x - fold覆盖率;19,21PacBio RSII CLR读数为赤霞珠,115X X-Fold覆盖14).(b)组成完整假分子的contig数目的分布。使用PacBio HiFi reads (101-14 mgmt, 110R和Kober 5BB)生成的草案程序集的碎片大大降低,导致构建假分子所需的contigs数量平均减少了15倍。
基于种内序列相似性的单倍分型。共享的亲本物种信息被用来分配每个单倍型诉锐利,诉rupestris,或诉? ?基于序列相似性。这允许解决装配错误(即单倍型开关)。(一个在Kober 5BB的第8号染色体上发现的单倍型开关的例子(左图)。伪分子搭建后,在Kober 5BB的第8染色体末端观察到一个单倍型开关。单倍型1上的contig Kober5BB_h1tg000016l的基因与101-14 Mgt单倍型2(红色)上的基因高度相似,表明Kober5BB_h1tg000016l来源于诉锐利,而Kober5BB_h1tg000038l的基因对应于110R的8号染色体2单倍型基因(浅绿色),表明Kober5BB_h1tg000038l来源于诉? ?.在单倍型2上观察到相反的模式,假分子前18.9 Mbp的基因与110R单倍型2的基因相似(浅绿色),后4.2 Mbp的基因与101-14 Mgt单倍型2的基因相似(红色)。通过将contig Kober5BB_h1tg000038l与替代单倍型中的相应区域互换来纠正单倍型开关,由Kober5BB_h2tg000109l和Kober5BB_h2tg000017l的4.2 Mbp组成(右侧面板)。(b)单倍型之间的序列相似性表示为与另一单倍型相比,不受结构变异(>50 bp)、snp或InDels影响的单倍体染色体集长度的平均百分比。(c)同一种单倍型与不同种单倍型序列相似度百分比(定义见B)的分布(采用两两Wilcoxon秩和检验进行统计检验,以adjust = 1, n = 4096, kernel =“cos”参数绘制密度图)。
基因预测和重复注释
中描述的程序预测基因结构注释https://github.com/andreaminio/AnnotationPipeline-EVM_based-DClab21.对于每个砧木,来自相应亲本种的Iso-Seq数据与新创在生成基因模型之前,从RNA-seq reads中组装转录本。Iso-Seq库使用IsoSeq3 v.3.3.0协议进行提取、解复用和错误校正(https://github.com/PacificBiosciences/IsoSeq).使用LSC v2.0进一步优化低质量和单一异构体数据集22.RNA-seq读取被质量过滤,适配器被Trimmomatic v.0.36和选项“ILLUMINACLIP:2:30:10前导:7拖尾:7滑动窗口:10:20 MINLEN:36”修剪。23.从中读取高质量的RNA-seq葡萄属(i) Trinity v.2.6.524用“新创协议,(ii) Trinity v.2.6.524使用“On-genome”协议,(iii) Stringtie v.1.3.4d25使用HISAT2 v.2.0.5和参数“-非常敏感”对基因组序列进行比对26.然后将三种组装方法共有的转录本序列与Iso-Seq读取结果合并。使用CD-HIT v4.6减少序列冗余27cd-hit-est -c 0.99 -g 0 -r 0 -s 0.70 -aS 0.99非冗余转录本采用PASA v.2.3.3进行处理28得到最终的训练模型集。结合来自公共数据库的数据,使用包括Exonerate v.2.2.0在内的多比对器管道将衍生的转录本和蛋白质证据在基因组组装上进行比对29和Pasa v.2.3.328.使用EvidenceModeler v.1.1.1生成最终一组共识基因模型30.,从头开始预测也使用Augustus v.3.0.3生成31, BUSCO v.3.0.232, GeneMark v.3.4733,以及SNAP v.2006-07-2834.对于重复注释,请使用RepeatMasker v.open-4.0.635是使用。要为每个基因模型分配功能注释,请参阅diamond v2.0.13.151的结果36,37blastp在Refseq植物蛋白数据库中的匹配(https://ftp.ncbi.nlm.nih.gov/refseq/,检索于2019年1月17日)和InterProScan v.5.28-67.038通过Blast2GO v.4.1.939.在110R、Kober 5BB和101-14 Mgt的基因组组装中,共注释了56,768个蛋白编码基因位点,59,807个。平均每个单倍型鉴定出124,991±36,197个蛋白质编码备选剪接变异。未放置序列由2,747±2,821个基因位点组成(表1).
单倍型间共线性分析
共线基因位点通过MCScanX v.11. 2013进行鉴定40.使用GMAP v.2019-09-12对三个砧木的注释蛋白编码序列进行比对41参数为“- b4 -x 30-split-output”。一致性和覆盖率均大于80%的对齐被保留。使用来自Bedtools v2.29.2的mapBed识别与注释的mRNA区域对应的比对42参数为“-F 0.75 -F 0.5 -e”。然后用mcscananx_h (MCScanX v. 11.11.2013)检测共线块40)工具使用以下参数“-s 10 -m 5 -w 5”。
单倍型之间序列多态性和结构变异的鉴定
伪分子序列使用MUMmer4 v.4.0.0.beta5的nucmer工具进行比对43.使用show-snps工具(MUMmer4 v.4.0.0.beta5)对单倍型之间的snp和短索引进行比对43),参数“-Clr -x”和更长的结构变量显示diff工具(MUMmer4 v.4.0.0.beta543),使用默认参数。
数据记录
测序数据保存在NCBI,生物项目号PRJNA858084, SRA序列SRR2081042144, SRR2081042245, SRR2081042346, SRR2081042447, SRR2081042548, SRR2081042649, SRR2081042750.基因组组装可在EMBL-EBI获得,生物项目号PRJEB5501351.基因组组装、基因注释和重复注释文件都在泽诺多下面https://doi.org/10.5281/zenodo.682432352,以及http://www.grapegenomics.com53.基因组浏览器和爆破工具可用于每个砧木在http://www.grapegenomics.com53.
技术验证
基因组组装评估二倍体序列和基因含量的完整性,以及正确的单倍型分期。每组19个假分子的平均大小为494.5±5.5 Mbp(二倍体基因组大小:1015.0±7.9 Mbp,补充图1),接近流式细胞术估计的亲本单倍体基因组大小(499.3±37.3 Mbp)54)表明这三个基因组是完全组装起来的。只有36.1 Mbp(3.5%)、19.9 Mbp(2.0%)和23.3 Mbp(2.3%)的草案序列不能被放置到101-14 mgmt、110R和Kober 5BB基因组的任何假分子中。未放置序列以重复序列为主(68.0%±12.3%)。这些结果与最新发布的诉酿酒用葡萄PN40024参考单倍体基因组组装,其中27.4 Mbp(5.6%)的位置尚未确定55.
利用植物基因的保守单拷贝直链(BUSCOs)和植物基因的单拷贝基因含量来评估每组19个假分子的基因空间完整性诉酿酒用葡萄PN40024。在每组假分子中发现BUSCO模型的完整副本为98.1±0.14%(补充表)1).同样,PN40024的单拷贝基因几乎全部与每组假分子对齐(95.01%±0.3%)。未放置序列中存在的基因空间限制在BUSCO模型的0.69±0.8%和PN40024基因的1.79±0.8%。基因空间的完整性又有力地证明了该组合是三个砧木二倍体基因组的完整代表。在101-14 Mgt的两个单倍型上,我们发现了更多的基因位点(33,379±328),而在110R和Kober 5BB(28,584±863)。需要进一步的全基因组基因表达分析,以确定在101 - 14mgt中鉴定的基因位点数量是否与其他砧木中表达的转录本数量相对应。
使用每个砧木的谱系信息(图。1 b),我们将每个假分子分配给它的亲本葡萄属物种,即诉锐利,诉rupestris,或诉? ?.对于每个假分子,我们确定了具有最高基因序列相似性的三对单倍型,并将它们分配给共享的亲本葡萄属物种。这使我们能够手动检测和纠正在草稿序列组装或假分子搭建过程中引入的相位错误(即单倍型开关)(图2)。3).对每个假分子的6个单倍型进行全序列比较,结果表明单倍型归属相同葡萄属种间相似性(80.5%±1.4%)高于种间差异(74.0%±3.3%);pvalue = 0.0003, W = 142, n = 30未配对Wilcoxon秩和检验;无花果。3 b, c).这些结果表明,3个砧木基因组的单倍型分期正确。尽管3个砧木基因组的序列多态性水平不同,但与起源种无关,其假分子高度共线。综合考虑基因序列相似性、基因顺序和物理位置,73.1%±3.5%的蛋白编码位点在具有相同亲本来源的单倍型之间至少在一个共线块上,不同物种单倍型之间的蛋白编码位点在71.5%±3.5%(补充图)2).总的来说,平均82.4%±2.6%的基因组序列被共线块覆盖(补充图3.),这反映了它们的染色体结构有显著的保守性葡萄属物种。
代码的可用性
用于基因结构和功能注释的管道可在https://github.com/andreaminio/AnnotationPipeline-EVM_based-DClab.
参考文献
Millardet,。不同原则的历史eteS et espéces原产地eRicaine qui re葡萄根瘤蚜(G. Masson,巴黎,1885)。
多德森·彼得森,j.c.等.基于加州Wolpert试验的葡萄砧木育种及其性能。在Cantu, D. & Walker, m.a.(编)葡萄基因组, 301 - 318,https://doi.org/10.1007/978-3-030-18601-2_14(施普林格国际出版,Cham, 2019)。
Pongracz, D. P.葡萄藤的根茎。出版商:开普敦(南非)大卫·菲利普(1983)。
Ravaz, L。我是葡萄藤eRicaines: porte-greffes et producteurs-directs: caractéres, aptitude(。古利特,1902)。
Riaz来说,S。等.葡萄砧木遗传多样性及亲本分析。理论与应用遗传学132, 1847 - 1860,https://doi.org/10.1007/s00122-019-03320-5(2019)。
L. P.砧木选择。加州的酿酒葡萄品种。加州大学奥克兰分校,美国12 - 15(2003)。
Dodson Peterson, J. C. & Andrew Walker, M.葡萄砧木对接穗发育和衰老启动的影响。催化剂:从发现到实践1现年48岁的https://doi.org/10.5344/catalyst.2017.16006(2017)。
郑丽华,郑丽华,杨晓明。葡萄砧木对植物寄生线虫的抗性研究。线虫学杂志44, 377-386(2012)。
Rahemi, A, Dodson Peterson, J. C. & Lund, K. T。葡萄砧木和近缘种(施普林格国际出版社,Cham, 2022)。
Girollet, N。等.葡萄河岸葡萄基因组的从头分期组装。科学数据6, 1 - 8,10 /ghdrm3(2019)。
帕特尔。等.美洲土著抗寒葡萄藤riparia Michx基因组草图。马尼托巴省37。园艺研究710 / gg53d4。ISBN: 4143802003162出版社:施普林格US(2020)。
程浩,李国涛,冯晓霞,张海华,李海华。基于相位装配图的单倍型解析从头装配。自然方法18中国农业科学,2017,10/ghz4s5(2021)。
Minio, A., Cochetel, N., Vondras, A. M., Massonnet, M. & Cantu, D.从草图基因组序列组装完整的二倍体阶段染色体。G3 | |基因组遗传基因jkac143,https://doi.org/10.1093/g3journal/jkac143(2022)。
陈国强等.分阶段二倍体基因组组装单分子实时测序。自然方法13, 1050-1054(2016)。10 / f9fv4w。
Blanco-Ulate, B., Vincenti, E., Powell, a. L. & Cantu, D.番茄转录组和突变分析表明植物应激激素在水果和灰霉病相互作用中的作用。植物科学前沿4, 1 - 16,10 /gkzg3v(2013)。
Manni, M., Berkeley, m.r., Seppey, M., Simão, f.a. & Zdobnov, e.m. BUSCO更新:新颖和精简的工作流程以及更广泛和更深入的系统发育覆盖,用于真核生物、原核生物和病毒基因组评分。分子生物学与进化38, 4647 - 4654,https://doi.org/10.1093/molbev/msab199(2021)。
", O。等.葡萄基因组序列显示主要被子植物门的祖先六倍化。自然449, 463-467, 10/ckfnh2(2007)。
邹,C。等.用rhAmpSeq单倍分型葡萄共线核心基因组提高了不同属的标记转移性。自然通讯11, 413, 10/ghdrnk。发行商:施普林格US(2020年)。
Massonnet, M。等.葡萄性别决定的遗传基础。自然通讯11, 2902, 10/gjxrfm。发行商:施普林格US(2020年)。
李,H. Minimap2:核苷酸序列的成对比对。生物信息学34, 3094-3100, 10/gdhbqt。_eprint: 1708.01492(2018)。
Cochetel, N。等.染色体规模的二倍体组装Muscadinia rotundifolia基因组支持染色体融合和抗病基因扩增期间葡萄属而且Muscadinia散度。G3 | |基因组遗传基因11jkab033,https://doi.org/10.1093/g3journal/jkab033(2021)。
Au K. F, Underwood, J. G., Lee L. & Wong W. H.通过短读对齐提高PacBio长读精度。《公共科学图书馆•综合》7, 1 - 8,10 /f383xz(2012)。
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学30., 2114-2120, 10/f6cj5w(2014)。
哈斯,b.j.。等.利用Trinity平台从RNA-seq中重建转录本序列,用于参考生成和分析。自然的协议8, 1494-1512, 10/f22qdv(2013)。
Pertea, M。等.StringTie可以改进RNA-seq读取的转录组重建。自然生物技术33, 290-295, 10/f64s85(2015)。
Kim, D., Langmead, B. & Salzberg, S. L. HISAT:低内存需求的快速拼接对齐器。自然方法12, 357-360(2015)。10 / f67q59。
Li, W. & Godzik, A. Cd-hit:用于聚类和比较大组蛋白质或核苷酸序列的快速程序。生物信息学22, 1658-1659, 10/ct8g72(2006)。
哈斯,b.j.。等.利用最大转录本比对集改进拟南芥基因组注释。核酸研究31, 5654-5666, 10/cgkkwd(2003)。
斯莱特,G. S. C. &伯尼,E.生物序列比较的启发式自动生成。BMC生物信息学61 - 11,https://doi.org/10.1186/1471-2105-6-31(2005)。
哈斯,b.j.。等.使用EVidenceModeler和程序组装拼接对齐自动真核基因结构注释。基因组生物学9,https://doi.org/10.1186/gb-2008-9-1-r7(2008)。
Stanke, M., Tzvetkova, A. & Morgenstern, B. AUGUSTUS在EGASP:使用EST,蛋白质和基因组对齐来改进人类基因组中的基因预测。基因组生物学7(增刊1),1 - 8,https://doi.org/10.1186/gb-2006-7-s1-s11(2006)。
Seppey, M., Manni, M. & Zdobnov, e。基因预测:方法和协议,分子生物学方法1962卷(施普林格纽约,纽约,NY, 2019)。
洛萨兹,A., Ter-Hovhannisyan, V., Chernoff, Y. O. & Borodovsky, M.基于自训练算法的新型真核基因组基因鉴定。核酸研究33, 6494-6506(2005)。10 / bz9c2v。
Korf, I.新基因组中的基因发现。BMC生物信息学5, 59, 10/cdvb5x。ISBN: 1471-2105(电子)(2004)。
Smit, A. F. A., Hubley, R. & Green, P. repeatmask Open-4.0。页数:2013-2015http://www.repeatmasker.org(2013)。
布赫芬克,B,谢,C. &胡森,D. H.快速和敏感的蛋白质比对金刚石。自然方法1259-60,https://doi.org/10.1038/nmeth.3176(2015)。
布赫芬克,B.,路透,K. & Drost, H.-G。使用DIAMOND在生命树尺度上对敏感蛋白进行比对。自然方法18, 366 - 368,https://doi.org/10.1038/s41592-021-01101-x(2021)。
琼斯,P。等.InterProScan 5:基因组级蛋白质功能分类。生物信息学(牛津,英国)30., 1236 - 40,10 /f53532(2014)。
Conesa,。等.Blast2GO:用于功能基因组学研究的注释、可视化和分析的通用工具。生物信息学21, 3674 - 3676,https://doi.org/10.1093/bioinformatics/bti610(2005)。
王,Y。等.MCScanX:用于检测和进化分析基因同步性和共线性的工具包。核酸研究40, 1 - 14,10 /fzn3xm(2012)。
Wu, T. D. & Watanabe, C. K. GMAP: mRNA和EST序列的基因组定位和比对程序。生物信息学21, 1859-1875, 10/cjb8q8(2005)。
昆兰,a.r.和霍尔,i.m. BEDTools:一套用于比较基因组特征的灵活工具。生物信息学26, 841 - 842,https://doi.org/10.1093/bioinformatics/btq033(2010)。
Marcais G。等.MUMmer4:一个快速和通用的基因组比对系统。PLoS计算生物学14, e1005944, 10/gcw64s(2018)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810421(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810422(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810423(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810424(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810425(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810426(2022)。
序列读存档https://identifiers.org/ncbi/insdc.sra:SRR20810427(2022)。
ENA欧洲核苷酸档案,https://identifiers.org/ena.embl:PRJEB55013(2022)。
Minio, A., Cantu, D., Cochetel, N., Massonnet, M. & Figueroa-Balderas, R.支持数据:葡萄砧木110R, Kober 5BB和101-14 Mgt的HiFi染色体规模二倍体组合。Zenodohttps://doi.org/10.5281/zenodo.6824323(2022)。
Minio, a . & Cantu, D. Grapegenomics.com:一个提供野生和栽培葡萄基因组数据和分析工具的门户网站。Zenodohttps://doi.org/10.5281/zenodo.7027886(2022)。
Lodhi, M. A. & Reisch, B. I.核DNA含量的葡萄品种,栽培品种,和其他属的葡萄科。理论与应用遗传学90, 11 - 16,10 /cgwkss(1995)。
Canaguier,。等.葡萄藤参考基因组组装(12X.v2)及其注释(VCost.v3)的新版本。基因组学数据1456 - 62https://doi.org/10.1016/j.gdata.2017.09.002(2017)。
确认
的RNAseq数据诉rupestris由康奈尔大学的杰森·朗多博士提供。这项工作由美国国家科学基金会拨款#1741627资助,部分资金由路易·p·马丁尼葡萄栽培基金支持。
作者信息
作者及隶属关系
贡献
a.m.、北卡罗来纳和华盛顿构思了这项工作。A.M.进行生物信息学分析。R.F.-B。完成与项目相关的所有湿实验室活动。上午,北卡罗来纳,下午,华盛顿写的手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
米尼奥,A.,科舍泰尔,N.,马索内,M.。et al。葡萄砧木110R、Kober 5BB和101-14 Mgt的HiFi染色体规模二倍体组合。科学数据9660(2022)。https://doi.org/10.1038/s41597-022-01753-0
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41597-022-01753-0