摘要
严重急性呼吸综合征冠状病毒2型(SARS-CoV-2)感染是全球一个主要的卫生问题。基因组流行病学是评估2019冠状病毒病大流行的重要工具。通过对SARS-CoV-2的基因组分析,已经报告了几个突变。在本研究中,我们对大流行早期(2020年3月- 6月)和局部传播突然激增(2020年8月- 9月)收集的标本中病毒基因组特征的30个全基因组序列进行了突变和系统发育分析。处于感染早期阶段的4个样本属于B.6谱系,属于新加坡和马来西亚同时采集的样本的分支,而乘坐救援飞机返回的5个样本的谱系为B. 1.36.1(3个来自印度)、B.1.1(1个来自印度)和B.1.80(1个来自中国)。然而,没有证据表明这些返回者在当地传播。此外,在突然激增的本地传播中收集的所有19个全基因组序列均显示为B.1.36谱系。SARS-CoV-2感染的第二波激增与一种变体(B.1.36)的单一传入有关,这可能是严格限制国际旅行和遏制措施造成的。这些基因组数据为疾病控制和预防策略提供了有用的信息。
简介
由严重急性呼吸综合征2型冠状病毒(SARS-CoV-2)引起的新型呼吸道冠状病毒病(COVID-19)在全球范围内引起了广泛关注。缅甸于2020年3月报告了第一例COVID-19病例,由于采取了有效的病毒控制措施,到2020年7月底仅报告了少数当地病例。这些措施包括对所有返回者和确诊病例的接触者实施21天的严格隔离政策;第一周及21天前对所有接触者进行实时荧光定量PCR检测,排除感染,所有确诊病例均住院监测,直至检测到高病例连续2例RT-PCR阴性和居家隔离。此外,自2020年3月起,开始限制国际航班,以防止病毒从其他国家传入。只有选择性地允许乘坐救援航班返回的缅甸公民,并在抵达后21天的隔离期内用分子方法对他们进行了两次棉签检查。因此,截至2020年8月18日,缅甸仅报告了379例确诊病例1.然而,8月19日之后,缅甸西部若开邦报告了与已知病例没有接触史的当地病例突然增加。一周内,特大城市仰光报告了本地传播。截至2020年9月30日,共记录确诊病例13373例,死亡310例2.
新型冠状病毒的分子流行病学研究为制定新型冠状病毒的防控策略提供了有价值的信息3.,4.突变分析和系统发育研究表明,起源于武汉的病毒被许多国家的突变变体所取代5.然而,到目前为止,缅甸还没有关于SARS-CoV-2基因组分析的已知研究。
具有出国旅行史的返回者输入确诊病例的初步数据表明,来自不同地理领土的不同变体已被引入缅甸。我们报告了在缅甸检测到的SARS-CoV-2的全基因组序列分析,为病毒在该国的传播和传播动态提供遗传证据。
方法
道德声明
该研究由医学研究部机构审查委员会(IRB)审查并批准(批准ID: Ethics/DMR/2020/073)。在本研究中,我们使用剩余的匿名样本进行诊断;IRB放弃了知情同意。该研究在缅甸卫生研究登记处注册,ID: PLRID-00476_V5。所有程序都是根据负责任的研究行为的机构准则进行的。
研究人群
本研究使用分子诊断后确认阳性的鼻咽拭子剩余样本。根据ARTIC协议6其中涉及Ct值较低的样本(real-time PCR检测所有靶基因均< 30个周期)。根据每日确诊的COVID-19阳性感染率,将大流行浪潮的早期阶段定义为2020年8月15日之前的持续时间,之后在缅甸观察到感染的突然激增,这是第二波。通过随机选择返回者和当地报告的病例,对低Ct样本进行抽样。我们在大流行浪潮的早期阶段选择了5个当地报告的样本,以及5个来自中国和印度返回者的样本。2020年9月采集了来自若开邦感染突然激增的9个样本和来自菲律宾的1个返回者样本,以及另外10个样本,其中9个是仰光的死亡病例(补充表)1,补充图。1).
提取RNA,确认感染
我们遵循之前描述的全基因组测序程序7.简单地说,根据制造商的说明,使用QIAGEN QIAamp virus RNA Mini Kit从鼻咽拭子中提取核酸。样本中SARS-CoV-2感染由新型冠状病毒(2019-nCoV)核酸诊断试剂盒(sanure Biotech, Republic of China)采用BioRad CFX96触摸式实时PCR检测系统,按照厂家说明书进行检测。
排序程序
根据ARTIC nCo-V-2019方案进行SARS-CoV2全基因组扩增6.简单地说,cDNA是用GoScript逆转录酶试剂盒(Promega)合成的。使用Q5热启动高保真DNA聚合酶(NEB)进行扩增。在使用引物池的每次扩增中包含一个阴性对照。采用毛细管电泳(LabChip GXTouch 24核酸分析仪)检测扩增子。阴性对照必须在每次扩增电泳中均无可见条带,以排除污染。用Illumina样品纯化珠(Illumina, USA)纯化400 bp重叠扩增子。清理扩增子用Qubit3 dsDNA HS (High sensitivity) Assay Kit (Invitrogen)定量,文库制备前用Nextera DNA Flex文库制备Kit (Illumina)归一化。我们使用500ng输入扩增DNA用于文库制备。采用毛细管电泳法进行了质量检测,并用Qubit3进行了定量。 Normalization was carried out before pooling on ten samples in each run. These pooled samples were denatured by NaOH, and 11 pmol libraries were loaded with 10% PhiX spike control to run 151 paired-end in MiSeq next-generation sequencer.
序列分析
输出序列通过BBDuk和BBMap (BBTools 38.57)进行读取质量控制。序列由基因组检测病毒工具(https://www.genomedetective.com/)8.比对SARS-CoV-2参考基因组序列(NC_045512.2)。变量调用是由CoV-GLUE完成的(http://cov-glue.cvr.gla.ac.uk/)9和NextClade v0.5.0 (https://clades.nextstrain.org/)10分配分支。通过穿山甲COVID-19谱系分配器评估SARS-CoV-2谱系(https://pangolin.cog-uk.io)11.与参考序列武汉-胡-1 (NC_045512.2)比对,用iTOL v5.6.3 (https://itol.embl.de/).
我们向NCBI SRA提交了序列,登录号为SAMN15733924-15733932、SAMN15722976、SAMN15921629-SAMN15921638和SAMN16362251-SAMN16362260。序列也被保存在GISAID (https://www.gisaid.org/).
结果
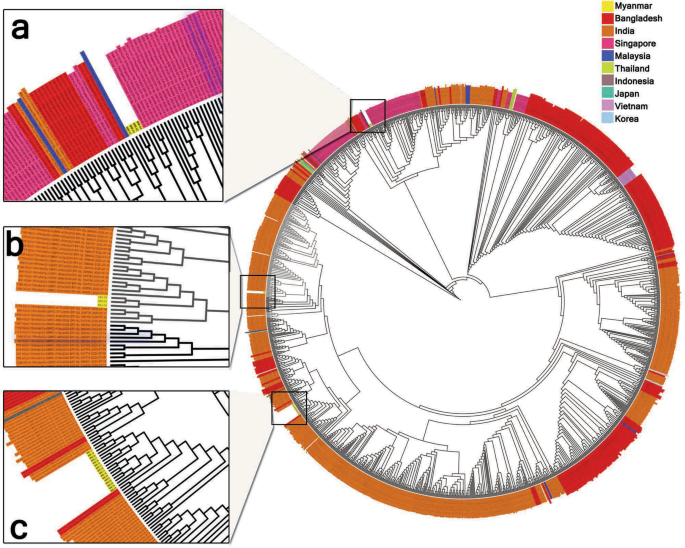
我们分析了30例COVID-19患者的鼻咽拭子样本,进行全基因组分析。将Reads与参考基因组对齐,生成一致序列。剔除低质量短reads和适配器(质控)后,reads的中位数为2,228,704条,平均测序深度为8376倍,覆盖所有样本的99%以上基因组。我们已经确定了平均12个突变,其中包含30个同义替换,34个非同义替换,以及5′utr位点上的5个突变1).
在大流行早期传播的五宗本地病例中,有四宗为B.6谱系(穿山甲分析:https://pangolin.cog-uk.io/),一个是B1.1血统。从印度返回的3例为B.1.36.1, 1例为B.1.1。此外,我们在2020年8月之前仅检测到一个本地传播病例样本,在核蛋白(N)基因上的刺突蛋白D614G突变和三核苷酸群突变28,881-28,883 GGG>AAC (R203K和G204R)。对来自印度(4例)、中国(1例)和菲律宾(1例)的海归样本进行突变分析,均为G型(D614G突变体)。
在我们的分析中,在病例突然激增期间,包括了来自若开邦的9个样本和来自仰光市的10个样本。全基因组分析均显示B1.36谱系。19个样本中有相同的11个突变(见表2)1).ORF1b均显示同义突变G18756T。此外,我们观察到所有来自突发性局部传播的样本中,5’utr中C241T, ORF1a中C3037T同义突变,ORF1b中C14408T (P314L), ORF1b中C18877T同义突变,Spike中C22444T同义突变,ORF3a中G25494T同义突变,ORF3a中G25563T (Q57H),膜蛋白中C26735T,核蛋白中C28854T (S194L)(表2)1).
系统发育分析显示,在缅甸大流行波的早期阶段,SARS-CoV-2可能多次传入。当时,当地报告的病例与新加坡和马来西亚位于同一分支,返回者的样本与印度位于同一分支。然而,在大流行波的突然激增中观察到一个单一的传入病例,该病例与孟加拉国和印度的序列处于同一分支。此外,死亡病例中的SARS-CoV-2变体与康复病例中的SARS-CoV-2变体没有差异。
系统发育分析显示,缅甸大流行波早期报告的所有4例病例与新加坡和马来西亚报告的病例在同一分支中观察到,其中一例与印度在同一分支中确认。然而,在感染突然激增时收集的所有病例都显示为B.1.36,并且位于印度和孟加拉国的同一分支(图2)。1).
讨论
遗传流行病学为疾病的预防和控制提供了有用的信息。全基因组数据可用于当地传播病例的疾病跟踪,为决策者提供基因组证据。基因组研究显示,SARS-CoV-2的突变率与影响全球疾病传播的其他RNA病毒的突变率相似12.基因组监测对于进一步采取遏制、控制和预防疾病的行动来了解疾病的传播非常重要13.
在报告的突变中,非同义突变D614G于2020年初首次被观察到,但在几个星期内就在许多国家广泛传播。Spike D614G突变被认为可以在体外感染SARS-CoV-2,这表明它增强了病毒的传染性,导致全球G变体的增加14,15.
在本研究中,在早期大流行感染的局部传播病例中,仅观察到一例GR变异(spike蛋白为D614G, ORF14为G204R)。在返回者中,观察到来自印度的GH变异(ORF3a中的D614G和Q57H)和来自中国和菲律宾的GR变异。由于严格的检疫规定,这些返回者的感染可能不会在缅甸当地传播。
谱系B.6主要于2020年3月和4月在东南亚发现16.在我们的研究中,本地传播病例与从新加坡返回的确诊病例有一次和二次接触史。在四月和五月从印度返回的样本中,我们鉴定出血统B.1.36.1。同时,来自中国和菲律宾海归的样本均为血统B.1.80(中国)和血统B.1.1(菲律宾)。根据穿山甲分析,B.1.36.1是在印度流传的印度血统。从印度入境人员中检测到的SARS-CoV-2呈B.1.36.1血统。这两名从印度返回的人是肾移植接受者和捐赠者,他们一直住院,直到连续两次RT-PCR阴性。因此,这些病例可能不会在缅甸进一步传播。
但8月15日之后,在缅甸西部若开邦,在没有已知与确诊病例接触史的情况下,当地传播突然增加,随后在缅甸大城市仰光等地区出现。我们对这次突然激增的19个样本的分析显示GH变异,B.1.36谱系仅具有相似的突变模式。
5'UTR区域突变可能影响病毒的转录和复制,导致病毒感染周期加快17,18.在我们的研究中,所有D614G变体都与C241T在5'UTR共存。虽然G18756T突变的作用尚不清楚,但在印度有报道,在南美洲有第二次感染样本19.
在本研究检测到的所有突变中,前导序列突变C241T与三个重要突变C3037T、C14408T和A23403G共同进化,分别导致nsp3(同义突变)、RNA引物酶(P323L)和穗糖蛋白(S蛋白,D614G)的氨基酸突变。这些共突变是RNA复制和与ACE2受体结合的关键蛋白质20..然而,感染的毒力可能与本研究中检测到的变异无关,因为死亡病例的SARS-CoV-2基因特征与康复病例相似。
缅甸限制国际旅行,只有返回缅甸的缅甸公民被允许乘坐救援航班。因此,在2020年8月之前的早期大流行浪潮中,尽管观察到多次引入SARS-CoV-2,但仅观察到少数本地病例。尽管采取了遏制措施,但仍观察到一次可能来自印度或孟加拉国(世系B.1.36)的GH变体传入并传播,导致缅甸于2020年8月下旬突然爆发COVID-19疫情。
我们研究的一个局限性是只包括了实验室确认样本的一个子集。应进一步开展分子监测研究,以更好地了解缅甸的SARS-CoV-2。
结论
在我们的研究中,对全基因组数据的突变和系统发育分析表明,缅甸有多次引入SARS-CoV-2。自2020年3月起,出现SARS-CoV-2的本地传播。尽管如此,基因组数据表明,早期本地传播病例为B.1.6谱系,这与2020年8月后本地传播病例突然激增的样本(B.1.36谱系)不同。尽管这些毒株在致病性和毒力的另一个方面的实际影响尚未确定,但在控制COVID-19大流行时,应深切关注与感染突然激增相关的病毒序列动态。
参考文献
卫生和体育部。缅甸COVID-19疫情报告第134号。第134号报告,公共卫生部。卫生和体育部中央流行病学股,缅甸内比都,https://www.mohs.gov.mm/page/11947(2020)。
卫生和体育部。缅甸COVID-19疫情报告。第136号报告(公共卫生部中央流行病学股)。卫生和体育部,缅甸内比都,https://www.mohs.gov.mm/page/11950(2020)。
Sironi, M。et al。SARS-CoV-2和COVID-19:遗传、流行病学和进化的角度。感染。麝猫。另一个星球。84, 104384-104384(2020)。
Yuki, K., Fujiogi, M. & Koutsogiannaki, S. COVID-19病理生理学:综述。中国。Immunol。215, 108427-108427(2020)。
Mercatelli, D.和Giorgi, F. M. SARS-CoV-2突变的地理和基因组分布。前面。Microbiol。11, 1800(2020)。
Sevinsky, J。et al。利用ARTIC协议测序illumina MiSeq的SARS-CoV-2:第一部分:tiling PCRProtocols.io。https://doi.org/10.17504/protocols.io.bfefjjbn(2020)。
NA管道研发et al。COVID-19 ARTIC v3 illumina文库构建及测序方案V.3。Protocols.io.https://doi.org/10.17504/protocols.io.bgq3jvyn(2020)。
Cleemput, S。et al。Genome Detective冠状病毒分型工具,用于快速识别和表征新型冠状病毒基因组。生物信息学.36(11): 3552 - 3555。https://doi.org/10.1093/bioinformatics/btaa145(2020)。
Singer, J., Gifford, R., Cotten, M. & Robertson, D. CoV-GLUE:用于跟踪SARS-CoV-2基因组变异的web应用程序。2020060225.https://doi.org/10.20944/preprints202006.0225.v1(2020)。
哈德菲尔德,J。et al。Nextstrain:实时跟踪病原体进化。生物信息学34, 4121-4123(2018)。
兰姆伯特,一个。et al。SARS-CoV-2谱系的动态命名建议以协助基因组流行病学。Microbiol Nat。5, 1403-1407(2020)。
赵,Z。et al。SARS冠状病毒基因组的中等突变率及其意义。BMC另一个星球。医学杂志。4, 21(2004)。
Ortiz-Prado E。et al。SARS-CoV-2病毒和2019冠状病毒病(COVID-19)的临床、分子和流行病学特征,综合文献综述。成岩作用。Microbiol。感染。说。98, 115094(2020)。
柯尔柏B。et al。追踪SARS-CoV-2峰值的变化:D614G增加COVID-19病毒传染性的证据。细胞182, 812 - 827。e819(2020)。
张,L。et al。SARS-CoV-2刺突蛋白D614G突变减少S1脱落,增加传染性。Nat Commun。11(1): 6013。https://doi.org/10.1101/2020.06.12.148726v1(2020)。
钟元明。et al。SARS-CoV-2谱系B.6是马来西亚早期大流行传播的主要因素。PLoS nel Trop Dis。14(11): e0008744。https://doi.org/10.1371/journal.pntd.0008744(2020)。
刘鹏,莱博维茨J.(2010)冠状病毒5 '和3 '非翻译区的RNA高阶结构及其在病毒复制中的作用。in: Lal S.(编辑)sars冠状病毒的分子生物学47 - 61。https://doi.org/10.1007/978-3-642-03683-5_4(施普林格,柏林,海德堡,2010)。
金,D。et al。SARS-CoV-2转录组结构。细胞181, 914-921(2020)。
普拉多-维瓦尔等人。由系统遗传学上不同的SARS-CoV-2变体再次感染COVID-19,这是南美洲首次确认的事件。SSRN.https://doi.org/10.2139/ssrn.3686174(2020)。
尹,C.冠状病毒SARS-CoV-2基因分型:方法和意义。基因组学112, 3588-3596(2020)。
确认
这项研究得到了缅甸卫生和体育部IR/OR2020赠款计划的支持。
作者信息
作者及隶属关系
贡献
M.h.n, h.o.m.s, k.t.a。,H.M.T., Z.T.H., W.W.A., Y.Y.K. coordinated the protocol and experiments. M.H.N., H.O.M.S., K.T.A. performed the NGS experiments and data analysis, A.K.K., A.Z.L., T.W.M., P.W.E., M.M.W., A.A.W., Y.M.H., K.M.Z., K.T.H., L.L.S., N.A.T.O., H.L., N.C.N.M., K.T.Y., K.L.H., Y.K.K., T.H.H.H., K.M.O., H.A., S.S.H., S.M.W., W.M.T., T.T.A., M.S.H., W.Y.M., P.P.T. performed the RNA extraction and laboratory experiments. M.H.N., H.M.T. wrote the manuscript, all read and approved the manuscript.
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
纽特,m.h.,索,h.o.,艾,K.T.et al。与缅甸2020年单一病毒株传入有关的严重急性呼吸综合征冠状病毒感染激增。Sci代表11, 10203(2021)。https://doi.org/10.1038/s41598-021-89361-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-021-89361-7