摘要
自2019年12月以来,一种导致严重急性呼吸系统综合征(SARS-CoV-2)的新型冠状病毒导致了一场重大的大流行局势。B.1.1.7毒株作为一种高传染性变体的出现,加速了全世界对追踪SARS-CoV-2变体发生的兴趣。同样,还描述了其他极具传染性的变异,由于大流行病持续的时间很长,预计还会发现其他变异。所有描述的SARS-CoV-2变体在编码Spike蛋白的基因中都存在几个突变,参与宿主受体识别和进入细胞。因此,在此,我们建议将重点放在SPIKE区域,以增加候选样本的数量,而不是对整个病毒基因组进行测序以跟踪变异;这是加速诊断的一个重要方面,也是变体出现/进展监测的一个重要方面。这种概念验证研究同时完成了群体规模的诊断和变异跟踪。该策略依赖于(1)使用便携式MinION DNA测序仪;(2) DNA条形码和以SPIKE基因为中心的变异跟踪,增加每次检测的候选数量;(3)借助我们的软件RETIVAD进行实时诊断和变体跟踪监控。 This strategy represents an optimal solution for addressing the current needs on SARS-CoV-2 progression surveillance, notably due to its affordable implementation, allowing its implantation even in remote places over the world.
介绍
自2019年12月以来,导致严重急性呼吸系统综合征(SARS-CoV-2)的新型冠状病毒的出现造成了一场重大大流行,迄今已导致全球近300万人死亡。2020年初,开展了主要的病毒诊断工作,包括制定新的策略,旨在(1)缩短诊断时间,(2)降低每次检测的成本,(3)提供人群规模的解决方案,以及(4)提供高灵敏度和特异性,特别是在区分无症状病例方面。虽然成本和每次检测所需的时间在早期就通过开发基于免疫的测试得到了解决1,它们相对于病毒核酸靶向诊断的敏感性最初受到质疑,但最终在几个国家被接受,主要是因为基于RT-qPCR检测的诊断通量低2。
为了充分利用病毒核酸靶向诊断的优势,同时提高候选病毒的吞吐量,提出了基于大规模并行DNA测序的群体诊断策略3.,4,5。这些努力依赖于(1)在样品制备过程中结合DNA分子条形码;(2)通过使用大规模平行DNA测序,汇集条形码样本进行“一次运行”诊断分析;(3)利用生物信息学处理对诊断结果进行分层。虽然这些策略被证明可以将诊断能力提升到每次检测超过数千个候选对象,但它们需要一个DNA测序平台,其中大型仪器(Illumina Sequencers)由专业技术工程师驱动。因此,这一策略仍然适用于高度发达的国家(由于所需DNA测序仪的成本过高),这些国家还需要专门的管道策略来将收集的样品运送到测序中心。
除了诊断要求之外,发现高传染性的SARS-CoV-2变种b .1.1.7——起源于英格兰南部——极大地加速了全世界对追踪SARS-CoV-2变种发生的兴趣。最近描述了另外两种极具传染性的变种,即在巴西马瑙斯市发布的P.1型和最初在南非发现的B.1.351型,预计还会发现其他变种,这并不令人意外,特别是由于大流行病持续的时间很长6。
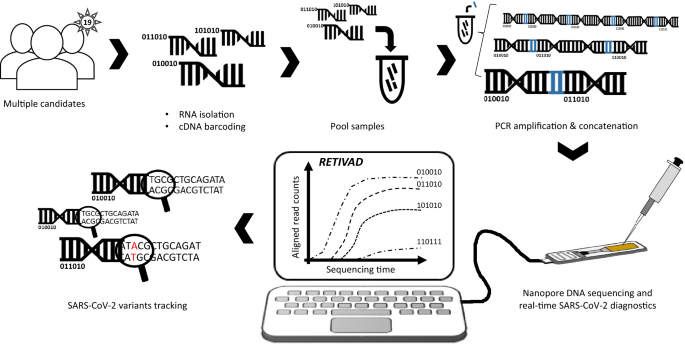
在此,我们描述了一项概念验证研究,用于群体规模的诊断和变体跟踪,使用MinION牛津纳米孔技术(ONT)进行大规模平行DNA测序。ONT MinION测序仪以其便携性,降低成本和使用简单而闻名;因此,即使在世界各地的偏远地区,这也是一项适用的战略(见7)。事实上,这项技术被广泛用于SARS-CoV-2全基因组测序,特别是使用由ARTIC网络(https://artic.network/ncov-2019)8。此外,还描述了一种结合环介导等温扩增(LAMP)和纳米孔测序的Covid-19诊断策略9。
除了专用的分子生物学方案外,我们还提供计算解决方案(RETIVAD: Real-Time variant Detector),在测序过程中实时提供诊断结果;可以争取时间撰写诊断报告,这在当前大流行形势下至关重要。最后,我们还证明了我们的策略能够跨多个候选对象进行SARS-CoV-2变体的检测,特别是通过靶向SPIKE基因而不是覆盖整个病毒基因组(图2)。1)。这种靶向变异的跟踪策略允许在分析过程中增加~ 10倍的潜在测序覆盖范围,从而允许将该资源重新分配给突变同时筛选大量候选基因。
总之,提出的策略满足了目前对群体诊断测定计数的需求,并区分了各种SARS-CoV-2变体,这些变体被认为可能呈现可变的感染特性和临床结果6,10。
结果
用于SARS-CoV-2多候选诊断的组合分子条形码策略
为了使用MinION测序仪对多个候选株进行SARS-CoV-2诊断,我们将常规的反转录测定与特定的PCR扩增相结合,从而产生长DNA串联体。这种策略允许靶向短扩增子,就像那些用于定量实时PCR策略的扩增子一样,但同时利用牛津纳米孔技术的长片段测序能力。
逆转录(RT)是用Covid-19特异性引物进行的,此外还有一个独特的分子条形码(20nt长度)和一个共同的序列适配器(Gibson序列;30nt长度)(图。2A).因此,多个候选样本被反向转录,每个候选样本具有独特的分子条形码,允许在大量平行DNA测序后进行鉴定。
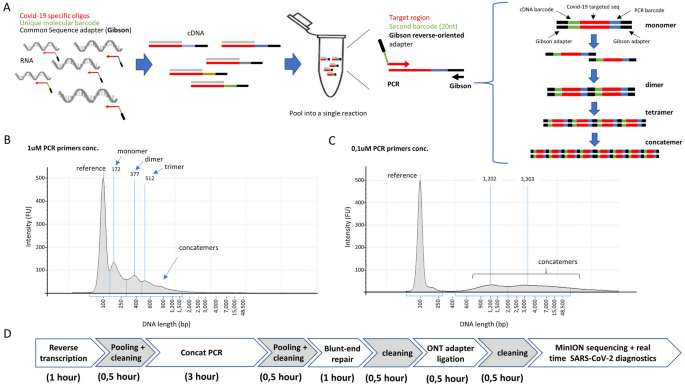
双分子DNA条形码和pcr驱动串联技术在SARS-CoV-2诊断中的应用(一个)利用针对Covid-19区域的寡核苷酸进行逆转录,但每个样本还包含一个独特的分子条形码和一个共同的适配器序列(Gibson)。反转录测定(cDNA)执行多个候选汇集在一起进行扩增。利用针对常见吉布森序列的寡核苷酸和含有第二分子条形码以及互补吉布森序列的covid -19特异性引物进行PCR扩增。在PCR周期中,由于Gibson侧翼序列,扩增产物能够连接。我们把这种特殊的PCR策略称为“Concat-PCR”。(B) DNA电泳图(Agilent TapeStation系统)显示了PCR引物在1uM终浓度下获得的串联产物。(C)用终浓度为0.1uM的PCR引物进行串联PCR试验的DNA电泳图,显示与(B)。(D)时间表说明了样品制备所需的各种分子生物学步骤,然后将样品加入MinION DNA测序。
在RT检测后,候选人的样品被汇集到一起进行PCR扩增成一个单一的反应。为此,将第二种Covid-19特异性引物与吉布森引物一起使用,该引物除了具有独特的分子条形码(20nt长度)外,还具有常见吉布森序列适配器的反向序列(30nt长度)。2A).该策略允许合并第二个唯一的分子条形码,从而为每个候选物提供组合条形码策略,这对于增加候选物的数量非常有用,而不必线性增加要购买的引物数量;这方面是主要的相关性,由于他们的长(bbb70nt)。
在PCR扩增产物的两端存在吉布森序列,可以在扩增过程中产生串联体(图2)。2A)。虽然这一过程在PCR扩增过程中以不受控制的方式发生,但低引物浓度显示优先产生长串联体(图2)。2B, C)。因此,用SPRI-select试剂(Beckman;B23318),没有丢失短放大子的风险。此外,长串联体的使用将MinION的平均测序覆盖率提高了约9倍(即100万个串联体序列对应900万个单体;数字S1和无花果。3.E),这样就可以像以前基于使用Illumina仪器的人群规模诊断工作一样,筛选大量的候选者3.,4,5。
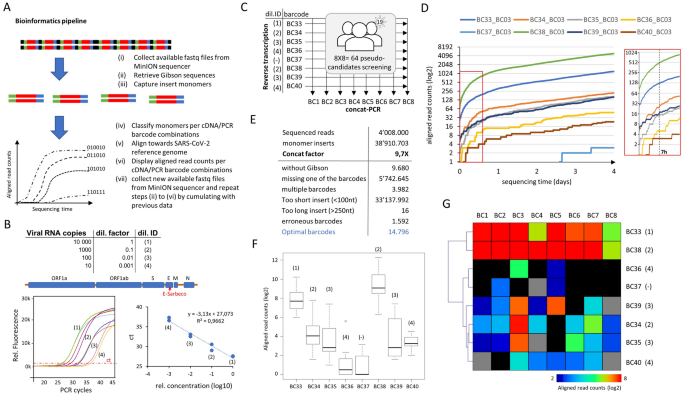
纳米孔DNA测序检测SARS-CoV-2诊断(一个) RETIVAD内生物信息学管道方案。在Gibson序列定位的帮助下,长串联序列读取在计算上分裂为单体。基于侧翼条形码(cDNA和PCR结合的条形码)对单体进行分层,并与Covid-19病毒参考基因组比对,以验证其特异性。考虑到牛津纳米孔仪器在测序过程中提供了测序数据,上述步骤是实时执行的,以便在MinION测序过程中评估和显示每个候选基因的对齐读取计数(由cDNA和PCR合并DNA条形码的组合定义)。(B合成SARS-CoV-2 RNA对照样品(Twist Bioscience;随后稀释武汉-胡-1株),针对E-Sarbeco病毒区进行反转录和RT-qPCR检测。(C)不同病毒RNA稀释度对应的8种条件的反转录试验方案(稀释度ID对应于(B)),目标是E-Sarbeco地区。每一种反转录检测都通过指定的分子条形码(BC33-BC40)进行鉴定。请注意,其中还包括一个没有病毒RNA的样本(BC37)。在实验结束时,将所有8个样本合并,并与其他8个针对E-Sarbeco基因(BC1-BC8)的条形码引物进行串联pcr扩增,提供对应64个伪候选基因的组合条形码策略。(D实时显示在纳米孔测序期间评估的每个双条形码组合(BC3与所有cDNA条形码)中与E-Sarbeco区域相关的对齐读取计数的数量。插图:放大视图,突出显示DNA测序的前七个小时足以显示样品之间的读取计数差异。(E) 4天DNA测序结束时评估的生物信息学管道总结。从检测到的单体数量与测序的串联体的初始数量之间的比率推断出串联因子。最佳条形码是指同时呈现cDNA和PCR条形码序列的最佳单体长度(> 100nt; < 250nt)序列的数量。(F箱形图显示了与每个cDNA条形码(BC33-BC40)和不同PCR条形码相关的总对齐读取计数。每个箱形图都标有相应的病毒RNA稀释度(dil)。在(B)).(G)热图显示与每个cDNA条形码(BC33-BC40)和不同PCR条形码(BC1-BC8)相关的总对齐读取计数。请注意(F,G)显示出100个病毒拷贝的灵敏度,因为下一个RNA稀释条件在某些情况下出现在阴性样品的相同水平。
总的来说,拟议的管道由标准的分子生物学策略组成,在纳米孔测序之前需要不到8小时的样品制备(图2)。2D和“方法”一节)。
对呈现不同数量病毒副本的多个伪候选病毒进行实时SARS-CoV-2诊断
基于大规模并行DNA测序的人口规模Covid-19诊断的主要瓶颈之一是提供诊断结果所需的时间。除了样品处理到测序文库制备所需的分子生物学工作流程外,在Illumina仪器上进行的DNA测序需要不可压缩的处理时间,这会延迟下游分析,因为它们只能在整个测序过程的最后进行。相反,Oxford Nanopore测序仪在滚动基础上提供测序读取(每个生成的fastq文件有4000个测序读取)。因此,我们开发了一个计算管道,专门用于(1)收集可用的fastq文件;(2)检索已测序分子中的Gibson适配器序列;(3)将其分解为单体;(4)评估其侧翼区域是否存在cDNA和PCR分子条形码;(5)将内部单体序列对准SARS-CoV-2参考基因组;(6)显示每个cDNA/PCR条形码组合的对齐读取计数。这个流水线被设置为在一个自动的时间间隔(例如每10分钟)重新启动,这样新的可用序列可以累积到以前的分析中,从而提供与cDNA/PCR条形码组合相关的对齐读取计数的实时视图(图2)。3.一个)。
为了验证所提出的诊断策略的性能,使用合成的SARS-CoV-2 RNA对照样品(Twist Biosciences)模拟每次检测的可变病毒RNA拷贝。具体而言,随后将合成的武汉- hu -1 (GeneBankID: MN908947.3) SARS-CoV-2 RNA对照样品稀释后进行反转录,然后对SARS-CoV-2 E基因上的一个区域进行定量PCR检测,该区域由前面描述的引物序列定义(E- sarbeco)。11。这种定量PCR可以检测到合成的SARS-CoV-2 RNA对照样品的10个病毒拷贝(图2)。3.B)。
对于基于纳米孔DNA测序的诊断,如图所示。2首先,我们设计了8个条形码E-Sarbeco反向引物(针对用于定量PCR检测的相同扩增子区域)和其他8个条形码E-Sarbeco正向引物(包含所需的Gibson序列),用于串联PCR扩增。这种组合策略允许模拟64个伪候选人的筛选(图2)。3.C)。
随后将合成的SARS-CoV-2 RNA对照样品稀释,用定义的条形码E-Sarbeco引物(BC33-BC36;BC38-BC40),此外还有一个不含病毒RNA物质的阴性对照样本(BC37)(稀释ID见图3)。3.C).实时诊断分析显示,基于E-Sarbeco靶区对齐读取计数的数量,呈现不同病毒RNA拷贝的样品之间存在显著差异。早在DNA测序后7小时就观察到这种差异,表明高病毒RNA滴度的样品在E-Sarbeco靶区呈现出系统性更高的对齐读取计数(图2)。3.D和图S2)。此外,经过4天的DNA测序(测序72小时,最后1天完成碱基召唤),总共测序了约400万reads,对应于约3800万个单体(9.7倍的串联因子)(图2)。3.E)。尽管在单体序列上获得了如此大的收益,但只有14000个最佳序列(即同时呈现cDNA和PCR切片条形码,且在预期的单体扩增子插入长度范围内)被用于诊断分析,所有其他序列要么插入太短(< 100nt),要么缺少一个或两个所需的条形码,这对鉴定样品至关重要。值得注意的是,对约14000个最佳序列的分析足以区分病毒RNA滴度在合成Covid-19 RNA材料的1至100倍稀释范围内的样品(图2)。3.F, G);相当于定量PCR检测到的100个拷贝(图2)。3.B)。事实上,在某些情况下,检测的灵敏度无法区分阴性对照样本(BC37)和病毒RNA滴度最低的样本(1/1000稀释;BC36)(无花果。3.F, G)。
SARS-CoV-2在多个伪候选病毒上的实时跟踪
考虑到当前对SARS-CoV-2变体的出现和在人群中的传播的跟踪需求,我们将上述SARS-CoV-2实时诊断策略演变为变体跟踪系统。由于目前描述的所有SARS-CoV-2变体在SPIKE基因中都存在大量突变,因此我们将实时跟踪系统的重点放在了该区域。事实上,通过集中对SPIKE基因(~ 3.8 kb)的测序工作,我们可以将诊断和变异跟踪结合到一个单一的分析中,这本身就代表了相对于当前策略的重大进展。
与上述针对短扩增子(E-Sarbeco)的诊断工作相反,对整个SPIKE基因的变异跟踪使用随机六聚体序列进行反转录步骤,并使用四个引物在不同区域靶向SPIKE基因(每个引物之间距离约1kb)(图2)。4A).与诊断试验的情况一样,用于逆转录的随机六聚体引物呈现出独特的分子条形码以及常见的吉布森序列。相反,PCR引物(P1-P4)具有独特的分子条形码,但缺乏吉布森序列。事实上,随机六聚体引物在cDNA上进行PCR扩增会导致不同长度的扩增子(包括片段> 1 kb;见图。4B &图S3),因此,在短放大策略上使用的串联步骤似乎不是必需的。为了简化其使用,PCR扩增是在针对SPIKE区域(P1-P4)的所有四种引物和Gibson引物序列(等摩尔浓度;即4 × Gibson引物对应1 ×每个SPIKE靶向引物)。
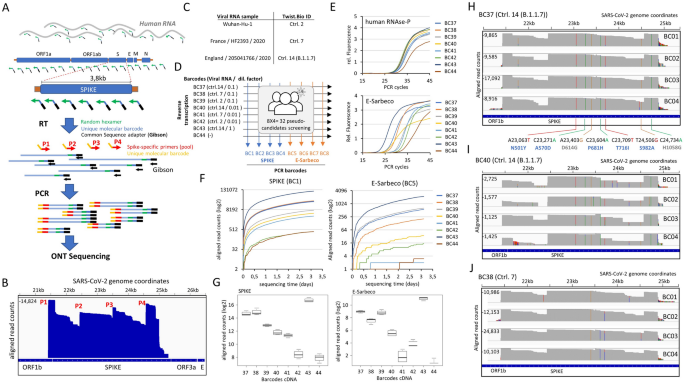
在SPIKE基因内追踪SARS-CoV-2变异。(一个)方案说明了使用牛津纳米孔技术(ONT)测序跟踪变异的SPIKE靶向策略。逆转录是执行随机六聚体引物呈现独特的分子条形码和吉布森适配器序列。这种方法可以产生覆盖整个病毒和人类基因组的可变长度的cDNA产物。条形码cDNA样本用四个spike特异性引物(每个引物包括一个共同的分子DNA条形码,间隔约1kb)和一个针对在逆转录过程中引入的适配器序列的Gibson引物进行PCR扩增。为了便于分析,所有四种SPIKE和Gibson引物都在等摩尔池中使用。PCR扩增的样品通过Oxford Nanopore MinION DNA测序进行分析。(B)基因组浏览器视图,显示ONT测序后获得的对齐读取计数。显示了每个SPIKE-targeting引物(P1-P4)的位置,并对应于SPIKE基因中呈现最高读取计数的区域。(C合成SARS-CoV-2病毒RNA样本汇总,对应武汉- hu -1对照样本(参考菌株)和两种描述变异菌株(HF2393和B.1.1.7)。(D) 7种条件下的反转录试验方案,对应于(C(BC37-BC43)和不含病毒RNA的对照样本(BC44)。在所有情况下,也引入了15 ng的人类RNA材料-从培养的细胞系中获得。所有8个样本在反转录实验结束时合并,用4个针对SPIKE基因(BC1-BC4)的条形码引物池或4个针对E-Sarbeco区域(BC5-BC8)的条形码引物进行PCR扩增,提供32个伪候选物的组合条形码策略。(E)针对人类RNAse-P基因或病毒E-Sarbeco区域的定量PCR,通过(D)。(F)通过RETIVAD进行实时Covid-19诊断,显示每个条形码cDNA样本的对齐读取计数(见(D)),用于基因SPIKE (BC1)或E-Sarbeco区域(BC5)。(G)箱形图显示了与基因SPIKE或E-Sarbeco区相关的每一种情况下(D)。(H)用RETIVAD对与相应PCR条形码(BC1-BC4)相关的条形码样本37 (BC37)进行变异分析。显示的基因组浏览器视图显示了多个核苷酸变体(彩色条)的存在,其中七个在所有四个样本(BC1-BC4)中检索到,并且具有显著的读取覆盖率。其中5架(A23,063T;C23,271A;C23,604A;C23,709T;T24,506G)对应于B.1.1.7 UK变异株(蓝色)中描述的错义突变。此外,还检测到另外两种错义突变;D614G(以前在SARS-CoV-2欧洲毒株中描述)和H1058G。(我)与(H)相似,但用于样品BC40,对应的RNA滴度比样品BC37低10倍。请注意,在(H)中可以观察到相同的错义突变(彩色条),尽管读取计数较低。(J)对BC38样本进行变异检测,对应于2020年在法国描述的SARS-CoV-2变异株HF2393。在这种情况下,在所有四个样本(BC1-BC4)中只检索到错义突变D614G。
为了更好地模拟真实的情况,诊断/变异跟踪分析是用从人类候选样本(例如鼻咽拭子)中收集的样本进行的,从培养细胞系中产生的人类RNA与合成病毒RNA样本结合在一起,这样用于逆转录分析的随机六聚体引物也可以为样本收集提供阳性对照计数的手段。此外,为了解决变体跟踪性能问题,除了武汉- hu -1 SARS-CoV-2 RNA对照样本外,我们还使用了与高传染性SARS-CoV-2变体B.1.1.7相对应的合成病毒RNA样本,该变体于2020年12月首次在英国被描述,以及自2020年1月以来在法国传播的特定进化支HF2393菌株12(无花果。4C)。
为了对多个伪候选病毒进行诊断和变异跟踪分析,我们使用了8种条形码随机六聚体引物,用于不同病毒RNA滴度(连续稀释)下的逆转录(BC37-BC44),并从上述合成病毒RNA菌株中发布(图2)。4为了增加伪候选基因的数量,我们用4个条形码引物扩增了8个条形码cDNA产物,分别针对SPIKE基因(BC1-BC4对应P1-P4库)或E-Sarbeco区(BC5-BC8)。
从条形码随机六聚体引物中获得的cDNA产物首先通过定量PCR扩增进行验证,目标是人类RNAse-P基因或病毒E-Sarbeco区域(图2)。4E)。然后,将针对SPIKE (BC1-BC4)和针对E- sarbeco区域(BC5-BC8)的条形码引物生成的PCR产物汇总在一起,进行ONT测序分析。对SPIKE基因或E-Sarbeco区域的实时诊断分析显示,基于对齐读取计数的数量,呈现不同病毒RNA拷贝的样品之间存在显著差异。具体来说,阴性对照样本(BC44)的SPIKE和E-Sarbeco区排列计数系统最少,相反,病毒RNA滴度最集中的样本(BC43)的上述靶区排列读数最多(图4)。4F, G)。
除了诊断结果外,RETIVAD还执行相对于SARS-CoV-2参考基因组的变异检测。事实上,如图2所示。4H, RETIVAD在BC37条形码cDNA (BC1-BC4 × BC37)的所有4个样本中检测到7个显著的SPIKE基因变异。其中有5个对应于之前描述的B.1.1.7菌株(N501Y、A570D、P681H、T716I和S982A);这与BC37相关样本是用与该SARS-CoV-2变体对应的合成病毒RNA生成的事实相一致(图2)。4同样,对来自BC40条形码cDNA (BC1-BC4 x BC40)的样本进行的变异检测显示出类似的特征,尽管它们的覆盖率较低(图2)。4事实上,与BC40相关的样品也是用B.1.1.7株对应的合成病毒RNA生成的,但其滴度比BC37低10倍(图1)。4D)。
最后,对BC38条形码cDNA (BC1-BC4 x BC38;法国菌株HF2393)发现了一个单一的共同突变,D614G,以前在法国菌株HF2393中描述过12在BC37和BC40的样本中检测到的变异中也观察到其他欧洲菌株,包括B.1.1.7。4H, I)。
讨论
尽管目前开展了疫苗接种工作,但大流行形势预计最近描述的变体会传播,也会出现新的变体,这主要是由于各国在获得最佳疫苗接种规划方面存在重大差异6。因此,使用Covid-19诊断方法进行计数以及在人口规模上跟踪变异仍然至关重要。
目前,诊断和变异跟踪分两步进行,这本身就延迟了获得完整诊断的时间。此外,变异的追踪依赖于SARS-CoV-2的全基因组测序,这是一项昂贵且劳动密集型的工作,与群体规模的测定不相容。最后,大多数变异的追踪工作依赖于使用大型和昂贵的测序仪器,需要将样本处理集中到专门的机构,而这在贫穷国家是相当缺乏的。
为了解决所有这些缺陷,我们在此提出了一种能够(1)同时进行SARS-CoV-2诊断和变体跟踪的方法;(2)通过使用实时跟踪系统缩短获取诊断/变异检测结果的时间;(3)充分利用小巧实惠的Oxford Nanopore MinION DNA测序仪,特别是在世界各地部署上述诊断和变体跟踪平台。
在这项研究中,我们通过在总共64个伪候选样本上靶向SARS-Cov-2 E基因(E- sarbeco)上的一个区域,验证了所提出的基于纳米孔DNA测序的诊断方法。3.)。重要的是,执行该诊断试验所需的最佳序列数量很少,这表明在MinION测序仪提供的测序深度范围内,可以进行更大的候选筛选(数百个)。
拟议的变体跟踪侧重于分析SPIKE基因,而不是整个SARS-CoV-2基因组。事实上,SPIKE基因集中了目前发现的大多数变异,这可以通过它编码与人类细胞相互作用所需的蛋白质这一事实来解释。值得注意的是,目前大多数针对Covid-19的疫苗也是基于SPIKE区域。因此,集中对该基因的变异跟踪似乎是增加候选基因数量的最佳折衷方案。最近的一项描述HiSpike方法的工作分享了这一理念,该方法在小型Illumina MiSeq仪器的帮助下,在SPIKE基因内进行变异检测13。Illumina仪器是一种短片段测序方法,HiSpike需要42对引物产生400个核苷酸长度的扩增子来覆盖SPIKE区域。相反,我们提出的策略,基于长DNA片段的使用,与纳米孔DNA测序技术兼容,依赖于4个针对SPIKE区域的引物,一个cDNA步骤的随机引物和一个共同的吉布森序列。事实上,专门用于在世界任何地方部署的SARS-CoV-2诊断和变体监测战略不仅需要使用低成本仪器,而且还需要减少试剂的数量,以便长期使用。
在本文的最终修订期间,来自B.1.1.7谱系的新变体获得了主要关注,特别是由于它们在英国,美国和其他地方广泛传播(B.1.617.1,也称为Kappa;B.1.617.2也被称为“Delta”和B.1.618也被称为“三重突变体”,怀疑是从B.1.617进化而来的)14。在所有病例中,都观察到了SPIKE基因上的新突变,进一步支持了我们针对该基因组区域进行变异监测的策略。
由于所有这些原因,我们相信我们提出的策略,加上专用的计算解决方案RETIVAD,可以代表一种改变游戏规则的方法,在接下来的几个月里追踪变异的进展。
方法
SARS-CoV-2合成RNA
本研究使用了以下合成SARS-CoV-2 RNA对照样品,以每微升100万拷贝的浓度递送:
wu - hu -1 (GeneBankID: MN908947.3);Biosc。ID: Ctrl2。
法国/ HF2393/2020 (EPI_ISL_418227);Biosc。ID: Ctrl7。
英格兰/ 205041766/2020 (EPI_ISL_710528);Biosc。ID: Ctrl14 (B.1.1.7)。
条形码引物设计
用于反转录的引物由(1)Gibson序列片段(redGibs: GAAGAACCTGTAGATAACTCGCTGT), (2) ONT PCR条形码扩增1 - 96试剂盒(EXP-PBC096)发出的条形码序列和(3)目标序列组成。针对SARS-CoV-2基因组内的E-Sarbeco区,Corman等人提供的反向引物序列。11,已被使用:
E_Sarbeco_R: ATATTGCAGCAGTACGCACACA
对于以spike为中心的变异追踪,目标序列已被随机的六聚体取代,特别是为了减少测定过程中所需引物的数量。
利用以下针对E-Sarbeco区的条形码引物进行逆转录:
redGibsBC33_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTCAGACTTGGTACGGTTGGGTAACTATATTGCAGCAGTACGCACACA
redGibsBC34_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTGGACGAAGAACTCAAGTCAAAGGCATATTGCAGCAGTACGCACACA
redGibsBC35_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTCTACTTACGAAGCTGAGGGACTGCATATTGCAGCAGTACGCACACA
redGibsBC36_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTATGTCCCAGTTAGAGGAGGAAACAATATTGCAGCAGTACGCACACA
redGibsBC37_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTGCTTGCGATTGATGCTTAGTATCAATATTGCAGCAGTACGCACACA
redGibsBC38_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTACCACAGGAGGACGATACAGAGAAATATTGCAGCAGTACGCACACA
redGibsBC39_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTCCACAGTGTCAACTAGAGCCTCTCATATTGCAGCAGTACGCACACA
redGibsBC40_E_Sarbeco_R: GAAGAACCTGTAGATAACTCGCTGTTAGTTTGGATGACCAAGGATAGCCATATTGCAGCAGTACGCACACA
使用以下条形码随机六聚体引物进行逆转录:
redGibsBC37_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTGCTTGCGATTGATGCTTAGTATCANNNNNN
redGibsBC38_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTACCACAGGAGGACGATACAGAGAANNNNNN
redGibsBC39_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTCCACAGTGTCAACTAGAGCCTCTCNNNNNN
redGibsBC40_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTTAGTTTGGATGACCAAGGATAGCCNNNNNN
redGibsBC41_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTGGAGTTCGTCCAGAGAAGTACACGNNNNNN
redGibsBC42_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTCTACGTGTAAGGCATACCTGCCAGNNNNNN
redGibsBC43_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTCTTTCGTTGTTGACTCGACGGTAGNNNNNN
redGibsBC44_Rhexamer_R: GAAGAACCTGTAGATAACTCGCTGTAGTAGAAAGGGTTCCTTCCCACTCNNNNNN
为了扩增,设计第二组条形码引物,其结构如下:(1)反向互补的Gibson片段(revGibs: ACAGCGAGTTATCTACAGGTTCTTCAATGT), (2) ONT PCR条形码扩增1 - 96试剂盒(EXP-PBC096)产生的条形码序列,(3)目标序列。针对SARS-CoV-2基因组内的E-Sarbeco区,Corman等人提供的正向引物序列。11,已被使用:
E_Sarbeco_F: ACAGGTACGTTAATAGTTAATAGCGT
本研究使用了以下针对E-Sarbeco区的条形码引物:
revGibs -_BC01_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTAAGAAAGTTGTCGGTGTCTTTGTGACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC02_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTTCGATTCCGTTTGTAGTCGTCTGTACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC03_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTGAGTCTTGTGTCCCAGTTACCAGGACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC04_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTTTCGGATTCTATCGTGTTTCCCTAACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC05_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTCTTGTCCAGGGTTTGTGTAACCTTACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC06_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTTTCTCGCAAAGGCAGAAAGTAGTCACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC07_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTGTGTTACCGTGGGAATGAATCCTTACAGGTACGTTAATAGTTAATAGCGT
revGibs -_BC08_E_Sarbeco_F: ACAGCGAGTTATCTACAGGTTCTTCAATGTTTCAGGGAACAAACCAAGTTACGTACAGGTACGTTAATAGTTAATAGCGT
对于以钉为中心的变异追踪,反向互补的吉布森序列被排除在外,特别是因为在随机六聚体生成的cDNA模板上进行的PCR扩增产生了大片段(> 1kb)。相反,E-Sarbeco靶向寡核苷酸产生的扩增子产物产生与定量PCR分析兼容的短扩增子,因此需要在纳米孔测序之前进行串联。
以覆盖SPIKE基因(~ 3.8 kb)为目标,设计了以下4条引物,以间隔~ 1 kb的SPIKE区域为目标:
Spike1_F: AGGGGTACTGCTGTTATGTCT
Spike2_F: TGCACTTGACCCTCTCTCAG
Spike3_F: GCAGGCTGTTTAATAGGGGC
Spike4_F: TGCAGACATATGTGACTCAACA
本研究使用的Spike靶向引物包括相应的条形码序列如下:
BC01_Spike_1_F: AAGAAAGTTGTCGGTGTCTTTGTGAGGGGTACTGCTGTTATGTCT
BC02_Spike_1_F: TCGATTCCGTTTGTAGTCGTCTGTAGGGGTACTGCTGTTATGTCT
BC03_Spike_1_F: GAGTCTTGTGTCCCAGTTACCAGGAGGGGTACTGCTGTTATGTCT
BC04_Spike_1_F: TTCGGATTCTATCGTGTTTCCCTAAGGGGTACTGCTGTTATGTCT
BC01_Spike_2_F: AAGAAAGTTGTCGGTGTCTTTGTGTGCACTTGACCCTCTCTCAG
BC02_Spike_2_F: TCGATTCCGTTTGTAGTCGTCTGTTGCACTTGACCCTCTCTCAG
BC03_Spike_2_F: GAGTCTTGTGTCCCAGTTACCAGGTGCACTTGACCCTCTCTCAG
BC04_Spike_2_F: TTCGGATTCTATCGTGTTTCCCTATGCACTTGACCCTCTCTCAG
BC01_Spike_3_F: AAGAAAGTTGTCGGTGTCTTTGTGGCAGGCTGTTTAATAGGGGC
BC02_Spike_3_F: TCGATTCCGTTTGTAGTCGTCTGTGCAGGCTGTTTAATAGGGGC
BC03_Spike_3_F: GAGTCTTGTGTCCCAGTTACCAGGGCAGGCTGTTTAATAGGGGC
BC04_Spike_3_F: TTCGGATTCTATCGTGTTTCCCTAGCAGGCTGTTTAATAGGGGC
BC01_Spike_4_F: AAGAAAGTTGTCGGTGTCTTTGTGTGCAGACATATGTGACTCAACA
BC02_Spike_4_F: TCGATTCCGTTTGTAGTCGTCTGTTGCAGACATATGTGACTCAACA
BC03_Spike_4_F: GAGTCTTGTGTCCCAGTTACCAGGTGCAGACATATGTGACTCAACA
BC04_Spike_4_F: TTCGGATTCTATCGTGTTTCCCTATGCAGACATATGTGACTCAACA
反转录
使用SuperScript™IV Reverse Transcriptase (Thermofisher Scientific;18090200;0.5ul上标IV (200U/ul)),标准反应量为20ul,其中包括0.5ul超级RNAseIn (Thermofisher Scientific;AM2694;20U/ul), 1ul DTT (0.1 M), 4 ul SSIV缓冲液(5X), 0.5 ul dNTP(各10 mM), 1ul合成SARS-CoV-2 RNA, 1ul 2uM基因特异性条形码寡核苷酸(例如E-Sarbeco条形码bc33-bc48;0.1uM终浓度)或1 ul 50uM随机六聚体条形码寡核苷酸(终浓度:2.5uM)。检测包括随机六聚体条形码引物,1ul人RNA (15 ng/ul;从培养细胞系中收集的)与合成的SARS-CoV-2 RNA一起加入实验,并事先加入随机六聚体。
合成的SARS-CoV-2 RNA在每个反应中分别以100万份拷贝(稀释系数,d.f: 1)、10万份拷贝(d.f: 0.1)、1万份拷贝(d.f: 0.01)和1万份拷贝(d.f: 0.001)加入。值得注意的是,这些连续稀释后,下游稀释后的拷贝数分别为1万、1千、100和10,通过定量PCR分析(图2)。3.B)。
在50°C条件下孵育30分钟,然后在80°C条件下孵育10分钟。将条形码cDNA样本混合(一次8个样本),并用SPRIselect试剂(Beckman;B23318),比为0.5倍。在80ul(初始清洗体积的一半)上回收清洗过的cDNA;即2倍浓度)。收集并清洗的cDNA 40ul用于PCR扩增(5ul × 8个PCR反应;见下文),而剩余的材料用于定量PCR评价。
SARS-CoV-2定量PCR验证
条形码cdna材料已通过以下引物序列进行定量PCR验证:
针对SARS-Cov-2 E基因区域的引物(退火温度:58℃)
E_Sarbeco_F:ACAGGTACGTTAATAGTTAATAGCGT
E_Sarbeco_R:ATATTGCAGCAGTACGCACACA
引物针对人类基因RnaseP(退火温度:60℃):
Human_RNASE_P-F: AGATTTGGACCTGCGAGCG
Human_RNASE_P-R: GAGCGGCTGTCTCCACAAGT
定量PCR检测采用定量SYBR绿色PCR试剂盒(编号:204,145),在1/10稀释的cDNA材料上进行。
串联PCR扩增和纳米孔测序
最终体积为25ul,包括12.5ul的Phusion Hot Start II High-Fidelity PCR Master Mix (ThermoFisher ref: F565), 2.5ul的1uM条形码-E-Sarbeco靶向引物(BC1-8), 2.5ul的1uM短Gibson引物(AGAACCTGTAGATAACTCGCTGT)和5ul汇集和清洁的cDNA。
对于短扩增子E-Sarbeco PCR扩增,使用以下热循环程序:
- 1.
98°C 30 s
- 2.
98°C 10 s
- 3.
61°C 20 s
- 4.
72°C 30 s
- 5.
重复2-4步25次
- 6.
98°C 10 s
- 7.
61°C 20 s
- 8.
72°C 2分钟
- 9.
重复6-8步20次
- 10.
72°C 10分钟
- 11.
保持在12°C
为了在随机六聚体生成的cDNA材料上扩增针对SPIKE和E-Sarbeco的PCR,使用一组带条形码的SPIKE引物和短gibson进行检测。将100ul条形码引物Spike1 (10uM) 10ul, Spike2 (10uM) 10ul, Spike (10uM) 10ul, Spike4 (10uM) 10ul, ShortGibson (10uM) 40ul与蒸馏水混合。然后在25ul的终体积上进行PCR扩增,其中包括12.5ul Phusion Hot Start II High-Fidelity PCR Master Mix (ThermoFisher ref: F565), 1.25ul汇集的条形码-SPIKE和shortGibson引物(每个SPIKE目标引物的终浓度为0.05uM, shortGibson的终浓度为0.2uM)和5ul汇集并清洗的cDNA。
使用以下热循环程序:
- 1.
98°C 30 s
- 2.
98°C 10 s
- 3.
61°C 45 s
- 4.
72°C 2分钟
- 5.
重复2-4步39次
- 6.
72°C 10分钟
- 7.
保持在12°C
在这两种情况下(E-Sarbeco串联PCR或SPIKE PCR扩增),PCR产物通过TapeStation自动电泳(Genomic DNA ScreenTape;Agilent编号:5067-5365)。
在不同条形码引物的存在下进行多次PCR分析,这样与条形码cDNA材料的结合可以产生很大的复杂性。具体来说,对于E-Sarbeco靶向检测,将8个条形码接触式pcr检测合并(8 × 25 = 200ul),并用SPRIselect试剂(Beckman;B23318),比为0.5×。同样,对于随机六聚体cDNA材料生成的SPIKE和E-Sarbeco PCR检测,每个靶区有4个条形码PCR检测(SPIKE: BC1-4;E-Sarbeco: BC5-8)收集(4 × 2 × 25 = 200ul),用SPRIselect试剂(Beckman;B23318),比为0.5倍。
汇集和清洁的材料在50ul上回收,并用于纳米孔测序文库制备(纳米孔连接测序试剂盒:SQK-LSK109)。使用MinION Mk1C仪器在纳米孔FLO-MIN106D流式细胞(R9)上对DNA文库进行测序(测序72 h;高精度基数呼叫)。
实时SARS-CoV-2诊断和变体跟踪
Mk1C仪器在测序过程中生成的fastq文件的实时处理使用我们内部开发的RETIVAD(实时变异检测器)软件进行。RETIVAD每十分钟从Mk1C仪器收集测序过程中产生的fastq文件,并(1)在测序reads中搜索Gibson序列(regex查询(https://pypi.org/project/regex)默认最多接受4个不匹配);(2)单体上的分裂序列,以鉴定的Gibson序列的存在为界;(3)查找逆转录过程中引入的条形码序列(cDNA条形码:BC33-BC48;正则表达式查询);(4)查找PCR扩增过程中引入的条形码(PCR条形码:BC1-BC8;正则表达式查询);(5)收集cDNA与pcr相关条形码之间的序列,与SARS-CoV-2参考基因组(Wuhan-Hu-1基因组:NC_045512;BWA对准器遵循纳米孔参数)。考虑到步骤(3-5)是在步骤(2)中检索到的单体中执行的,RETIVAD将cDNA/PCR条形码组合与对齐的结果进行匹配,从而为相关的伪候选物提供诊断结果。由于在10分钟的间隔内检索到的序列数量较少,并且SARS-CoV-2基因组的大小相对较短(~ 29 kb),因此RETIVAD能够在10分钟的间隔内提供这样的诊断结果。 Hence, at the next round of data collection, RETIVAD reiterates on the aforementioned steps over the newly collected fastq files and appends the outcome to the previous results. The continuous gain on aligned read-counts per region of interest (e.g. E-Sarbeco amplicon sequence, or SPIKE gene; defined as entry parameters) is visualized within scatterplot view (png file) updated during the processing (RETIVAD produces a png file per detected PCR barcode monomers; thus allowing to generate scatterplots displaying as many lines as detected cDNA barcode monomers; FigureS4)。
除了诊断结果,RETIVAD还被设计用于执行实时变异检测。为此,雷达信标包(版本1.4.20)用于原始的从头组装DNA,然后与Minimap2(版本2.17)。青鳉(版本1.2.5)用于从对齐的数据创建一致性序列和变量调用(变量调用默认需要> 20对齐读取)。RETIVAD生成每个cDNA/PCR条形码组合的BAM文件,其中包含碱基转换信息及其相关的覆盖范围(用于基因组浏览器的可视化,如进口)。此外,还生成了每个cDNA/PCR条形码组合的摘要文件,提供了检测到碱基转换的基因组位置,转换碱基的身份及其相关覆盖范围。与诊断处理的情况一样,变异检测是实时执行的,从而为用户提供了将结果可视化的可能性,并决定是否需要继续进行测序,或者收集的信息是否足以为相应的候选者完成诊断/变异跟踪。
如果在最后一次数据收集和处理40分钟后没有新的fastq文件可用,RETIVAD将为诊断结果生成最终摘要报告。
数据可用性
本研究中包含的所有基因组序列可根据要求提供。RETIVAD是免费的https://github.com/SysFate/retivad。
参考文献
范登堡,O.,马丁尼,D.,罗查斯,O.,范·贝尔库姆,A.和科兹拉基迪斯,Z.诊断性COVID-19测试的考虑。Nat Rev. Microbiol。19, 171-183(2021)。
La Marca, A。et al。SARS-CoV-2 (COVID-19)检测:分子和血清学体外诊断方法的系统综述和临床指南天线转换开关。生物医学。在线41中文信息学报,483-499(2020)。
Palmieri, D., Siddiqui, J., Gardner, A., Fishel, R. & Miles, W. O. REMBRANDT:一种用于COVID-19筛查的高通量条形码测序方法。bioRxivhttps://doi.org/10.1101/2020.05.16.099747(2020)。
Schmid-Burgk, j.l.。et al。LAMP-Seq:使用组合条形码进行人群规模的COVID-19诊断。bioRxivhttps://doi.org/10.1101/2020.04.06.025635(2020)。
布鲁姆,j.s.。et al。拭子- seq:大规模大规模SARS-CoV-2检测的高通量平台。MedRxivhttps://doi.org/10.1101/2020.08.04.20167874(2021)。
了解SARS-CoV-2的变体。《柳叶刀》397, 462(2021)。
Runtuwene, L. R., Tuda, J. S. B., Mongan, A. E.和Suzuki, Y.现场MinION测序。在单分子和单细胞测序(主编:Suzuki, Y.) 143-150 (b施普林格,2019)。https://doi.org/10.1007/978-981-13-6037-4_10。
Charre C。et al。基于ngs的SARS-CoV-2全基因组鉴定方法的评价病毒的另一个星球。6, 2(2020)。
詹姆斯,P。et al。LamPORE:基于纳米孔测序的SARS-CoV-2感染快速、准确和高度可扩展的分子筛选。MedRxivhttps://doi.org/10.1101/2020.08.07.20161737(2020)。
Nakamichi, K。et al。与COVID-19中SARS-CoV-2病毒分支相关的住院和死亡率。科学。代表。11, 4802(2021)。
科曼,v.m.。et al。实时RT-PCR检测2019新型冠状病毒(2019- ncov)欧元的测量员25, 2(2020)。
Gambaro F。et al。2020年1月24日至3月23日,法国SARS-CoV-2的引入和早期传播。欧元的测量员25, 2(2020)。
法斯,E。et al。HiSpike:一种高通量高性价比的SARS-CoV-2刺突基因测序方法。medRxivhttps://doi.org/10.1101/2021.03.02.21252290(2021)。
Sanyaolu,。et al。新出现的SARS-CoV-2变体令人担忧。其他。放置感染。说。8, 20499361211024372(2021)。
致谢
我们非常感谢Corinne Cruaud和她的团队在Genoscope,为牛津纳米孔测序技术培训。我们也感谢Veronique de Berardinis和Patrick Wincker为这个项目提供的后勤支持。
资金
这项工作得到了来自法兰西岛r
作者信息
作者及单位
贡献
概念化:M.A.M.P.方法论:J.L.P.和M.A.M.P.软件开发:F.S.和S.E.科学评价:M.A.M.P.写作、评审和编辑:J.L.P.、F.S.和M.A.M.P.资金获取:M.A.M.P.
相应的作者
道德声明
相互竞争的利益
作者声明没有利益冲突。
额外的信息
出版商的注意
b施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。
关于本文
引用本文
st
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-021-95563-w
这篇文章是由
临床实验室高通量SARS-CoV-2全基因组测序工作流程的开发和验证
科学报告(2022)