摘要
2019冠状病毒病(COVID-19)正在全球肆虐。这种可能致命的传染病是由严重急性呼吸综合征冠状病毒2 (SARS-CoV-2)引起的。然而,COVID-19的完整机制尚不清楚。因此,我们分析了COVID-19患者的基因表达谱,通过一种创新的机器学习方法来识别疾病相关基因,该方法支持数据驱动策略,从少量样本和许多候选数据集中进行基因选择。采用基于主成分分析的无监督特征提取(PCAUFE)对16例COVID-19患者和18例健康对照者的RNA表达谱进行分析。结果从60683个候选探针中确定了123个对COVID-19进展至关重要的基因,包括免疫相关基因。这123个基因富集在转录因子NFKB1和RELA的结合位点上,这些转录因子参与多种生物现象,如免疫应答和细胞存活:典型核因子- κ B (NF-)的主要中介κB)活性是异源二聚体RelA-p50。这些基因在组蛋白修饰H3K36me3中也富集,且与NFKB1和RELA的靶基因大量重叠。我们发现在COVID-19患者中重叠基因下调。这些结果表明,典型的NF-κH3K36me3抑制了COVID-19患者血液中的B活性。
简介
2019冠状病毒病(COVID-19)是由严重急性呼吸综合征冠状病毒2型(SARS-CoV-2)引起的传染病。它于2019年12月在中国湖北武汉首次被发现,并导致了持续的大流行1,2,3..COVID-19是一种潜在的人畜共患疾病,死亡率中等(2-5%),主要通过飞沫和与感染者或潜伏期携带者的直接接触传播4.大量轻症和无症状病例被认为是SARS-CoV-2的一个特征5,6,7.然而,它会严重影响肺部,COVID-19幸存者可能会遭受长期健康影响。虽然对COVID-19进行了大量研究,但我们对它的认识还远远不够全面。目前没有明确有效的COVID-19预防或治疗方法。COVID-19患者别无选择,只能接受支持性护理以缓解症状8.因此,阐明COVID-19的发病机制,寻找有效的治疗方法是当务之急。
“银弹”方法需要分析包含从样本中提取的RNA的RNA- seq数据。通过比较COVID-19患者与非患者的基因表达,我们可以获得更多关于传染病病理的信息。使用机器学习的数据驱动方法是预测机制的有效策略,这些机制难以通过生物学中传统的基于知识的分析应用来阐明。虽然获取COVID-19的各种组学数据并不困难,但由于这些数据往往包含数万个候选基因,样本很少,因此分析起来很困难。
近年来,基于主成分分析(PCA)的无监督特征提取方法被提出用于基因选择。这种方法被称为PCAUFE9,10,11,12,13,14,15,16,17,18,19,20.,21,22,23,24,25,26,27,可以分析具有少量样本和许多变量的数据集。该算法基于线性代数,计算量小,已被证实能很好地解决各种基因选择问题。例如,与创伤后应激障碍(PTSD)介导的心脏病相关的mRNA/miRNA表达的综合分析17以及各种癌症12发现了一个可能与这些疾病相关的候选基因。最近,通过使用PCAUFE对登革出血热患者血液进行整合基因表达分析,确定了46个对疾病进展至关重要的基因,而其他生物信息学分析方法无法获得这样的结果27.此外,对PCAUFE方法的理论论证已经在以前的研究中得到了发展(更多细节,请参见27).
在本文中,我们通过将PCAUFE应用于COVID-19患者和健康对照组的RNA表达谱,鉴定了与COVID-19相关的基因。数字1展示了我们的研究大纲。我们从生物学和机器学习的角度证实了所识别基因的可靠性。此外,我们使用生物信息学工具来识别调控上层选择基因的转录因子和组蛋白修饰。本文的新颖之处在于,与以下部分描述的其他典型基因选择方法相比,PCAUFE应用于基因表达谱提供了最少数量的基因,这些基因可以从机器学习和生物学角度合理解释COVID-19的发展。
结果
PCAUFE在COVID-19患者基因表达中的应用
本节描述了我们如何使用PCAUFE分析多个COVID-19患者的基因表达模式。
第一个例子(数据集1,GSE152418)由**am等人获得28.分为重症监护患者(IP)、重症患者(SP)、中度患者(MP)、恢复期患者(CP)和健康对照组(HC) 5类。通过对IP + SP + MP组(16例患者)与CP + HC组(18例非患者)的主成分(PC)负荷进行统计分析,我们发现了第二和第三个PC (PC2和PC3)。的P使用t-test拒绝了IP+SP+MP组内和CP+HC组内的平均负荷相同的零假设:9.69 × 10−5PC2和pc3.67 × 10−3.生物。虽然患者与非患者之间的PC1负荷有显著差异,但患者与非患者之间的PC1负荷有显著差异P值(1.83 × 10−2)的大小比pc2和pc3的大。如图S1所示,第2和第3 PCs比第1 PCs更清楚地将样本分为患者和非患者。这就是为什么我们选择了pc 2和pc 3,而不是pc 1。在这个平面上,我们选择嵌入PC分数的141个探针作为异常值,根据aχ2使用P由Benjamini和Hochberg (BH)准则调整的值29.表格1列出与141个探针相关的123个基因。
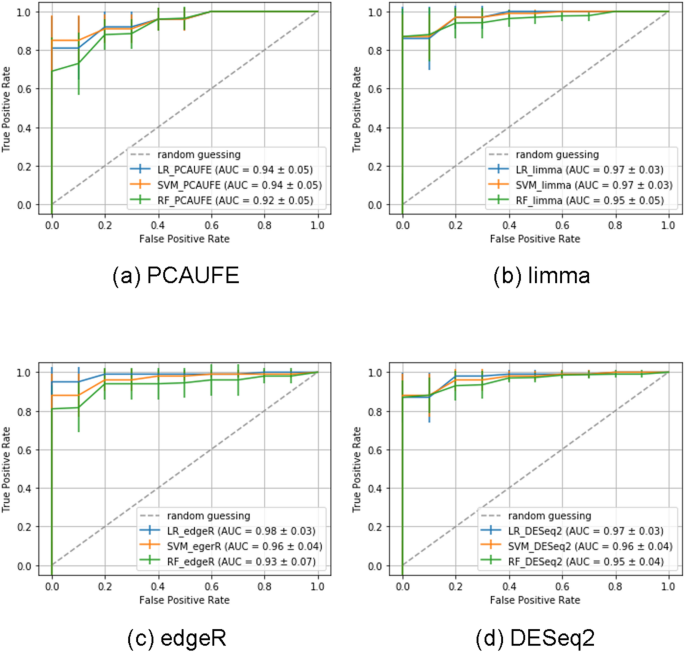
为了确认我们成功地选择了代表样本之间关系的关键基因,我们仅从PCAUFE选择的123个基因中构建了预测COVID-19患者的模型。我们使用数据集2,其中包括100名COVID-19患者和26名非COVID-19患者,来计算曲线下面积(AUC)。30..我们使用逻辑回归(LR)31,支持向量机(SVM)32,33,随机森林(RF)34作为分类模型。表格S1显示三个模型的每个超参数。我们通过对三个分类器的数据集2的样本进行随机洗牌来进行5次交叉验证。数字2(a)为受试者工作特征(ROC)曲线35,36每种模型。如图所示,每个模型的平均AUC都在0.9以上。从这些结果中,我们可以使用123个选定的基因来预测COVID-19的结果。
与其他基因选择方法的比较
为了证实我们结果的稳健性,我们用另外两种经典方法进行了基因选择:微阵列显著性分析(SAM)37微阵列数据线性模型(LIMMA)38.通过将SAM和LIMMA应用于数据集1 (GSE152418),我们鉴定了与调整相关的基因P-值小于0.01。我们证实PCAUFE选择的大部分基因都包含在SAM和LIMMA选择的基因中。每次应用SAM,所选择的基因都会发生变化。因此,SAM的结果没有在本报告中显示。对于LIMMA,所选择的探针包含了PCAUFE所选择的所有基因。值得注意的是,PCAUFE可以将候选基因限制在比普通基因表达分析工具更少的数量;例如,LIMMA选择了18458个探针。通过进一步将18458个探针从最小调整到几乎与PCAUFE相同的数量P-值,我们还使用数据集2对LIMMA选择的基因进行了患者/非患者分类。使用LR、SVM和RF的结果如图所示。2(b).我们确认各模型的分类性能与PCAUFE的分类性能相当。
为了增加我们结果的稳健性,我们还选择了使用一些最近的R包的基因:edgeR39和DESeq240.与SAM和LIMMA一样,通过对数据集1应用edgeR和DESeq2,我们识别了与调整相关的基因P-值小于0.01。edgeR和DESeq2选择的探针数分别为4452和5696。因此,这些方法比PCAUFE选择了更多的基因。edgeR和DESeq2筛选的基因分别包含PCAUFE筛选的59个和64个基因。3种方法共筛选出57个共同基因。为了进一步与PCAUFE进行比较,我们根据edgeR和DEseq2选择的基因进行分类分析,预测样本是否为COVID-19患者。在分类分析中,我们限制edgeR和DEseq2各自选择的探针数量相同,并调整最小的探针数量P- PCAUFE中的值(即141),因为两种方法都选择了太多的基因,无法将其用作预测模型的解释变量(即4452和5696)。edgeR和DEseq2的相关基因数分别为111个和113个。数据2(c)和(d)显示了使用有限探针的患者预测模型的ROC曲线。他们的auc与PCAUFE的auc大致相等。如前所述,我们发现PCAUFE选择的基因数量比其他方法更少,对于预测COVID-19患者是否显著。
作为本小节的最后一部分,我们进行了加权基因共表达网络分析(WGCNA)。采用的分析程序41而且42,我们应用了WGCNA R包43到数据集1。我们在这里选择了幂β= 7作为构建无标度网络的软阈值。S2(A))。我们得到共表达网络中的99个模块,如图所示。S2(B).这些模块包括18882个探针,远远多于中金财经选择的141个探针。此外,PCAUFE选择的基因中有近一半(即58个)包含在WGCNA的基因中。作为参考,由前3个模块中最小的基因组成的蛋白-蛋白相互作用(PPI)网络P这些基因的值和富集分析结果也显示在补充图中。S3和表S2,分别。
如前所述,我们验证了我们的方法,采用PCAUFE进行基因选择,比WGCNA、edgeR和DEseq2等普通方法更有效地缩小候选基因的范围。
讨论
在这项研究中,我们首先使用PCAUFE鉴定了123个与COVID-19患者相关的基因。我们通过以下补充分析来证明线性降维方法的使用:为了与PCAUFE进行比较,我们还应用了线性降维方法t-分布式随机邻域嵌入(t-SNE)44均匀流形近似与投影(UMAP)45,46,47,两种典型的非线性降维方法,对数据集1进行基因选择。以与PCAUFE算法相同的方式,我们尝试搜索通过的探测χ2使用P由BH准则调整的值。然而,如图S4所示,我们没有发现调整后的探针P平面上的值小于0.01。PCAUFE的第一个算法搜索主成分(即轴),其负载在统计上将患者和非患者等两组分开。因此,异常值通过χ2PCAUFE第二算法中的test可视为部分患者与非患者相比异常上调或下调的探针。另一方面,t-SNE和UMAP强调在将高维数据集缩减为二维时,将探针的相似性保留为距离,低维空间中的轴并不总是对应于这两组。因此,单纯使用这些非线性降维方法进行基因选择并不合适,因为这并不意味着低维空间中远离原点的探针就可以作为COVID-19诊断的生物标志物。
此外,应该指出的是,在COVID-19等新型疾病的基因表达分析中,使用最先进的深度学习技术并不总是成功的:我们还应用了最近发表的一种名为使用分层自编码器(scDHA)的单细胞分解方法。48,该方法被描述为可靠地提取每个单元的代表性信息,以数据集1与PCAUFE进行比较。补充图S5为数据集1样本在scDHA降维空间中的散点图。如图所示,scDHA完全不能根据pbmc的基因表达谱将COVID-19患者与非患者区分开。原因可能是这个数据集的样本太少,而用于训练自动编码器的变量太多。PCAUFE比其他方法计算成本更低,因为它只需要以一种特殊的方式对基因表达矩阵应用一次PCA。因此,它已被成功地用于解决各种基因选择问题(详情见:49).如上所述,我们证明了这种非新颖但功能强大的基因选择方法对COVID-19患者基因表达谱数据集的有用性,并提出了COVID-19发展的新机制。
其次,我们根据PCAUFE筛选的123个基因,分别采用LR、SVM和RF三个独立模型对COVID-19患者和非患者进行分类,并证实所有模型的auc均高于90%。我们没有对分类模型使用最先进的深度学习技术的原因是用于交叉验证的数据集中的样本量(即126)不够大,不足以容纳解释变量的数量(即123)。例如,可以通过增加样本数量来构建深度学习分类模型,如50.Liu等。51我们使用卷积神经网络(cnn)根据海马体的fMRI图像来预测阿尔茨海默病患者,但我们没有使用它们,因为我们的患者预测模型中的解释变量是基因表达水平,其中不能假设图像中位置接近的元素之间的相似性。然而,如上所述,PCAUFE选择的基因的统计相关性已经得到了保证,因为我们发现传统的机器学习模型具有足够的预测精度。
为了进一步证明我们结果的稳健性,我们还使用数据集2进行基因选择,并使用数据集1进行聚类分析以验证所选基因的可分离性。PCAUFE、LIMMA、edgeR和DESeq2从数据集2中筛选出的基因数量分别为145、7360、4809和5018。LIMMA、edgeR和DESeq2分别包括PCAUFE使用数据集2选择的145个基因中的79个、82个和82个。另一方面,PCAUFE选择的123和145个基因之间重叠的基因数为38个。对于聚类分析,我们采用了一种无监督学习模型UMAP,因为数据集1只包含34个样本,太少了,无法使用它来训练和测试监督学习模型。结果见补充图。S6表明PCAUFE选择的基因与LIMMA、edgeR、DESeq2一样可以对COVID-19患者进行分类。最后,即使我们切换了基因选择和患者/非患者分类的数据集,我们也得到了类似的结果。
由于我们成功地证实了我们的结果的稳健性,我们接下来研究了123个选定基因的生物学可靠性。首先,我们将123个基因上传到3个富集分析服务器GeneSetDB52, Metascape53和TargetMine54,以补偿每个个体的丰富所带来的偏差。检测到多种免疫相关富集。例如,基因本体(GO)生物过程(BP)术语GO:0019221(细胞因子介导的信号通路)、GO:0060333(干扰素- γ介导的信号通路)和GO:0060337 (I型干扰素介导的信号通路)被三台服务器共同识别。GO细胞成分(CC)术语GO:0042613 (MHC II类蛋白复合物)通过GeneSetDB和TargetMine鉴定。反应组通路R-HSA-877300(干扰素γ信号)、R-HSA-6785807(白介素-4和白介素-13信号)、R-HSA-449147(白介素信号)和R-HSA-1280218(适应性免疫系统)通过metscape和TargetMine鉴定(更多细节,见补充)S1带有_File)。
其次,我们通过检查它们之间的相互作用来确认所识别基因的生物学验证。基因之间的紧密关系表明基因选择是可靠的,因为单个蛋白质很少在不与其他蛋白质合作的情况下发挥作用。因此,我们将123个基因上传到STRING服务器55他们在这些基因的产物中检测到了659种蛋白质之间的相互作用。因此,这123个基因由于其产物之间的功能协作,也丰富了蛋白质-蛋白质相互作用。这些富集分析表明,PCAUFE可以成功识别出一组生物学上可行的与COVID-19相关的基因。
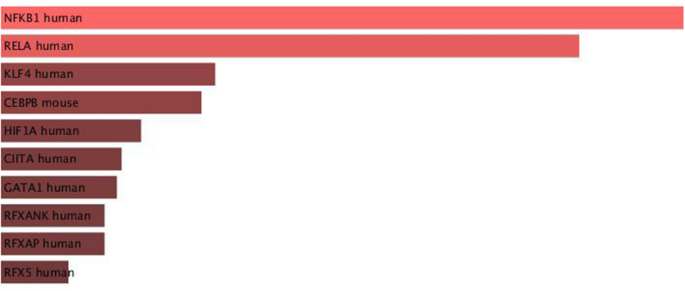
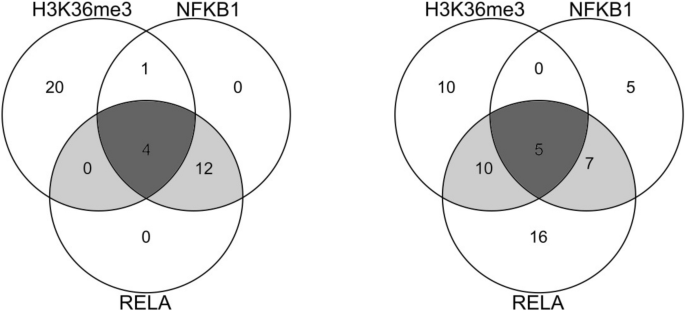
为了研究调控PCAUFE选择的123个基因的上游转录因子(tf),我们还将它们上传到了富贵网56,57,一个多功能的丰富分析服务器。在浓缩铀给出的结果中,NFKB1和RELA的调整较小P-“trust转录因子2019”的值,如图。3..我们还注意到“ENCODE TF ChIP-seq 2015”中排名最高的三个TF绑定:NELFE, RELA和KAT2A(更多细节,参见图2)。S7在补充部分S2带有_File)。
这123个基因也进行了多组蛋白修饰富集,结果见表2.此外,如图。4,组蛋白修饰相关基因与TF靶基因大量重叠。
核因子B (NF-κB) tf在触发和协调先天和适应性免疫反应中起着进化上保守和关键的作用58.NF -κB转录因子家族由5个成员组成:p50、p52、p65 (RelA)、c-Rel和RelB,分别由NFKB1、NFKB2、RelA、REL和RelB编码59.典型NF-的主要介质κB活性是异源二聚体RelA-p50,由RelA转录激活物和nfkb1蛋白p50组成60,61.
Nakshatri等人。62认为NF-κB活性被H3K36me3抑制,这与在这123个基因中观察到的NFKB1-和rela结合位点的富集一致。许多研究也报道了COVID-19患者鼻/口咽拭子和外周血单个核细胞(pmcs)中免疫信号相关基因的表达水平下调。米克等人。63与其他病毒性急性呼吸道疾病相比,COVID-19的特征是先天性免疫反应减弱,与toll样受体和白介素信号、趋化因子结合、中性粒细胞脱粒以及与淋巴样细胞相互作用相关的基因表达减少。Meckiff等人。64显示sars - cov -2反应性CD4+与流感反应细胞相比,T细胞表达的免疫相关转录物水平明显较低。欧阳等。65报道重度病例中表达不足的基因主要涉及th17细胞分化、细胞因子介导的信号通路和t细胞激活。李等人。66报道称,在严重的COVID-19 pbmc中,介导t细胞受体信号转导的蛋白质下调。
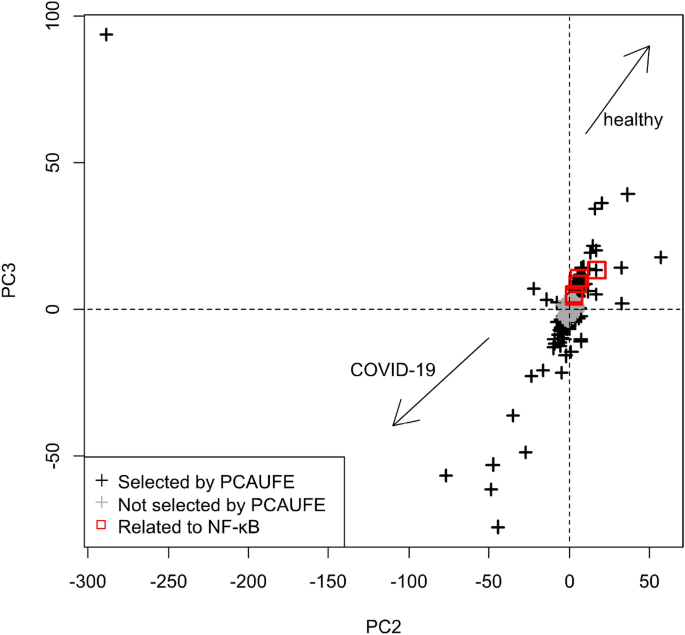
研究无花果中5个重叠基因的表达变化。4,我们通过如图所示的散点图确认数据集1的PC2和PC3分数。5.PC2和PC3阴性定位的探针在COVID-19中主要上调。另一方面,在两种pc上定位为阳性的探针在COVID-19中主要被下调。如图中红色方块所示。5,即Fig中的重叠基因。4对两台电脑都有积极的定位。因此,这些重叠的基因在COVID-19中被下调。这些分析结果与上述文献一致62,63,64,65,66.
NF -κB作为抗癌治疗的靶标,在制药公司中进行了许多积极的研究67.NFKB1和RELA的异常表达是通过mRNA的修饰介导的68.N4-乙酰胞苷(N4A)在RNA表达方面的最新进展也在癌症发展中发挥着关键作用69.段杰等。70据报道,核苷代谢物N4A通过诱导HMGB1信号通路激活小胶质细胞并维持NLRP3炎性小体的激活。通过N4A激活NF-释放HMGB1κB和诱导NLRP3表达70.NLRP3炎性小体被适当激活并能够释放成熟的IL-1β71,72,73.il - 1β是炎症反应的重要介质,参与多种细胞活动,包括细胞增殖、分化和凋亡74,75.
虽然我们通过PCAUFE和富集分析鉴定了123个与COVID-19相关的基因和上游转录因子,但我们尚未评估这些基因是否对COVID-19的发展具有潜在的因果影响。孟德尔随机化76,77,78,79,80方法已广泛用于研究基因与疾病结局之间的因果关系。例如,Zhang等。79使用MR方法调查PTSD和抑郁表型之间的因果关系。在另一项研究中80MR分析结果表明,遗传变异在神经质对心理健康和心血管疾病的因果影响中起中介作用。该方法使用遗传多态性信息作为操作变量,但不幸的是,我们使用的数据集不包括该信息。此外,多态性可能有几种与疾病相关的表型效应。因此,我们将MR方法应用于COVID-19患者的基因表达谱作为未来的工作。
综上所述,我们将PCAUFE应用于COVID-19患者和健康受试者外周血单核细胞的基因表达水平,筛选出123个COVID-19相关基因。然后,通过富集分析,我们确定了调控这些基因表达的转录因子和组蛋白修饰。这123个基因的表达可能涉及NELFE、RELA、KAT2A和NFKB1四种转录因子和组蛋白修饰H3K36me3。NFKB1、RELA和H3K36me3在调控表达的基因中被发现重叠。这两个转录因子与NF-有关κB, H3K36me3可以抑制它。事实上,当我们比较COVID-19患者和健康受试者之间GSE152418中NFKB1、RELA和H3K36me3重复基因的表达水平时,我们观察到COVID-19患者的表达水平下降。这些结果表明,典型的NF-κH3K36me3抑制了COVID-19患者外周血质的B活性。
数据集1的PC2和PC3分数的散点图。黑色叉表示PCAUFE选择的探针,而灰色叉表示未选择的探针。红色方块与图中五个重叠的基因有关。4.
方法
基因表达谱
两个在活的有机体内基因表达数据集,GSE15241828和GSE15710381,从基因表达Omnibus下载82.下面,我们将它们分别表示为数据集1和数据集2。PCAUFE应用于数据集1,该数据集描述了每个受试者pbmc中每种mRNA的表达水平。探针数量为60683个。然后使用数据集2来确认PCAUFE选择的基因的统计有效性。该数据集还描述了每个受试者pbmc中每个基因的表达水平。数据包括COVID-19患者和患有与SARS-CoV-2无关的急性呼吸窘迫综合征(ARDS)的非COVID-19患者。基因数量为19472个。每个基因的表达水平我(= 1,2……N)为PCAUFE标准化,即我们设定\(\frac{1}{N}\sum_{i} x_{ij} = 0\)而且\(\frac{1}{N}\sum_{i} x_{ij}^{2} = 0\).这些基因表达谱中包含的样本的详细信息见表S3在补充部分S2带有_File。
PCAUFE
下面简要解释本研究中使用的PCAUFE程序(更多详细信息,请参见9,12,17,27,49).让x我j表达我的-th mRNA探针jTh样本,让\(\frac{1}{N}\sum_{i} x_{ij} = 0\)而且\(\frac{1}{N}\sum_{i} x_{ij}^{2} = 0\),在那里N为m-RNA探针的数量。首先,我们对行和列分别为基因和样本的数据集应用主成分分析。与通常使用的PCA相比,其中样本是嵌入的,而基因是嵌入在这个实现中。通过使用t-test中,我们指定了两种主成分(PCs),其负载按最小顺序将患者与健康对照样本进行统计学上的区分P价值。注意,与普通的PCA不同,该操作并不保证前两台pc将被选中。第二,通过使用χ2使用P由BH准则调整的值29,我们确定了异常值PC评分(即与调整后的基因相关的基因)P-值小于0.01)以及指定的pc作为疾病相关基因的候选。请注意,PCAUFE中的PC评分与特征(即mRNA探针)相关,而不是与样本相关,这与PCA的普通用法相反。
患者/非患者分类模型
为了验证PCAUFE选择的基因对COVID-19患者的诊断有用,我们使用三种标准预测模型,根据所选基因进行患者/非患者分类:logistic回归(LR)31),支持向量机(SVM32,33)和随机森林(RF34).三种模型各超参数的详细说明请参见补充表S1.根据非COVID-19患者和COVID-19患者的不同,每个样本的目标变量分别为0或1。解释变量为与PCAUFE、edgeR或DESeq2选择的基因相关的探针的基因表达。我们随机将数据集2的80%分配给训练集,剩余的分配给测试集。绘制各模型的受试者工作特征(ROC)曲线,计算曲线下面积(AUCs)。
参考文献
吴,F。et al。中国与人类呼吸道疾病相关的新型冠状病毒。自然579, 265 - 269。https://doi.org/10.1038/s41586-020-2008-3(2020)。
周,P。et al。与一种可能来自蝙蝠的新型冠状病毒有关的肺炎爆发。自然579, 270 - 273。https://doi.org/10.1038/s41586-020-2012-7(2020)。
朱,N。et al。2019年中国肺炎患者的新型冠状病毒。心血管病。j .地中海382, 727 - 733。https://doi.org/10.1056/nejmoa2001017(2020)。
关,W。et al。2019年中国新型冠状病毒感染的临床特征心血管病。j .地中海382, 1708 - 1720。https://doi.org/10.1101/2020.02.06.20020974(2020)。
他,X。et al。covid-19病毒脱落和传播的时间动态。Nat,地中海。26, 672 - 675。https://doi.org/10.1101/2020.03.15.20036707(2020)。
魏,W。et al。新加坡,2020年1月23日至3月16日,新冠病毒症状前传播。MMWR Morb。致命的工作。代表。69, 411-415(2020)。
杨锐,桂霞,熊勇。2019年武汉地区无症状与有症状冠状病毒病临床特征比较。JAMA Netw。开放3., e2010182。https://doi.org/10.1001/2Fjamanetworkopen.2020.10182(2020)。
美国疾病控制与预防中心(2021年2月2日)。https://www.cdc.gov/coronavirus/2019-ncov/hcp/clinical-guidance-management-patients.html.
Taguchi, Y. H.基于无监督特征提取的主成分分析应用于公开的基因表达谱,为组蛋白去乙酰化酶抑制剂的作用机制提供了新的见解。Neuroepigenetcis8队。https://doi.org/10.1016/j.nepig.2016.10.001(2016)。
田口,中州。,Iwadate, M. & Umeyama, H. Principal component analysis based unsupervised feature extraction applied to budding yeast temporally periodic gene expression.BMC医学基因组学9, 69 - 79。https://doi.org/10.1186/s12920-016-0196-3(2016)。
Taguchi, Y. H. Sfrp1是非小细胞肺癌表观遗传治疗的可能候选者。个人经历分钟。9, 22岁。https://doi.org/10.1186/s12920-016-0196-3(2016)。
Taguchi, Y. H.使用基于无监督特征提取的主成分分析鉴定多种癌症中更可行的MicroRNA-mRNA相互作用。Mol科学17(5), 696年。https://doi.org/10.3390/ijms17050696(2016)。
在E13和E16大鼠F3代vinclozolin谱系的原始生殖细胞中,与异常启动子甲基化相关的异常基因表达的鉴定。BMC Bioinform。16, s16(2015)。
田口,中州。基于主成分分析的无监督特征提取对非小细胞肺癌细胞系重编程过程中基因表达和启动子甲基化的综合分析。在黄里,d - s。,Han, K. & Gromiha, M. (eds.)生物信息学中的智能计算,卷8590信号, 445-455(施普林格国际出版社,海德堡,2014)。
田口,中州。,Iwadate, M., Umeyama, H., Murakami, Y. & Okamoto, A. Heuristic principal component analysis-based unsupervised feature extraction and its application to bioinformatics. In Wang, B., Li, R. & Perrizo, W. (eds.)生物信息学和医疗保健中的大数据分析(IGI Global, 2015)。
田口,中州。,Iwadate, M. & Umeyama, H. Heuristic principal component analysis-based unsupervised feature extraction and its application to gene expression analysis of amyotrophic lateral sclerosis data sets. In生物信息学与计算生物学中的计算智能(CIBCB), 2015年IEEE会议1 - 10,https://doi.org/10.1109/CIBCB.2015.7300274(2015)。
田口,杨,岩田,M. & Umeyama, H.基于主成分分析的无监督特征提取在创伤后应激障碍介导的心脏病药物开发中的应用。BMC Bioinform。16, 139年。https://doi.org/10.1186/s12859-015-0574-4(2015)。
Umeyama, H., Iwadate, M. & Taguchi, Y. H. TINAGL1和B3GALNT1是抑制非小细胞肺癌转移的潜在治疗靶基因。BMC基因组学15, S2。https://doi.org/10.1186/1471-2164-15-s9-s2(2014)。
村上,Y。et al。肝内胆管癌和肝细胞癌转录组和代谢组分析的综合分析。科学。代表。5, 16294年。https://doi.org/10.1038/srep16294(2015)。
村上,Y。et al。通过下一代测序和微阵列评估肝细胞癌miRNA表达谱的比较。《公共科学图书馆•综合》9, e106314。https://doi.org/10.1371/journal.pone.0106314(2014)。
村上,H。et al。综合分析外周血miRNA表达可以诊断肝脏疾病。《公共科学图书馆•综合》7, e48366。https://doi.org/10.1371/journal.pone.0048366(2012)。
周,X。et al。异常表达的miR-193b-3p通过调节转化生长因子-β信号通路参与子痫前期。Sci代表。29(6), 19910年。https://doi.org/10.1038/srep19910(2016)。
Taguchi, Y. H. & Murakami, Y.基于主成分分析的特征提取方法识别循环microRNA生物标志物。《公共科学图书馆•综合》8, e66714。https://doi.org/10.1371/journal.pone.0066714(2013)。
Kinoshita, R., Iwadate, M., Umeyama, H. & Taguchi, Y. H.鳞状细胞癌基因型特异性DNA甲基化相关基因作为候选药物靶点。BMC系统。医学杂志。8S4。https://doi.org/10.1186/1752-0509-8-s1-s4(2014)。
石田,S., Umeyama, H., Iwadate, M. & Taguchi, Y. H.利用FAMS进行自身免疫性疾病基因的生物信息学筛选和蛋白质结构预测用于药物发现。蛋白质Pept。21, 828 - 839。https://doi.org/10.2174/09298665113209990052(2014)。
田口,中州。& Okamoto, A.细菌蛋白质组学分析的主成分分析。涉谷,T.,鹿岛,H., Sese, J. & Ahmad, S.(编)生物信息学中的模式识别,卷7632信号, 141 - 152,https://doi.org/10.1109/BIBMW.2011.6112520(施普林格国际出版社,海德堡,2012)。
田口,中州。基于无监督特征提取的主成分分析应用于登革出血热患者血液基因表达分析。科学。代表。7, 44016年。https://doi.org/10.1038/srep44016(2017)。
**am, p.s.。et al。人类对轻度和重度COVID-19感染的免疫系统生物学评估。科学369, 1210 - 1220。https://doi.org/10.1126/science.abc6261(2020)。
Benjamini, Y. & Hochberg, Y.控制错误发现率:多重测试的一种实用而强大的方法。J. R.统计社爵士。B(方法论)57, 289-300(1995)。
Bradley, A. P. ROC曲线下面积在机器学习算法评估中的使用。Recognit模式。30., 1145-1159(1997)。
二元序列的回归分析(附讨论)。J. R.统计社爵士。B (Methodol。)20., 215 - 232。https://doi.org/10.1111/j.2517-6161.1958.tb00292.x(1958)。
范志强,李志强,李志强,等。基于广义人像方法的模式识别。奥特曼。遥远。控制。24, 774-780(1963)。
Cortes, C. & Vapnik, V.支持向量网络。马赫。学习。20., 273 - 297。https://doi.org/10.1007/2FBF00994018(1995)。
布莱曼,《随机森林》。马赫。学习。45, 5-32。https://doi.org/10.1023/2FA%3A1010933404324(2001)。
王,X。et al。孕妇妊娠三个月期间维生素D状态与新生儿脐带血25(OH)D浓度之间的关系:一项前瞻性上海出生队列研究Eur J Nutr。https://doi.org/10.1007/s00394-021-02528-w(2021)。
Yu H。et al。LEPR低甲基化与男性胃癌显著相关。Exp、Mol、Pathol。https://doi.org/10.1016/j.yexmp.2020.104493(2020)。
杜淑芳,朱晓明,朱晓明。微阵列对电离辐射响应的显著性分析。PNAS98(9), 5116 - 5121。https://doi.org/10.1073/pnas.091062498(2001)。
里奇,m.e。et al。用于rna测序和微阵列研究的limma幂差表达分析。核酸测定。43(7) e47。https://doi.org/10.1093/nar/gkv007(2015)。
Robinson, m.d., McCarthy, d.j. & Smyth, G. K. edgeR:用于数字基因表达数据差异表达分析的Bioconductor包。生物信息学26, 139 - 140。https://doi.org/10.1093/bioinformatics/btp616(2010)。
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组医学杂志。15, 550年。https://doi.org/10.1186/s13059-014-0550-8(2014)。
李,H。et al。共表达网络分析确定了甘油三酯和游离脂肪酸代谢关键的枢纽基因是小鼠年龄相关血管功能障碍的关键调节因子。老龄化(纽约州奥尔巴尼)11(18), 7620 - 7638。https://doi.org/10.18632/aging.102275(2019)。
陈,J。et al。产前营养不良影响大鼠海马的遗传调控子网络和关键调控基因:对主要脑疾病的影响。老龄化(纽约州奥尔巴尼)12(9), 8434 - 8458。https://doi.org/10.18632/aging.103150(2020)。
朗菲尔德,P.和霍瓦特,S. WGCNA:加权相关网络分析的R包。BMC Bioinforma。9, 559年。https://doi.org/10.1186/1471-2105-9-559(2008)。
Van Der Maaten, L. & Hinton, G.使用t-SNE可视化数据。j·马赫。学习。Res。9, 2579-2605(2008)。
McInnes, L., Healy, J., Saul, N. & Großberger, L. UMAP:均匀流形近似和投影。J.开源软件。3.(29), 861年。https://doi.org/10.21105/joss.00861(2018)。
白克,E。et al。使用UMAP可视化单单元数据的降维。生物科技Nat。》。37, 38-44。https://doi.org/10.1038/nbt.4314(2019)。
McInnes, L., Healy, J. & Melville, J. UMAP:统一流形近似和投影降维。arXivhttps://arxiv.org/abs/1802.03426(2020)。
Tran, H. D., Nguyen, T. B., Vecchia, L. C., Luu, N. H. & Nguyen, T.使用分层自编码器进行快速和精确的单细胞数据分析。Commun Nat。12(1), 1029。https://doi.org/10.1038/s41467-021-21312-2(2021)。
田口,中州。应用于生物信息学的无监督特征提取:基于PCA和基于TD的方法(施普林格国际出版社,2019)。
冯,C。et al。基于基因表达数据的深度学习模型用于药物性肝损伤的提前准确预测。j .化学。通知。模型。59, 3240 - 3250。https://doi.org/10.1021/acs.jcim.9b00143(2019)。
刘,M。et al。用于阿尔茨海默病海马体自动分割和分类的多模型深度卷积神经网络。科学杂志https://doi.org/10.1016/j.neuroimage.2019.116459(2020)。
Araki, H., Knapp, C., Tsai, P. & Print, C. Genesetdb:基因集分析的综合元数据库,统计和可视化框架。2月Openbio2, 76 - 82。https://doi.org/10.1016/j.fob.2012.04.003(2012)。
周,Y。et al。metscape为分析系统级数据集提供了面向生物学家的资源。Commun Nat。10(1), 1523。https://doi.org/10.1038/s41467-019-09234-6(2019)。
陈,Y.-A。,Tripathi, L. & Mizuguchi, K. TargetMine, an integrated data warehouse for candidate gene prioritisation and target discovery.《公共科学图书馆•综合》6(3) e17844。https://doi.org/10.1371/journal.pone.0017844(2011)。
Szklarczyk D。et al。STRING v10:蛋白质-蛋白质相互作用网络,整合在生命树上。核酸测定。43(D1) D447-D452。https://doi.org/10.1093/nar/gku1003(2015)。
陈,E。et al。enrichment:交互式和协作的html5基因列表丰富分析工具。BMC Bioinform。14, 128年。https://doi.org/10.1186/1471-2105-14-128(2013)。
Kuleshov, M。et al。enrichment:一个全面的基因集富集分析web服务器2016年更新。核酸测定。44(W1) W90-W97。https://doi.org/10.1093/nar/gkw377(2016)。
高希,S. & Karin, M. NF-kappaB拼图中缺失的部分。细胞109, S81-S96。https://doi.org/10.1016/s0092 - 8674 (02) 00703 - 1(2002)。
海登,高希。NF-的共同原则κB信号。细胞132, 344 - 362。https://doi.org/10.1016/j.cell.2008.01.020(2008)。
莫尔西,k。et al。20S蛋白酶体处理NF-κ将bb1 p105转换为p50。EMBO J。25, 1945 - 1956。https://doi.org/10.1038/sj.emboj.7601081(2006)。
Basak, S., Shih, F. V. & Hoffmann, A. NF-kappaB信号系统中多种二聚体转录因子的生成和激活。细胞生物学28(10) 3139 - 3150。https://doi.org/10.1128/mcb.01469-07(2008)。
Nakshatri, H。et al。NF -κ使用新型抗癌剂DMAPT进行b依赖和-独立的表观遗传调制。细胞死亡。6(1) e1608。https://doi.org/10.1038/cddis.2014.569(2014)。
米克,E。et al。上呼吸道基因表达将COVID-19与其他急性呼吸道疾病区分开来,并揭示了SARS-CoV-2抑制先天免疫反应。medRxiv4, e1608。https://doi.org/10.1101/2F2020.05.18.20105171(2020)。
梅基夫,j.b.。et al。调节性和细胞毒性sars - cov -2反应性CD4的不平衡+COVID-19中的T细胞。细胞https://doi.org/10.1016/j.cell.2020.10.001(2020)。
欧阳,Y。et al。COVID-19患者在疾病进展过程中基因表达谱下调和免疫反应发生变化。中国。感染。说。ciaa462, 1 - 9。https://doi.org/10.1093/cid/ciaa462(2020)。
李,J。et al。病毒-宿主相互作用组和蛋白质组研究揭示了影响SARS-CoV-2发病机制的潜在毒力因素。Med (N Y)。https://doi.org/10.1016/j.medj.2020.07.002(2020)。
Escárcega, r.o, Fuentes-Alexandro, S, García-Carrasco, M., Gatica, A. & Zamora, A.转录因子核因子-kappa B与癌症。中国。肿瘤防治杂志。19(2), 154 - 161。https://doi.org/10.1016/j.clon.2006.11.013(2007)。
Ferrero-Andrés, A., Panisello-Roselló, A., Roselló-Catafau, J. & Folch-Puy, E.急性胰腺炎NLRP3炎症体介导的炎症。Int。理学。21(15), 5386年。https://doi.org/10.3390/ijms21155386(2020)。
金刚,徐敏,邹明,段松。系统综述了n4 -乙酰胞苷对RNA的作用、基因调控、生物学功能及临床意义。摩尔。其他。核酸20., 24里面。https://doi.org/10.1016/j.omtn.2020.01.037(2020)。
段,J。et al。n4 -乙酰胞苷是小胶质细胞中通过HMGB1途径持续激活NLRP3炎性小体所必需的。手机信号58, 44-52。https://doi.org/10.1016/j.cellsig.2019.03.007(2019)。
曼根,m.s.j。et al。在炎症性疾病中靶向NLRP3炎性小体。Nat. Rev.药物发现17(9), 688年。https://doi.org/10.1038/nrd.2018.149(2018)。
Zaki, M. H., Lamkanfi, M. & Kanneganti, T. D. Nlrp3炎性小体:对肠道稳态的贡献。Immunol趋势。Immunol趋势。32(4), 171 - 179。https://doi.org/10.1038/nrd.2018.149(2011)。
郑,S。et al。免疫缺陷促进宿主肠道微生物群的适应性改变:小鼠的观察性宏基因组研究。Microbiol前面。1(10), 2415年。https://doi.org/10.3389/fmicb.2019.02415(2019)。
Tulotta, C. & Ottewell, P. IL-1B在乳腺癌骨转移中的作用。Endocrine-Relat。癌症25(7) R421-R434。https://doi.org/10.1530/2FERC-17-0309(2018)。
闫霞,赵霞,李娟,何玲,徐敏。早期营养不良对神经发育和神经精神障碍的影响及其可能机制。掠夺。Neuropsychopharmacol。医学杂志。精神病学。83, 64 - 75。https://doi.org/10.1016/j.pnpbp.2017.12.016(2018)。
载脂蛋白E亚型,血清胆固醇和癌症。《柳叶刀》327, 507 - 508。https://doi.org/10.1016/s0140 - 6736 (86) 92972 - 7(1989)。
吴,Y。et al。五种精神疾病全基因组关联研究的多特征分析。Transl精神病学。10(1), 209。https://doi.org/10.1038/s41398-020-00902-6(2020)。
王旭,方旭,郑伟,周俊,宋忠,徐明,闵俊,王峰,铁含量与2型糖尿病因果关系的遗传支持:孟德尔随机化研究。j .中国。性。金属底座,2021。
张,F。et al。神经质对心理健康和心血管疾病的因果影响。嗡嗡声。麝猫。https://doi.org/10.1007/s00439-021-02288-x(2021)。
张,F。et al。遗传证据表明创伤后应激障碍是重度抑郁症的一个亚型。j .中国。Investig。27, 145942年。https://doi.org/10.1172/jci145942(2021)。
欧弗迈尔,k。et al。COVID-19严重程度的大规模多组学分析。细胞系统。12队。https://doi.org/10.1016/j.cels.2020.10.003(2020)。
Edgar, R., Domrachev, M. & Lash, A.基因表达综合:NCBI基因表达和杂交阵列数据存储库。核酸测定。30.(1), 207 - 210。https://doi.org/10.1093/nar/30.1.207(2002)。
确认
这项研究得到了冲绳县“促进健康和医疗行业使用信息技术项目”(20G1000012)的支持。
作者信息
作者及隶属关系
贡献
k.f.、y.t.和R.M.设计了这项研究;k.f.、m.s.和R.M.撰写了手稿;K.F.和M.S.进行了分析;k.f., y.t., s.i.和R.M.解释了结果;S.I.和R.M.监督了这项研究;所有的作者都审阅了手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
藤泽,K, Shimo, M,田口,YH。et al。基于pca的无监督特征提取技术用于COVID-19患者基因表达分析。Sci代表11, 17351(2021)。https://doi.org/10.1038/s41598-021-95698-w
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-021-95698-w
这篇文章被引用
基于pca的无锗纳米表面形貌的亚表面结构和缺陷分析
科学报告(2022)