文摘
正在SARS-CoV-2大流行性病毒测试需求和后续扩展导致问题诊断实验室努力和确保基本供应样品的收集和处理。这动机科学界努力建立改进协议的可伸缩性更强,更少的资源密集型和便宜。一个这样的发展努力导致了一个名为“Swab-Seq”的分析,之所以这么叫是因为它最初开发与干鼻拭子样本。现有的黄金标准测试由RNA提取从鼻咽拭子(NP)进行定量逆转录聚合酶链反应(存在)。新一代测序读出Swab-Seq适应这种方法。通过这一修改搭配extraction-free抽样技术,Swab-Seq达到高可伸缩性,每样低成本,合理的周转时间。我们评估的有效性分析在社区监测环境中通过测试样本收集从有症状和无症状的个体使用传统的NP拭子。此外,我们评估extraction-free抽样技术(包括口腔唾液和盐水漱口样本)。我们发现试验存在一样敏感的临床试验,适应多种样本类型,并可以很容易地适应数以百计的样品。我们提供独立的验证Swab-Seq和延长其效用样品类型和样品的稳定性。 Assays of this type greatly expand the possibility of routine, noninvasive, repeated testing of asymptomatic individuals suitable for current and potential future needs.
介绍
随着SARS-CoV-2病毒传播世界各地在2019年末和2020年初,有关病毒一样迅速传播的信息。虽然最初的病例首次报道中国以外的2019年12月30日1病毒的全基因组是存入基因库2020年1月5日(加入MN908947数量2)。令人印象深刻的是,在1月13日,世界卫生组织(世卫组织)已经发布了一个定量逆转录酶聚合酶链反应(存在)试验,开发使用的部分SARS-CoV-2序列之前被释放创建一组三个不同的探针/引物对3。
虽然许多额外的测试策略也已经被开发出来,最常见的样本采集工作流程检测病毒的存在包括鼻咽拭子(NP)由医疗服务提供者,放置在传输媒体和转移到诊断实验室4。在这一点上,RNA提取来自媒体和用作模板中存在测试。几种不同的采样技术进行测试5然而,疾病控制中心(CDC)和NP棉签作为主要的上市推荐抽样技术在大流行6,7。然而,理想的抽样类型仍未得到解决4,8,9,10,11。在2020年显著增加测试需求迅速扩大,导致全球短缺这个工作流所需的许多组件。这些短缺反过来又促使许多团体实现替代现有的方法(例如Ref。12)或开发协议,消除许多标准的步骤(例如参考文献。4,13,14,15)。尽管各种新的化验报告广泛的返回结果,可伸缩性和敏感性,许多组织强调的最重要的特性充分分析是可扩展的,允许重复测试无症状人群16,17,18。
这样一个试验,称为“Swab-Seq”,主要由加州大学洛杉矶分校的组织开发和八分仪生物14。Swab-Seq试验使用一组针对飙升的S1亚单位基因的引物(虽然叫“S2”引物),来自裁判。19,停在同一地区的合成记录添加到每个反应在一个固定的拷贝数为内部标准化跨井,可以区别病毒序列的基础上,6个碱基对(bp)条形码。测序放大cDNA产品后,病毒载量的读出一个对数转换的数量比病毒RNA数量除以合成“激增”记录的数量计数(“S2 /激增”比率)。开发试验后,创造者发布了公开的协议在一个所谓的“COVID许可证”,建立了一个用户组来促进这个和其他协议的实现。
动机的一个明显不足的测试自己的亚利桑那州20.,我们参加这些会议,开始评估实施Swab-Seq作为可行的测试策略的可能性在我们的社区。我们第一次评估的分析灵敏度试验使用的样本(即在我们的手中。,samples to which synthetic viral RNA had been added), and then by leveraging samples collected as a part of ongoing studies at our campus testing facility, we explored the suitability of Swab-Seq for routine monitoring of asymptomatic populations by directly comparing Swab-Seq results with the results obtained using the traditional qRT-PCR assay on the same samples. In addition, we used Swab-Seq to test non-traditional sample types: saliva and saline gargle. We found that the assay was easy to implement, comparable to qRT-PCR in terms of clinical sensitivity in asymptomatic individuals (even in our non-CLIA setting), and that it is flexible to various sampling methods (including non-invasive and extraction-free sampling methods). Our results validate and extend recent method development4,13,14,15并展示了潜力更精简工作流在当前流感大流行病毒测试和在未来。
结果
Swab-Seq强劲跨各种PCR试剂盒和模板
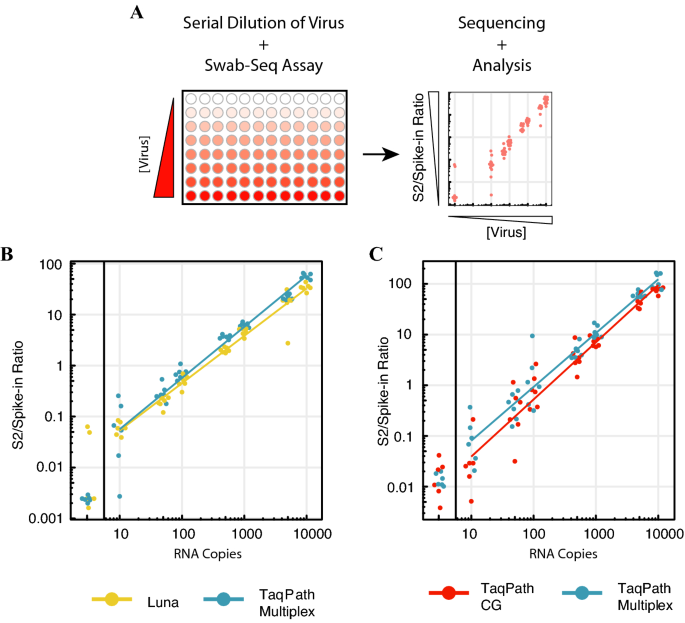
评估Swab-Seq的实现,我们最初执行一组质量控制实验(图。1在裁判的)。。14,我们使用连续稀释的合成病毒RNA(扭转生物科学)或heat-inactivated SARS-CoV-2(写明ATCC)建立分析检测的灵敏度和线性度。合成RNA,我们估计,分析了检测极限(LOD)大约10份病毒基因组/反应(相当于每毫升2000基因组复制等价物或“全球教育运动”的示例):10的12从负控制在这个复制的病毒载量在我们最初的实验与AB TaqPath互译多元主混合工具(蓝点在无花果。1B, C,参见补充图。S1补充表S1,S2),和一个额外的12 12复制在这个负载的底片不管我们使用合成RNA或灭活病毒颗粒在随后的实验(补充图。S2补充表S3)。虽然我们估计不符合FDA欧洲大学协会阈值(要求19 20复制的LOD),我们注意到,在两个现有的欧洲大学协会批准Swab-Seq LOD决心125 GCE /毫升21和250年全球教育运动/毫升22。还值得注意的是,在这两种情况下申请人停在病毒颗粒在RNA提取之前,这将有效地集中病毒RNA。因此,我们估计2000年~ GCE /毫升代表估计LOD没有集中的样本。我们还证实,分析作品与三个不同的互译中存在工具:内卢娜和两种不同配方的AB TaqPath(无花果。1B, C,补充图。S1补充表S1,S2),部分重叠的裁判所使用的工具。14。与月神装备,TaqPath工具包包含尿嘧啶-N糖基化酶()),这有助于消除污染以前结转PCR产品。因此,我们使用一个TaqPath存在包、TaqPath的互译混合多元主装备,对于大多数实验。
Swab-Seq社区监测测试
Swab-Seq以前证明区分症状患者样本和消极的控制14FDA规定的紧急使用授权(协会)的指导方针21,22,23。然而,由于测试方案已经变得更加普遍通用监测项目,我们想看看分析将执行相对于黄金标准在一般人群中存在以及在有症状的个体(图。2)。评估与Swab-Seq社区监测的可行性,我们测试了四个96孔板的样本,其中包括257从NP拭子中提取RNA样本和127控制井(控制进一步解释”方法”)。这些盘子的样品代表最初收集了来自两个团体组81名个人和176名无症状的个体症状。这些样本(控制)之前已经经过测试与标准中存在方法使用CDC-recommended引物(N1和N2位点)CLIA条件下。
评价Swab-Seq作为一个社区监测分析。(一个)测试方案的示意图。RNA提取后,所有样品都测试通过存在,Swab-Seq 40周期放大,或Swab-Seq 50周期的放大。(B)Ct值存在一式三份(平均)N1引物组(x轴)Ct值相比存在的一式三份)(平均N2底漆(轴)。(C一式三份)Ct值(平均)N1引物组(x轴)相比,S2 / Swab-Seq激增率与40个周期(轴)。(D一式三份)Ct值(平均)N1引物组(x轴)相比,S2 / Swab-Seq激增率50周期(轴)。黑色竖线分割“负面”从“积极”的样本。每个点是一个单独的样品。对角线是最小二乘拟合线将所有样品化验的“积极的”。(E- - - - - -G)表样本的数量分为正面或负面的存在病毒N1存在之间的比较分析,提出了(EN2中存在,F)Swab-Seq40, (G)Swab-Seq50。正(Pos)、负(“底片”),和不确定的(“公司”)两两比较结果。积极(PPA)和消极% %协议协议(NPA)为每个提供比较,连同95%置信区间(在括号中)与裁判的“威尔逊”方法计算。25。
在样品提供给我们的研究中,60%(49/81)的症状个人和9%(16/176)的无症状的人存在积极的N1引物。N1和N2的Ct值是高度相关(皮尔森的r = 0.97, p = 4.2×10-38年,无花果。2B)对所有样品检测到病毒,尽管两组引物没有完全整合在确定阳性样本Ct阈值(65 N1、N2 63 59发现共同之处)。比较两个存在的临床敏感性分析,积极%协议N2和N1存在确定为91%(95%可信区间(CI) 81.3 - -95.7%;无花果。2E)。- %协议是97.9% (95% CI 94.8 - -99.2%)。科恩kappa系数(κ)24,一个两分的可靠性指标,比较为0.9 (95% CI 0.83 - -0.96)。
在测试与Swab-Seq(无花果。2补充图。S3补充表S4),三个主题(和一个控制样本)样品测序深度不符合我们的阈值(> 500读取映射到S2和激增控制相结合),Swab-Seq被认为是不确定的。我们发现存在N1和Swab-Seq日志的Ct值10转换S2 /激增比率是高度相关(皮尔森的r =−0.96 61个样本被两种方法,假定值= 1.0×10-34年)。尤为明显的相关性的~ < Ct < 20 ~ 40(图。2C),尽管Swab-Seq似乎显示一些饱和度很高的病毒载量。令人放心的是,Ct值RPP30(人类控制基因)不与对数转换Swab-Seq比率(皮尔森的r =−0.04, p = 0.5,补充图。S3E)的相关性RPP30水平从Swab-Seq试验中存在的Ct值RPP30最初低样本(皮尔森的r =−0.46, p = 1.0×10-13年补充图。S3F)。然而,这个我们过滤掉样品后大幅上涨如此之高病毒载量,他们镇压激增控制数量,我们利用规格化因素Swab-Seq方法(皮尔森的r = 0.73, p = 3.2×10-35年不包括34个样本后< 600激增控制读取,补充图。S3G)。此外,我们观察到没有证据表明板块效应的影响之间的关系中存在和Swab-Seq(补充图。S4)。重要的是,Swab-Seq积极%协议为94%(61/65)和N1存在(95% CI 85.2 - -97.6%;无花果。2F),高于N2中存在底漆。- %协议是96.4% (95% CI 92.7 - -98.2%)。κ系数为0.89 (95% CI 0.83 - -0.95)。Swab-Seq也检测到病毒RNA S2-positive控制井。124负控制井(包括四个含有N控制序列但不是S2), Swab-Seq给予积极结果四,有三四个已经积极的N1存在试验,因此建议Swab-Seq假阳性率为0.8% (1/121)。
虽然这与40个循环执行初始测试Swab-Seq (“Swab-Seq40”从这里),我们指出,50个周期(“Swab-Seq50”)已经在某些情况下使用Ref。14据称,更好的灵敏度。为了测试这个直接,我们重新处理所有的样品使用Swab-Seq50(无花果。2A、D补充图。S5一、补充表S5)。三个样本和负控制的四个主题不符合我们的阈值结合S2和激增读取(> 500读相结合)这一次。比较Swab-Seq50和存在,积极%协议为90.8%(95%可信区间81.3 - -95.7%;无花果。2G),皮尔森的r =−0.95 (p = 3.9×10-31年积极的化验(图)样品。2D)。有趣的是,Swab-Seq40的- %协议从96.4%下降到93.2% (95% CI 88.8 - -96.0%),这可能表明Swab-Seq50更能够识别真正的阳性或更容易出现假阳性。κ系数为0.81 (95% CI 0.73 - -0.89)。比较这两个Swab-Seq直接测试,皮尔森的r为0.98 (p = 5.2×10-40年样品都积极的主题,和Swab-Seq50发现88%(57/65)的样品检测与Swab-Seq40积极。也明显,S2 /激增比率值较低的负样本和更高的正样本Swab-Seq50比Swab-Seq40(补充图。S5B),表明有可能真正的增益灵敏度。Swab-Seq40确定8阳性证实了这不是Swab-Seq50(四也积极通过N1存在),而Swab-Seq50确定12没有检测到Swab-Seq40(其中两个还N1存在积极的)。包含S2的两个积极的控制序列被Swab-Seq50发现看作是积极的(就像他们已经通过Swab-Seq40)。然而,11负控制样本检测的积极Swab-Seq50化验(三也检测到存在),显示一个潜在的假阳性率高达6.8% (8/118)。我们解释这些数据表明,在某些情况下Swab-Seq50错过了真正的优点,包括假阳性,而一般趋势似乎是Swab-Seq50整体能够发现更多的阳性(图。2补充图。S5B)。
Swab-seq在唾液
在原始Swab-Seq手稿14,作者证明了Swab-Seq可以用来检测病毒RNA后直接从唾液样本(不含开采)协议开发的参考。13。确认唾液协议工作在我们的手中,我们停在heat-inactivated病毒在特定浓度的唾液收集确认-主题,加热样品为30分钟95°C,稀释1:1的混合此种Tween-20,并加载样品直接到互译PCR反应。在这个实验中,我们既包括96和384孔板来评估可伸缩性。我们发现试验是敏感~ 10 - 50份/反应(4000 - 20000拷贝/毫升唾液中稀释前,无花果。3补充图。S6补充表S6在两种格式)。进一步探索这个试验的重要变量,我们测试了三个主要的比较在一个实验:(1)样本加热的时间(原出版的唾液条件表明,30分钟在95°C所需最佳存在效率13),(2)TaqPath酶的性能在这个更有挑战性的场景中(即在唾液的存在),和(3)的周期数放大(原Swab-Seq手稿,作者使用50周期direct-to-PCR样本对象)。而不同时期的热处理和两种不同酶制剂给等价的结果在我们的手中(补充图。S7补充表S7),我们观察到的一个主要的性能改善消极控制50周期相对于40周期(无花果。3B)。
Swab-Seq在盐水漱口
而唾液结果是有前途的,有挑战的工作saliva-including可变粘度和可变体积,以及一些轶事沉默的个人的想法吐成管状。这增加了难度设计可伸缩、样品处理自动化解决方案(尽管原始Swab-Seq手稿的作者提出一个潜在的方法14)。另一种方法我们大学最近开发和实现是使用生理盐水漱口。在本协议主题给出5毫升丸的盐水漱口的嘴前很短的时间内沉淀的解决方案集合管。该方法降低了粘度和有助于规范化问题卷从每个人,虽然代价大幅稀释的病毒。的基础上列出的优势,我们大学最近盐水漱口作为标准测试实践实现的。与Swab-Seq50评估这个集合的方法,我们首先进行了系列稀释的灭活病毒,我们所做的与其他样品类型。虽然最初的唾液协议测试一系列热失活的时间和温度,发现样本在95°C的孵化30分钟的最优检测病毒,漱口协议最初是用30分钟的65°C实现失活。评估这个Swab-Seq50的上下文中,我们测试了65°C 30分钟和95°C 10, 20或30分钟在一个实验中(补充图。S8补充表S8)。与原报告唾液(每反应评估在100册)1365°C 30分钟出现相当于95°C 10, 20或30分钟到100册/反应。每反应浓度达到或低于50份,样品在95°C的数据显示检测到病毒的一些潜在的退化。
因为这个漱口协议已经实现在我们的校园社区日常测试中,我们也能够测试样本漱口一些无症状感染者。对于这个实验,我们测试了17 qRT-PCR-positive样品和20 qRT-PCR-negative Swab-Seq50在复制样品。比较Ct值CDC-designed存在Swab-Seq50试验,我们发现所有17个阳性样本检测与Swab-Seq50复制集(图。4A, B,补充表S9),一个从负样本池似乎也积极通过Swab-Seq50(无花果。4C)。相关Swab-seq50阳性样本的存在产生了皮尔森的r =−0.75 (p = 5.4×104)和−0.81 (p = 1.0×104)复制集1和2,分别。积极的%协议是100% (95% CI 81.6 -100.0%,无花果。4D, E)中存在与每个Swab-Seq50复制,和消极的%协议是95% (95% CI 76.4 - -99.1%)。κ系数之间的复制和存在是0.95 (95% CI 0.84 - -1.00)。比较Swab-seq50相互复制,皮尔森的r为0.97 (p = 2.2×10-12年),积极%协议是100% (95% CI 82.4 -100.0%,无花果。4F),负%协议是100% (95% CI 83.2 - -100.0%),和κ系数为1.00(95%置信区间CI: N / A)。尽管Swab-Seq只发现一个额外的阳性样本,这些结果提出了这样一种可能性:即Swab-Seq50或许能更好地检测阳性比常规监测中存在的社区使用盐水漱口的协议。
Swab-Seq和盐水漱口社区监测。盐水漱口可检测到病毒样本来自17个主题(存在)和20个样品没有检测到病毒(存在)。样本为Swab-Seq50异形一式两份。(一个)N1的比较中存在和S2 /复制集激增率1。(B)N1的比较中存在和S2 /复制集激增率2。(C)比较S2 /激增比例复制集1复制集的比2。黑暗的垂直和水平线表示识别正样本的阈值在每个试验(Ct < 40的存在,比> 0.002 Swab-Seq50)。行是最小二乘拟合线将“积极”的所有数据点(一个),(B)。在(C),线是斜的。(D- - - - - -F)表的数量样品归类为积极或消极的存在病毒之间呈现(D)复制1或(E)复制2和N1中存在的比较分析,以及(F)之间复制2和复制1比较器。正(Pos)、负(“底片”),和不确定的(“公司”)两两比较结果。积极(PPA)和消极% %协议协议(NPA)为每个提供比较,连同95%置信区间(在括号中)与裁判的“威尔逊”方法计算。25。
SARS-CoV-2口服抽样方法稳定,与Swab-seq兼容
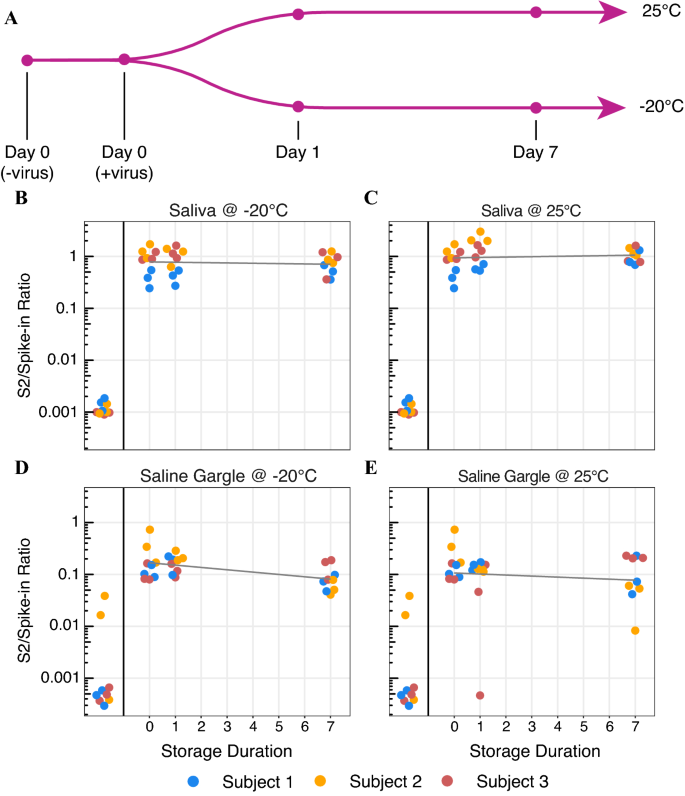
据报道,这一事实可检测SARS-CoV-2病毒RNA在唾液非常稳定26,27可能允许的灵活性对于这样一个试验样品的收集(包括家庭样本的收集和下降)。因此,我们试图确认Swab-Seq50能够检测病毒样本存储长时间−20°C或室温(无花果。5)。这些被选出来代表的极端温度诊断实验室可能会考虑存储样本。测试的稳定性(heat-inactivated)病毒,一次实验课程设置通过收集唾液和盐水漱口三个样本(virus-negative)科目。然后我们上升在heat-inactivated病毒浓度100册/μL唾液和50份每μL盐水漱口(浓度的差异是考虑到后续此种/ Tween-20唾液中的稀释步骤协议)。样本在−20°C或储存在室温下长达一个星期。样本加热30分钟(95°C的唾液,盐水漱口65°C),唾液样本(不含漱剂样品)稀释了此种+ Tween-20,所有样本冷冻,然后所有化验是一套完整的最后时间。与先前的报道唾液中的一致15,27,病毒似乎是高度稳定的同时存储温度样本集合类型(图。5中,补充表S10)。为了评估这种关系统计中,我们使用一个似然比的测试框架(参见“方法“详情)来确定存储时间对病毒丰度有显著影响的四个条件。我们没有找到一个显著影响的任何条件在7天时间(假定值:唾液−20°C = 0.52;唾液RT = 0.99;漱口−20°C = 0.09;在RT漱口= 0.65)。然而,盐水漱口样品表现出一个压缩S2 /激增比率(我们解释说明PCR抑制)。然而,病毒颗粒稳定检测在个人和储存条件。
稳定的病毒在唾液和盐水漱口Swab-Seq评估。(一个)病毒的稳定性研究设计的示意图。唾液和盐水漱口样本收集从三个个人和灭活病毒急剧上升,这样最后的病毒基因组PCR将包含250份。样本然后整除,储存在25°C或−20°C表示的时间,此时样品加热和存储。(B- - - - - -E每次课程)Swab-Seq50结果:(B)唾液−20°C, (C)唾液25°C, (D在−20°C)盐水漱口,(E在25°C)盐水漱口。每个测试样本一式三份。每个样本类型(如唾液和盐水漱口),一组负控制样品和第0天样本建立作为存储条件,参考的数据点和负面的第0天控制是精确复制在两个面板。轴表示存储时间天,轴显示S2 /激增比率,和颜色的点表示。
讨论
在这项研究中,我们评估了敏感性、可伸缩性和灵活性Swab-Seq作为潜在的SARS-CoV-2社区监测分析。我们发现的分析灵敏度分析是~ 10份/反应在我们的研究中设置,以及(与以前的结果一致14,21,22)很可能在CLIA环境更加敏感。化验兼容几个酶包和样本类型,包括RNA提取调查(unextracted)唾液和盐水漱口样本。唾液和盐水漱口样本收集方法都很有希望,因为他们需要相对较少的设备或技术集合,是稳定的,至少一个星期在室温下,用最小的预处理和可以作为模板。在我们的测试中,唾液似乎比漱口降低检测极限,可能由于稀释系数和PCR抑制效果中使用的盐水漱口。然而,我们的研究的一个限制是,我们所有的测试包括唾液进行停在病毒粒子,与我们的实验用棉签和盐水漱口的研究对象样本评估临床敏感性和特异性。未来的工作将被要求比较唾液和盐水漱口样品直接NP拭子样本相同的主题。
有几个警告这些结果。首先,虽然Swab-Seq50确实出现在一般分析更敏感比Swab-Seq40在一对一的比较,也似乎容易假阴性和假阳性(主要是在样品检测极限附近)。我们怀疑这可能是用PCR扩增偏差来解释28,虽然我们没有这种直接评估。第二,尽管试验相当streamlined-implementing extraction-free样本收集方法和测序短这一方法并不代表一个快速诊断。经过全面的考虑,我们希望可以过程样品大约在相同的利率标准在诊断实验室环境中存在。Swab-Seq的优点是它可以扩展更多的页码引物和标准热循环,而存在实时热循环试验需要额外的(昂贵的)。然而,Swab-Seq还需要访问一个Illumina公司音序器。此外,详细的在以前的出版物14,29日,Swab-Seq容易受到这种现象被称为“条形码交换”,纳入残余引物的PCR产品在音序器池样本,从而可以换出正确的条形码错误。实验中我们使用了独特的双指数(UDI),因此是最好的动力检测条形码交换,读取的百分比匹配个人条形码代表无效对条形码NextSeq音序器范围从0.03%到2% MiSeq音序器(补充表S11)。虽然NextSeq数据显示较低的交换率,这些样本也汇集与其他库,是稀释在引物的浓度和稀释的浓度目标扩增子从相同的库(相对于扩增子从其他库)。最后,我们强调了使用extraction-free处理样品的方法。这些重要的优势,包括处理潜在的传染性样本有限,在室温下长期稳定,加工速度,和可伸缩性。然而,提取RNA集中样本和删除背景污染物可以抑制PCR。我们观察到一个特定的降低检测极限盐水含漱剂相对于唾液和提取RNA,尽管这个样例类型是最适合缩放处理。尽管如此,在我们看来Swab-Seq保证其使用的优势在促进应对当前流感大流行的和未来的。
展望未来,我们的立场是,Swab-Seq(和化验)将受益于进一步发展的几个修改为更大的适用性和精确性。首先,多路复用分析可能会提高灵敏度,使同时测试多个病毒。这将有助于抗击SARS-CoV-2流行和使更多的常规季节性病毒进行检测。第二,条形码问题交换是一项主要挑战。提出的解决方案包括使用UDI,每个好测序一起收到一个惟一的索引扩增子的两端。这使得识别条形码交换明确,但没有规模well-profiling 384个样本需要1536独特的引物。第二个选择是使用所谓的“semi-combinatorial”索引,即不是所有的引物组合是如何实现的。相反,几套独立的引物可用于组合方式(例如,一组用于板的每个象限)。这增加了整体的多样性相对于组合索引和条形码的数量也会增加无效的组合,可以实现更好的排除一些交换活动。然而,这使得一些条形码交换察觉和仍然有问题scaling-splitting 384分成四组,每个收到独立仍然需要160个引物组合。 A third solution would be to introduce a unique molecular index (UMI) into the library design. This degenerate sequence would be introduced during reverse transcription and would uniquely tag each RNA molecule profiled. Through PCR amplification, proper UMIs would be copied in large quantities. Barcode swapping occurring later on the sequencing instrument, would result in a rare pairing of a UMI and a barcode combination that should be easy to detect and exclude. There are challenges in implementing a UMI in the current 1-step qRT-PCR workflow, but we think this would be an important future improvement. In addition, if the library construction design incorporated a third level of barcoding, 384 samples could reasonably be profiled by 48 primers in one configuration.
在我们评估Swab-Seq监测测试,我们发现分析的实现简单和灵活。至少临床敏感如当前黄金标准分析和可伸缩性方面的潜在优势,灵活性有关样品类型,和成本,现有的技术应该允许快速、经济适应这个测试框架的一个医疗保健设置。虽然有一定的局限性的分析在目前的设计中,可以进一步改进,可以允许更精确、灵活、可扩展的测试在未来。
方法
合成S2激增
中描述的合成S2激增Ref。14。短暂,S2模板cDNA T7启动子和体外转录的条形码被放大1×10获得6合成的副本SARS-CoV-2 RNA(扭转生物科学)使用Luna普遍一步RT-qPCR工具包(与400海里内,# E3005S)的正向和反向引物(补充表中引物序列S12)。循环条件如下:55°C 10分钟,95°C 1分钟,其次是29日周期95°C的10年代和60°C 30年代。合成cDNA分成两个相等的整除,清洗使用Zymo DNA清洁& Concentrator-5工具包(Zymo研究# D4013)或2 x AMPure XP珠子。没有明显差异在凝胶上运行两个分数,结合下游合成整除。500 ng的模板DNA受到体外转录使用内HiScribe T7工具包(内,# 0204 s)后,制造商的指示。反应混合物孵化16 h在37°C。使用RNA合成S2激增是净化清洁& Concentrator-5工具包(Zymo研究# ZR1013)根据制造商的指示和筛选了nuclease-free水。通过量子位激增合成RNA是量化,RNA是运行验证了它的大小与控制S2激增整除(请八分仪提供的生物)6% Novex此种凝胶(表达载体)。
设置稀释实验
连续稀释的合成SARS-CoV-2 RNA(生物科学,# 102019)或heat-inactivated SARS-CoV-2病毒(写明ATCC # vr - 1986港元)RPP30DNA (IDT, # 10006626)刚做好每一个实验。连续稀释为每个实验都做一次,并复制代表额外的整除相同的稀释。稀释都由同一个人完成的。TaqPath比较的互译多元主混合(Thermofisher, # A28521)与卢娜普遍一步RT-qPCR工具包(内,# E3005S),稀释的混合合成病毒RNA和2000份RPP30每个反应制备DNA裂解缓冲Cells-to-cDNA II工具包中包含(表达载体,# AM1722)然后在75°C加热10分钟裁判中描述。14。为稀释实验比较TaqPath互译多路主加入TaqPath CG (Thermofisher # 15299),合成病毒RNA和的混合物RPP30DNA制备nuclease-free水没有供暖。为稀释实验比较病毒RNA病毒颗粒,串行稀释nuclease-free也准备了水没有加热。看到补充表S12在这些实验中使用的引物序列。
从对象中提取RNA的临床诊断
RNA样本收集的对象作为常规监测的一部分测试亚利桑那大学的校园。NP拭子样本收集从上呼吸道系统的每个参与者的监督下卫生保健专业,放入3毫升的CDC配方病毒传输媒体(VTM,https://www.cdc.gov/csels/dls/locs/2020/new_sop_for_creating_vtm.html)。拭子在VTM被储存在4°C到RNA提取,这是执行72 h内所有样本的收集。测试200μL VTM用于使用Zymo RNA提取快速96病毒rna工具包(Zymo研究)和在30μL筛选了。所有样本存储在96孔板与SARS-CoV-2−20°C和测试中存在试验24小时内孤立。所有样品残余存储长期存在测试后−80°C。通过医生总订单所有样本收集。所有的参与者在这个研究,下面描述的样本提供知情同意和批准的这项研究是亚利桑那大学COVID-19数据治理委员会和机构审查委员会。所有的方法都是按照指导方针和有关规定执行。
从对象中存在的RNA提取NP拭子
的SARS-CoV-2存在测试过程使用的是CDC建议的修改协议(https://www.cdc.gov/coronavirus/2019-ncov/lab/virus-requests.html)。试验实现了底漆/探针对2019 - ncoveua - 01诊断面板中提供引物组(IDT)][集成DNA技术,包括人类RNaseP基因(把这里称为“RPP30”)作为一个功能的RNA隔离控制标志。另外,2019 - ncov_n_positive控制(IDT)是在串联运行在每个临床样本的96孔板测试。一步法中存在的反应是使用5μL执行示例运行单独的三个区域(N1,N2和RPP30)的反应总额10μL使用TaqPath™互译RT-qPCR CG大师混合(Thermofisher # A15300)。所有测试都使用了ABI棱镜®7900 ht序列检测系统(应用生物系统公司)和所有样本处理运行包含至少一个积极的,消极的,没有模板控制。切断生周期阈值(Ct)值< 45是用来确定所有3本研究地区的积极性。
4板用于这些分析的样本包括257个样本来自两个来源的无症状或症状的个体(样本ID从“ASYMPT”或“SYMPT”), 125负控制井(表示样本ID的术语“底片”井只含有水,“EXT”井含有RNA提取细胞无毒性,或“非营利组织”IDT 2019 - ncov_n_positive控制N基因控制)和2积极控制样品(“检测”)从池创建之前积极的样本。
准备唾液样本稀释/稳定性实验
稀释/稳定性实验中,受试者为阴性COVID-19唾液样本提供5毫升唾液吐到无菌50毫升锥形管。唾液样本涡前转移100μL 1.5毫升与给定浓度的连续管和混合稀释,heat-inactivated病毒彻底涡流和移液上下。唾液样本与病毒颗粒上升在95°C加热10 - 30分钟,这取决于实验中,1:1稀释100μL 2 x Tween-20此种+ 1%,然后放在冰到下一步。
准备盐水漱口样品稀释/稳定性实验
稀释/稳定性实验中,受试者为阴性COVID-19漱口提供样本,通过倾斜头部,漱口5毫升0.9%氯化钠盐水丸(AddiPak猫# 200 - 59)至少30年代,在吐痰盐水集合管。与唾液样本,样本漱口涡前转移200μL 1.5毫升与给定浓度的连续管和混合稀释,heat-inactivated病毒彻底涡流和移液上下。漱口样本与病毒颗粒上升在65°C加热30分钟或95°C 10 ~ 30分钟,这取决于实验中,然后放在冰到下一步。
漱口样本对象为临床诊断
盐水漱口样本收集的对象作为常规监测的一部分在大学校园。受试者提供5毫升丸无菌生理盐水0.9%氯化钠(AddiPak猫# 200 - 59)和无菌收集管和指示的盐水漱口头部倾斜30年代后跟5 s嗖嗖声,反复3次沉淀收集管和限制的解决方案示例。立即样本存储在4°C。在转移到诊断实验室,样本在65°C的环境了30分钟,然后储存在−80°C,直到准备测试。样本收集下医生总订单。
中存在的盐水漱口样本主题
所有样品都测试一式三份的卷10μL使用CDCN1和RPP30使用相同的存在试剂和底漆/探针集条件描述NP拭子,除了样本放大40周期而不是45。
Swab-Seq PCR反应
Swab-Seq反应进行了卢娜一步普遍RT主混合或混合两种TaqPath大师(多路复用或CG)在20μL反应体积使用(Bio-Rad) thermocycler C1000联系。一步掌握组合包含500份S2激增/反应和卢娜或TaqPath试剂被分发到每个PCR板。接下来,引物的混合编码S2(400海里最终浓度为引物)RPP30(50 nM最终浓度为)引物被添加到每个。5μL模板添加到相应的井作为最后一步之前运行PCR。盘子是密封光学海豹(Bio-Rad # MSB1001)。每个rt - pcr试剂盒的自行车资料如下:
卢娜工具包:55°C 10分钟,95°C 1分钟,40 95°C的周期10年代和60°C 30年代。
TaqPath多路复用:25°C 2分钟,10分钟53°C, 95°C 2分钟,40或50周期95°C的10年代和60°C 30年代。
TaqPath CG: 25°C 2分钟,15分钟50°C, 95°C 2分钟,40或50周期95°C的3 s和60°C 30年代。
存在其他的样品
确认受试者负稀释/稳定性实验中,一些样品受到中存在。这些样本的存在是对TaqPath互译多元混合和大师N1Taqman探针(IDT # 10006713) 20μL反应体积使用它连接实时PCR系统(Bio-Rad)。5μL模板加载/反应。热循环概要文件是Swab-Seq上面描述的一样。
测序的纯化和制备图书馆
Swab-Seq PCR产品被转移5μL汇集每进入一个新的1.5毫升管然后清理2 x AMPure XP珠子(贝克曼库尔特,# A63881)。一卷样品池和两卷的珠子和孵化5分钟在室温下混合,随后将样品管在磁站~ 2分钟将珠子和解决方案。上层清液被丢弃。愿望之后,珠子和200μL 80%乙醇洗两次磁站,在室温下干燥5分钟,然后从磁站在20μL resuspended EB缓冲区(试剂盒,# 19086)。孵化2分钟后,样品被放置在磁站单独额外的珠子~ 2分钟。然后将上层清液转移到一个新的管。筛选了DNA的浓度决定利用量子位1×dsDNA海关化验设备(表达载体,# Q33231)。而Swab-Seq库被发现产生可变带大小在图书馆、大乐队的大小(200个基点),代表S2 PCR产物,验证了通过运行6% Novex此种凝胶(补充图。S9)。我们假设平均库大小为200 bp当ng /μL浓度转换成一个摩尔浓度使用以下方程:(浓度在ng /μL) /(660克/摩尔×平均库大小bp)×106在海里=浓度。
凝胶成像设备和设置
PCR凝胶成像的产品,每个产品1μL加载在一个1加上1××此种凝胶橙色凝胶装入染料(内,# B7022S)和运行在180 V 30分钟。50 ng 100个基点的DNA梯(内,# N3231S)也与样品作为标准运行。跑后,凝胶染色1×SYBR黄金(表达载体,# S11494) 1×此种缓冲大约1分钟和成像通过蓝光System-BL Axygen凝胶文档。补充图。S9的凝胶被暴露在蓝光1 s和143 ms。
测序
测序,净化图书馆刚稀释与EB 2 nM缓冲区,然后10μL图书馆与10μL变性0.1 N在室温下孵化的氢氧化钠5分钟。添加980μL HT1缓冲区(Illumina公司)停止了变性和稀释图书馆20点。PhiX (Illumina公司)控制(当Swab-Seq并不与其他样品池类型)也准备晚上20点同样,变性库(60%)和PhiX控制(40%)混合在这些实例。库被进一步稀释8点(MiSeq v2工具包),下午15点(MiSeq v3工具包),或1.5点(中期NextSeq工具包)HT1缓冲区。
自定义为Read1引物和Index1(也称为“i7”)(和Index2(也称为“i5”),如果测序NextSeq)飙升到标准的底漆水库。100年MiSeqμL水库12个引物混合物的混合6.8μL定制Read1底漆的混合物(50µM S2和底漆RPP30),然后替换在水库12 MiSeq试剂盒。S2和i7引物混合物RPP30被加载到水库13用同样的方法。NextSeq 500/550, 52μL Read1底漆的混合物(每个引物的最终浓度10 uM)加载到水库22。此外,85μL i7引物混合物(10μM每个引物)和85年μL i5底漆的混合物为每个引物(10µM)加载到水库23。除非容纳其他库类型在相同的运行,测序运行26读周期1和10个周期的每个索引。看到补充表S11测序结果的总结。注意,我们re-sequenced一个示例中,“Saliva_dilutions_96_vs_384”,由于低聚类在音序器的三倍。这里有三个运行进行分析相结合。
分析
从测序BCL文件被转换为fastq文件在本地个人电脑使用Illumina公司bcl2fastq程序实现为一个码头工人形象(https://hub.docker.com/r/genomicpariscentre/bcl2fastq2)。因为一些样品是多路复用与其他实验中,我们开发了一个自定义python脚本分工fastq文件的基础上,将条形码而占其他条码测序运行同样允许测序条形码和预期之间的汉明距离为1 /可能的条形码。为了一致性,甚至实验测序独自去复用这个管道。多路分解脚本可以从github库(https://github.com/cusanovichlab/swabseq)。板地图生成,然后转换成使用代码的样例表可以从八分仪生物的github库(https://github.com/octantbio/SwabSeq)。元数据文件和白名单的条形码生成使用以前公布的预处理方法29日(Swab-Seq Jupyter笔记本中概述的分析https://github.com/pachterlab/BLCSBGLKP_2020/blob/master/notebooks/swabseq.ipynb)。Fastq文件被映射到一个自定义的引用,其中包括S2,在控制和RPP30序列和总结样本条形码使用“Swabseq10”命令可用的“covid”分支kallisto Github库(https://github.com/pachterlab/kallisto.git)以及bustools30.,31日。我们实现这些步骤在一个码头工人本地容器。
后生成一个数据矩阵,提供读计数为每个记录分配给每个样本条形码,我们计算S2 /激增在R比率和可视化数据通过修改脚本的八分仪Github库。我们提供我们的脚本数据处理和可视化作为一个Github库(https://github.com/cusanovichlab/swabseq)。从无症状的个体样本收集(提取RNA和盐水漱口),我们获得的平均Ct值三个存在复制从父学习。我们认为任何样本检测Ct值正的。Swab-Seq40,我们设置了S2 /激增比积极的样品在0.01。对于Swab-Seq50,我们设置阈值为0.002。所有滴定和稳定性实验组合条形码,这样的每个板收到i5和i7条形码的独特组合,所有样本都包括在下游分析无论测序深度。所有与UDI涉及主题样本进行了实验,这意味着每一个好评一个独特的i5条形码和一个独特的i7条形码。这些UDI引物请八分仪提供的生物。至少有500只样品读取映射到S2或者激增控制被认为是在下游分析试验样品。虽然Ct值代表一式三份的平均值,S2 /激增比率值确定没有复制。
诊断测试的比较
对比诊断试验是评估的正面/负面百分比协议和科恩kappa系数24。95%置信区间在积极的协议和消极的百分比协议百分比指标计算使用Ref的“威尔逊”方法。25。科恩kappa系数和95%置信区间计算使用“科恩。卡巴”功能的“心理”包32在r为指标,包括不确定的样本作为第三类。
似然比的测试框架对病毒的稳定性
为了统计评估病毒颗粒的稳定存储在各种条件,我们采用了一个似然比的测试框架使用固定效应线性模型。即我们符合线性模型的数据(Y =μ+βX +αZ +ε,Y代表Swab-Seq估计病毒丰度(假定为正态分布),“μ”代表的意思是丰富,“X”表示存储的持续时间(0、1或7天作为数值),“β”表示存储时间的影响,“Z”代表了个人测试(因为有三个科目包括在这项研究),“α”表示个人的影响意味着病毒丰度,并“ε”表示残差。嵌套模型不包括术语存储时间(即Y =μ+αZ +ε)。日志可能计算与“logLik”和“lm”函数R,并假定值计算基于χ从测试统计数据2分布在两个自由度。
数据可用性
基因表达数据为本研究可从生成综合(GSE176224)。
引用
ProMED-mail。确诊Pneumonia-China(湖北):请求信息。ProMED-mail 2019。20191230.6864153 (2019)。
吴,F。et al。一种新的冠状病毒与人类呼吸道疾病在中国。自然579年,265 - 269 (2020)。
武汉冠状病毒诊断检测实时RTPCR 2019年。https://www.who.int/docs/default-source/coronaviruse/wuhan-virus-assay-v1991527e5122341d99287a1b17c111902.pdf?sfvrsn=d381fc88_2。2021年5月15日通过。
Srivatsan, S。et al。SwabExpress:端到端的协议extraction-free COVID-19测试。bioRxivhttps://doi.org/10.1101/2020.04.22.056283(2020)。
王,W。et al。检测SARS-CoV-2在不同类型的临床标本。《美国医学会杂志》323年,1843 - 1844 (2020)。
麦克菲DRB。COVID-19实验室检测/美国疾病控制与预防中心的指导方针。说我的。66年101067 (2020)。
世界卫生组织。2019实验室检测冠状病毒病(COVID-19)疑似人间病例:临时指导,2020年3月2。世界卫生组织;2020年。报告没有。:WHO/COVID-19/laboratory/2020.4.https://apps.who.int/iris/handle/10665/331329。2021年5月15日通过。
Koskinen,。et al。并发症COVID-19鼻咽拭子测试。JAMA Otolaryngol。头颈外科杂志。https://doi.org/10.1001/jamaoto.2021.0715(2021)。
帕尔马斯G。et al。鼻拭子作为首选临床标本COVID-19儿童的测试。Pediatr。感染。说。J。39e267 (2020)。
卡拉汉,C。et al。Nasal-swab测试了SARS-CoV-2低病毒载量患者。medRxivhttps://doi.org/10.1101/2020.06.12.20128736(2020)。
威利,a . L。et al。唾液或SARS-CoV-2鼻咽拭子标本进行检测。心血管病。j .地中海。383年,1283 - 1286 (2020)。
摩尔,C。,Corden, S., Sinha, J. & Jones, R. Dry cotton or flocked respiratory swabs as a simple collection technique for the molecular detection of respiratory viruses using real-time NASBA.j .性研究。方法。153年,84 - 89 (2008)。
Ranoa d·r·E。et al。唾液分子测试SARS-CoV-2绕过RNA提取。bioRxivhttps://doi.org/10.1101/2020.06.18.159434(2020)。
布鲁姆JS,萨瑟L, Munugala C,琼斯EM, Gasperini M, Lubock NB,et al。Swab-Seq:高通量大规模扩大SARS-CoV-2测试平台。medRxiv。2020.08.04.20167874 (2021)。
Vogels, c·b·F。et al。SalivaDirect:简化和灵活的平台提高SARS-CoV-2测试能力。地中海(N Y)。2,263 - 280。e6 (2021)。
大卫•帕。,Zheng, A. & Walensky, R. P. Assessment of SARS-CoV-2 screening strategies to permit the safe reopening of college campuses in the United States.JAMA Netw开放。3e2016818-e2016818 (2020)。
Hellewell, J。,Russell, T. W., The SAFER Investigators and Field Study Team, The Crick COVID-19 Consortium, CMMID COVID-19 working group, Beale, R.,et al。估计的有效性常规无症状的PCR检测的测试在不同频率SARS-CoV-2感染。medRxiv。2020.11.24.20229948 (2020)。
Larremore, d . B。et al。测试灵敏度是次要为COVID-19监测频率和周转时间。medRxivhttps://doi.org/10.1101/2020.06.22.20136309(2020)。
赢了,J。et al。发展laboratory-safe和低成本检测协议SARS-CoV-2冠状病毒病的2019 (COVID-19)。经验,一般人。29日,107 - 119 (2020)。
Kliff,美国亚利桑那州“不知所措”与美国需求测试系统显示压力。《纽约时报》,2020年6月25日。https://www.nytimes.com/2020/06/25/upshot/virus-testing-shortfall-arizona.html。(2021年5月29日通过)。
美国食品和药物管理局。紧急使用授权(欧洲大学协会)总结螺旋COVID19门店测试。https://www.fda.gov/media/140917/download。访问2022年1月13日。
美国食品和药物管理局。紧急使用授权总结UCLA SwabSeq COVID-19诊断平台。https://www.fda.gov/media/142805/download。访问2022年1月13日。
中心设备、放射卫生。政策COVID-19测试期间突发公共卫生事件(修订)。2020年。https://www.fda.gov/regulatory - information/search fda -指导- documents/policy冠状病毒疾病- 2019测试- -公共卫生紧急修改。2021年5月29日访问。
科恩,j .协议名义尺度系数。建造。Psychol。量。20.,37-46。https://doi.org/10.1177/001316446002000104(1960)。
奥特曼,D。统计数据与信心:置信区间和统计的指导方针(威利,2000)。
Griesemer, s B。et al。评估唾液标本类型和稳定SARS-CoV-2测试的解决方案。j .中国。Microbiol。https://doi.org/10.1128/JCM.01418-20(2021)。
奥特,i M。et al。只是唾液:稳定否定SARS-CoV-2检测需要昂贵的设备集合。medRxivhttps://doi.org/10.1101/2020.08.03.20165233(2020)。
傅,Y。,Wu, P.-H., Beane, T., Zamore, P. D. & Weng, Z. Elimination of PCR duplicates in RNA-seq and small RNA-seq using unique molecular identifiers.BMC染色体组。191 - 14 (2018)。
新浪Booeshaghi, A。et al。从高度可靠和准确的诊断多路复用测序分析。科学。代表。101 - 7 (2020)。
布雷:L。,Pimentel, H., Melsted, P. & Pachter, L. Near-optimal probabilistic RNA-seq quantification.生物科技Nat。》。34,525 - 527 (2016)。
Melsted, P。等。模块化和高效的单细胞RNA-seq预处理。bioRxivhttps://doi.org/10.1101/673285(2019)。
雷夫尔,w .程序心理、心理和人格研究[R包心理版本2.1.9]。2021年https://CRAN.R-project.org/package=psych。访问2022年1月13日。
确认
我们要感谢美国Kosuri m . Gasperini j .布鲁姆和e·汤普森的八分仪生物试剂的慷慨的分享和建议。我们还要感谢“Covid测试按比例增长”的成员松弛工作组提供丰富的信息。我们感谢j . Galina-Mehlman和b Fransway亚利桑那大学的遗传学核心的慷慨援助。我们感谢d间谍他宝贵的援助与集装箱码头工人。谢谢拉弗勒尔b, d . Billheimer和r .经脉的建议和有益的讨论。研究报告在这个出版得到了医学科学研究所的美国国立卫生研究院在奖R35GM137896 (D.A.C.)。
作者信息
作者和联系
贡献
香港和D.A.C.构思、设计和执行实验,分析数据,并写了手稿。S.A.响亮的帮助评估中存在的分析,设计和执行的稳定性测试。R.S.试剂提供样品,和辅助实现盐水漱口的协议。郭台强和M.H.帮助构思和设计的稳定性测试和解释结果。M.H.写了手稿。所有作者回顾和批准了手稿。
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
出版商的注意
施普林格自然保持中立在发表关于司法主权地图和所属机构。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
康,H。,一个llison, S., Spangenberg, A.et al。Swab-Seq评估作为一个可伸缩的、敏感分析社区SARS-CoV-2感染的监测。Sci代表123047 (2022)。https://doi.org/10.1038/s41598 - 022 - 06901 - 5
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598 - 022 - 06901 - 5