摘要
基因表达、DNA甲基化及其组织关系通常在肺鳞状细胞癌(LUSC)中发生改变。为了阐明这些复杂的相互作用,我们分别通过部分信息分解和逆相关算法重建了差异表达基因网络和差异甲基化胞嘧啶(DMC)网络。然后进行图并,对网络进行整合。整合网络的群落检测和富集分析揭示了LUSC中细胞周期、角化、免疫系统和异种代谢基因集之间的密切相互作用。DMC分析表明,低甲基化靶向负责细胞周期、角化和NRF2通路的基因集。另一方面,高甲基化基因影响循环系统发育、免疫系统、细胞外基质组织和纤毛组织。通过中心性测量,我们发现NCAPG2、PSMG3和FADD是与其他节点高度连接的枢纽基因,可能在LUSC基因调控异常中发挥重要作用。我们还发现,中间中心性高的基因比中间中心性低的基因更容易影响患者的生存。这些结果表明,综合网络分析使我们能够全面了解LUSC的相互作用和规则。
简介
肺鳞状细胞癌(LUSC)是仅次于肺腺癌的第二大常见肺癌亚型,占所有肺癌诊断的20%1.其特征是肺上皮细胞角化和/或细胞间桥2.突变和表观遗传异常的逐渐积累是常见的,并驱动LUSC的进展3..LUSC研究的进展揭示了TP53、PI3KCA、FGFR1等基因异常在LUSC发病和治疗中的作用4.对LUSC的表观遗传学研究也发现了癌症的重要驱动因素,如NFE2L2、SOX2和TP63的甲基化5,6.然而,只有少数研究对LUSC中这些驱动因素之间的组织和层次相互作用进行了探索和分析。研究这些基因及其调控因子之间的相互作用对于了解LUSC的发病机制和帮助治疗LUSC至关重要。
基于网络的建模是分析变量之间相互作用的强大方法。网络或图是由边(或链接)连接的顶点(或节点)组成的数学结构。顶点和边可能有一些属性来描述它们的特征。基于网络的模型已用于研究基因和胞嘧啶甲基化的关系。例如,一项针对白血病的网络研究发现,基因表达和甲基化都持续影响Ras、PI3K-Akt和Rap1信号通路7.另一项研究通过整合甲基化数据和蛋白质-蛋白质相互作用网络,确定了新的癌症相关途径8.这些研究使用了从开源数据库中获得的网络。
在我们的研究中,我们计算重建和整合了差异表达基因(DEG)网络和差异甲基化胞嘧啶(DMC)网络。这种方法的优点是,它使我们能够找到现有数据库中没有包含的新的交互作用。该综合图可以提供癌症中基因-基因和甲基化-基因相互作用的蓝图。通过分析图的拓扑结构,我们可以获得很多信息,例如通过中心性测量来识别重要的调控基因7,9并利用群体检测分析对相似节点进行聚类10.通过整合DMC网络和DEG网络,我们可以同时研究细胞系统在基因和甲基化水平上的协调性。我们的分析可能为LUSC中新的相互作用和核心调控基因的鉴定提供基础。
结果
网络的特点
利用基因组数据公共-癌症基因组图谱肺鳞状细胞癌(GDC-TCGA-LUSC)数据集的数据,我们进行了基因表达和胞嘧啶甲基化的差异表达分析。然后,我们使用部分信息分解和上下文(PIDC)和甲基化/表达关系增强子链接(ELMER)算法分别重建了DEG和DMC网络。简而言之,部分信息分解是将基因之间的相互信息分解为唯一的、冗余的和协同的成分。PIDC将基因之间的关系计算为独特成分的平均比例。然后,PIDC将返回基因与其秩之间所有可能的边。DEG网络中的边代表了PIDC排名中最高的1%。对于甲基化网络,ELMER算法为每个DMC选择最近的10个上游基因和最近的10个下游基因。然后,检测DMC甲基化与基因表达的负相关关系。DMC网络中的边表示显著的(调整后的-\(p < 0.01\))逆相关。接下来,我们取两个图的并集,并提取图的巨大组成部分。图中的流程图。1总结了本研究的分析步骤。
最终的综合网络有9748个节点和228246条边。在9748个节点中,7903个被鉴定为deg,其余1845个为DMC探针。这些边由224149条基因-基因边和4097条探针-基因相互作用边组成(3369条低甲基化边和728条高甲基化边)。2).我们在补充表中列出了所有的节点和边S1而且S3.
社区认同分析
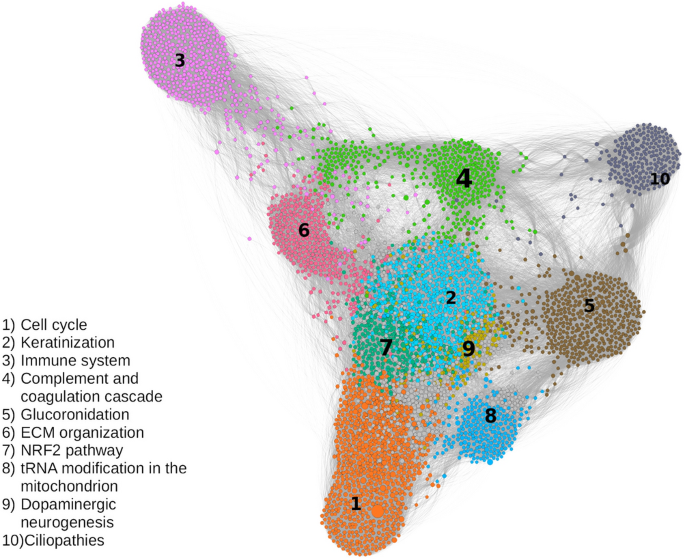
使用莱顿算法10,我们确定了10个最大的社区,它们至少有200个节点,占总节点的大约50%。我们根据社区中的节点数量对每个社区进行命名和排名(例如,最大的社区是社区1,第二大社区是社区2)(图2)。3.).基因集富集分析揭示了各群落的功能类。例如,最大的群落群落1,主要包括DNA复制和细胞周期的基因。第2、3和4个最大的群落分别包括负责角化、免疫系统和补体-凝血级联途径的基因。我们在表中列出了10个最大的社区及其一些功能类1.完整的清单见补充表S3.
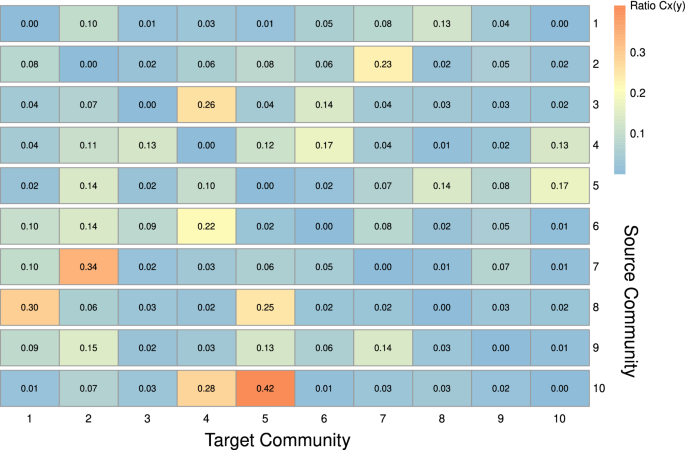
数字3.帮助可视化社区之间的交互。我们使用ForceAtlas2作为网络布局算法来在二维图像中显示网络12.ForceAtlas2是一种力导向的布局算法,其中节点像带电粒子一样相互排斥,而边缘像弹簧一样吸引节点。在这个算法中,社区之间的交互越强,它们就越亲密。例如,社区3与社区6和社区4的关系比与社区8的关系更密切。为了定量地测量相互作用的强度,我们计算了\ (cx (y) \),这是社区之间连接的链接的比率x而且y等于“社区”上的社区间链接总数x.社区间链接是连接一个社区到另一个社区的链接。数字4显示比例热图\ (cx (y) \)来自源社区x针对目标群体y.我们以Community 3为例。如热图所示,第3行Community 3的大多数社区间链接连接到Community 4(26%)和Community 6(14%)。的显著性\ (C_ {x} (y) \)使用网络随机化测试。我们发现\ (C_ {x} (y) \)图中的值。4不是随机的(补充表S4).
中心性测度分析
采用中心性测度分析方法研究了节点的作用及其对网络的影响。有各种各样的中心性度量方法,如度中心性、接近中心性和中间中心性。在这里,我们关注的是中介中心性。节点的中间中心性度量通过该节点的最短路径的数量。在基因调控网络中,有时系统中最重要的节点并不是边缘数量最多的节点,而是连接群体的中间人或对信息流拥有最大控制权的节点。中介中心性衡量的是一个节点对信息流的影响程度,其数学公式如下:
在哪里\ (u \)是一个节点,\ \(σ_{圣}\)节点间最短路径的总数是多少\ (\)而且\ \ (t),\ \(σ_{圣}(u) \)节点间最短路径的数量是多少\ (\)而且\ \ (t)传递节点\ (u \).中间中心性已被用于鉴定对白血病患者生存有高度影响的基因7以及乳腺癌细胞的核心调节因子9.我们计算每个节点的中间中心性(补充表S1).中间中心性最高的三个基因是NCAPG2、PSMG3和FADD。
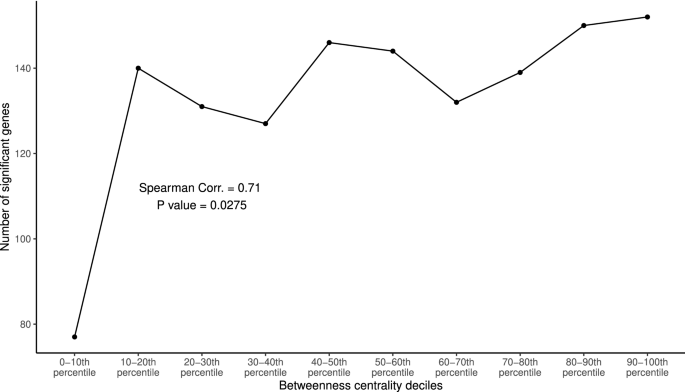
在我们的研究中,我们调查了影响生存的基因数量与中间中心性之间的相关性。我们根据中间中心性的十分位数将基因分为10组。然后,我们对每个基因进行单变量Kaplan-Meier生存分析(补充表S5).具有fdr调节-的基因\(p < 0.25\)被认为对生存有显著影响。我们发现正相关(斯皮尔曼相关系数\(\rho = 0.71\),\(p = 0.075 \)影响生存的基因数量与中间中心性之间的关系(图;5).
甲基化分析
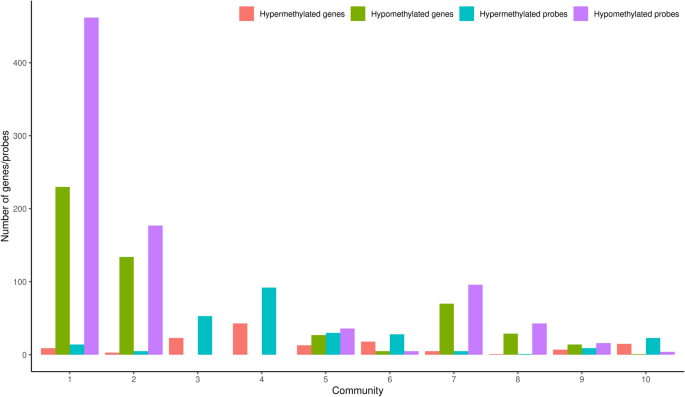
在我们的网络中有1842个低甲基化基因和313个高甲基化基因。低甲基化基因和探针在群落1、2、7和8中占主导地位,它们分别调控线粒体中的细胞周期、角化、NRF2通路和tRNA修饰。相比之下,高甲基化基因和探针在群落3(免疫系统)、4(管发育和血管形态发生)、6(循环系统发育和细胞外基质(ECM)组织)和10(纤毛运动和组织)中占主导地位(图3)。6).
当一组dmc在特定的样本子集中充当增强子或沉默子时,这通常是上游主调控转录因子(TF)改变的结果。5.通过使用get。富贵。母题和get。tf在ELMER包中的功能,我们鉴定了LUSC中甲基化变化的富集基序和主要调控因子。对于低甲基化探针,富集最多的基序是FOSL2、FOSB、FOSL1和FOS。我们发现ZNF74、TP63、KLF5、TFAP4和ZFP64是低甲基化基因的主调控因子。对于高甲基化探针,ZBT14、E2F2、SP1和SP2是最富集的motif, CREB3L1、CXXC5和ETS1是主调控子。我们还调查了每个社区的tf。只有至少有10个DMC探针连接到它的社区才会被考虑(表2).全球和社区层面的所有顶级丰富主题和主调控因子tf都列在补充表中S6而且S7,分别。
讨论
在这项研究中,我们进行了基于网络的建模,以研究LUSC中基因组和甲基组组谱之间的相互作用。由于甲基化和基因表达数据的性质不同,我们使用了两种不同的方法来重建DEG和DMC网络,分别是PIDC和ELMER算法。PIDC和ELMER算法经过了良好的测试,并已在许多研究中用作网络推理方法5,13,14.
许多网络推理方法,如GENIE315,偏相关16,和真诚17,也可以重建DEG网络。然而,我们选择了PIDC,因为它有很强的数学背景,可以检测噪声或非线性关系,速度相当快,并且不需要时间序列数据来重建基因网络13,18.PIDC算法采用多元信息测度来识别基因之间的关系。信息度量可以量化变量之间的依赖关系,而无需对依赖关系的性质进行假设19.对于通常在癌症转录组数据集中看到的噪声和非线性关系,它是理想的。PIDC将变量之间的信息分为冗余、惟一和协同类别。通过这样做,PIDC可以区分组内一对变量的唯一信息和多个变量共享的冗余信息18.PIDC算法已被证明比其他网络推理方法具有更高的准确性、稳定性和效率13.PIDC的局限性在于它不能提供关于边缘方向或关系效应(例如,抑制或兴奋)的信息。我们建议需要边缘方向和节点符号信息的研究人员使用其他网络推断方法(例如,用于有向图的GENIE3或用于有符号网络的部分相关)。在真实的临床环境中,PIDC已被用于研究急性淋巴细胞白血病的调节网络20.其中,t细胞来自SARS-CoV-2患者21,以及接受内分泌治疗的乳腺癌22.
ELMER使用肿瘤中顺式调节模块的甲基化变化作为DMC网络的中心枢纽。然后,通过相关分析将其与上游调控因子tf和下游靶基因进行关联23.因此,ELMER不仅可以重建甲基化网络,还可以推断与甲基化基序结合位点结合的主调控因子tf。ELMER算法已在一些研究中用于调查许多癌症的甲基化情况5,14.它也被用于甲状腺癌转录因子分析的临床和实验研究24鳞状细胞癌25脑膜瘤,26,以及早衰症综合症27.
通过对DEG网络和DMC网络进行图并,实现了网络层面的集成。我们通过群落识别、中心性测量和基因集富集分析来发现集成网络中的关系模式。
社区检测分析揭示了子网社区,同一社区中的节点之间的交互作用强于不同群体中的节点。使用功能富集分析,我们发现每个群落都针对特定的生物过程或途径。DNA复制、细胞周期、ECM组织和免疫系统通路是许多癌症中常见的改变通路28.角化、纤毛组织和表面活性剂代谢是LUSC和肺细胞的特征。补体-凝血级联途径反映了补体在调节肿瘤微环境中的重要性29,30.LUSC发生凝血功能障碍的风险31.我们假设在LUSC中主要群落的基因严重失调。
另一个重要的发现是,10个主要群落中有2个与解毒相关途径有关:群落5与葡萄糖醛酸化途径有关,群落7与NRF2和谷胱甘肽代谢途径有关。葡萄糖醛酸化和谷胱甘肽代谢途径与代谢异种生物的II相酶有关32.NRF2通路是抗氧化反应的主要调节因子33.癌症的主要危险因素,吸烟,可能为解毒相关途径改变的原因提供了解释34.解毒相关途径的改变也有助于增加耐药性35,36.
网络可视化如图。3.揭示了不同群落在LUSC中的相互作用。然后,我们使用了连接这个术语\ (x - y \)来描述社区之间的互动x而且y.例如,我们探讨了社区2和7或连接2-7之间的关系。群落2影响角化,群落7在解毒中起作用,如NRF2和谷胱甘肽代谢途径。在无花果。3.,我们可以看到Community 2的位置与Community 7非常接近。大多数社区间的联系(% \ \ 23日下午(\ \))在社区2连接到社区7。反之亦然。大部分社区7的社区间联系(34个\ % \ \下午(\))连接到社区2。这表明了两个社区之间的密切互动。在Ishitsuka的一篇写得很好的评论中et al。,他们讨论了NRF2在角化中的广泛重要性33.他们指出,KEAP1/NRF2通路在角化、鳞状上皮组织外部反应和解毒的调节中起着重要作用。基于我们的发现,我们假设连接2-7中大量的边表明肺上皮细胞对外界或毒性刺激的反应能力失调。研究表明NRF2缺乏会导致化学致癌物易感性,这也支持了这一点37吸烟会改变NRF2和谷胱甘肽通路34,38.
另一个例子是免疫系统的Community 3、补体-凝血级联的Community 4和ECM组织的Community 6之间的相互作用。的比率\ (C_3 (4) \)而且\ (C_3 (6) \)分别为0.26和0.14。补体系统是先天免疫和凝血系统的关键角色之一。另一方面,免疫系统细胞,如肿瘤相关巨噬细胞(TAMs)和肿瘤相关中性粒细胞(TANs),在重塑肿瘤ECM中起着主要作用39,40.已经开展了许多研究来调查肺癌中的ecm -免疫细胞-补体相互作用。补体C1q蛋白可以激活和招募tam41.肺泡TAMs中C9下调,导致肺癌进展42.巨噬细胞还可以调节c3独立的C5a的生成,从而促进鳞状癌的发生43.其他关于TANs的研究表明,C5a可以通过刺激白三烯B4的释放和IL-1的产生来招募TANs44,45.此外,c3ar依赖性的中性粒细胞细胞外陷阱(NETs)也可积聚TANs46.有研究表明,肺癌细胞中C3a和C5a的浓度和表达高于正常肺细胞47,48.
下一步是使用中心性测量来识别高度连接的节点/基因或中心基因。在图论中,与去除非枢纽基因相比,去除网络中的枢纽节点会增加不可达节点组的比例。因此,枢纽节点对于维护全球网络结构非常重要。在许多生物中,去除枢纽基因比去除非枢纽基因更可能致命。这就是所谓的中心性-致命性规则49,50.中心性度量的例子有度中心性、接近中心性和中间中心性。研究表明,中介中心性和程度中心性比紧密中心性更适合于识别枢纽基因9,51.我们统计了在每十分位数的中间中心性中显著影响生存的基因的数量。我们使用FDR-adjusted-\(p < 0.25\)作为生存分析的排斥阈值。这是因为我们想要找到相对于基因表达数据固有的噪声有适度生存差异的相关基因。我们发现影响存活的基因数量与中间中心性呈正相关(图2)。5).这一结果支持了中心性-致命性规则。在我们的网络中,中介中心性高的基因表达比中介中心性低的基因表达更有可能影响LUSC患者的生存。
NCAPG2、PSMG3和FADD是我们整合网络中中介中心性最高的三个基因。NCAPG2蛋白是凝聚蛋白II复合体的一个亚基,在有丝分裂染色体的组装和分离中起作用。上调NCAPG2促进肺癌细胞增殖52.PSMG3是一种伴侣蛋白,促进20S蛋白酶体的组装。据作者所知,还没有论文研究PSMG3失调对LUSC的影响。而PSMG3的反义长非编码RNA PSMG3- as1在LUSC中高表达,其抑制降低了侵袭性53.在我们的研究中,我们发现PSMG3的表达与患者的生存期显著相关(补充表S5).fas相关死亡结构域蛋白(FADD)传递死亡受体传递的凋亡信号。非小细胞肺癌细胞释放FADD与侵袭性和转移性相关54.在我们的研究中,MYADM是另一个被发现具有高中间中心性并与生存率相关的基因。MYADM是最小的P在Kaplan-Meier生存分析的前10百分位基因的中心性值。它在内皮性炎症反应中调节质膜和皮层细胞骨架之间的连接55.它还有助于平滑肌改变肺动脉高压和结核病气管支气管狭窄56,57.然而,它在肺癌中的作用还没有得到很好的研究。PSMG3和MYADM的进一步研究可能使它们成为潜在的LUSC预后标志物或治疗靶点。
该网络的甲基化分析显示,低甲基化探针靶向细胞周期(Community 1)、NRF2和谷胱甘肽代谢通路(Community 7)、角化(Community 2)和线粒体通路中的tRNA修饰(Community 8)。在许多研究中,这些通路的上调与侵袭性、治疗耐药性、吸烟和预后不良有关28,34,36,58,59.相比之下,高甲基化DMC探针下调了与癌症抑制和组织正常发育/分化相关的途径,如免疫系统(Community 3)、导管发育和血管形态发生(Community 4)、循环系统发育和ECM组织(Community 6)和纤毛组织(Community 10)。
主调控因子tf的获得(对于低甲基化探针)或丢失(对于高甲基化探针)可以改变dmc的甲基化状态。我们使用ELMER包来识别这些上游主稳压器tf。在我们的研究中,我们发现TP63、KLF5和SOX2都过表达,并在全球和群体水平上成为低甲基化探针的顶级tf。这一结果得到了先前染色质免疫沉淀测序研究的支持,该研究发现TP63、SOX2和KLF5是决定食管鳞状细胞癌染色质可及性、表观遗传修饰和基因表达模式的核心调控因子60.相反,我们发现CXXC5和FOXP1的抑制表达是高甲基化的调节因子。CXXC5是一种核锌指蛋白,由DNA甲基转移酶、DNA去甲基酶、组蛋白甲基转移酶和组蛋白去甲基酶组成,通过优先结合未甲基化的CpG岛来参与转录调控61.CXXC5是Wnt/ β -连环蛋白通路的负反馈调控因子62以及促进tgf - β诱导的细胞周期阻滞的肝癌抑制剂63;此外,它是DNA损伤诱导的p53激活所必需的64.FOXP1是一个TF,属于叉头箱家族的P亚家族。FOXP1是一种前列腺癌抑制因子,可调节雄激素受体和FOXA165.FOXP1还与肺癌患者生存率的提高有关66.
综上所述,我们的所有研究结果表明,整合DMC网络和DEG网络有可能揭示基因及其调控因子(例如,tf和甲基化胞嘧啶)之间的复杂相互作用。我们的分析工作流程不仅可以用于LUSC,也可以用于其他癌症和疾病。我们相信,更深入地了解基因调控网络的整体组织结构将有助于LUSC的诊断和治疗管理。
方法
利用PIDC对LUSC中的DEG网络和DMC网络进行了计算重构18和ELMER算法23,分别。然后,结合DEG网络和DMC网络,提取图的巨大分量。该图由代表基因或甲基化探针的节点和代表调控连接的无向边组成。
肺癌数据集的准备
502个LUSC原发肿瘤样本和49个正常组织样本的HTSeq-FPKM-UQ基因表达数据使用Bioconductor包TCGAbiolinks从NCI基因组数据共享(GDC)数据门户网站下载67.使用Silva等人的TCGAbiolinks工作流处理基因表达数据。68.简而言之,我们通过使用tcgaanalyze_预处理函数执行阵列-阵列强度相关性来去除异常值、失败的杂交或错误的样本。接下来,我们分别使用TCGAanalyze_Normalization和TCGAanalyze_Filtering函数对mRNA转录本进行归一化,并过滤样本中低信号的基因。通过TCGA分析和dea分析,筛选出两倍差异表达的基因。然后,我们表演了日志(1 + x) \ \ ()所选基因表达的转化和标准化。
使用ELMER包中的getTCGA函数从相同样品中下载基于Illumina Infinium HumanMethylation450 (HM450) BeadArray平台的TCGA 3级DNA甲基化数据23.
网络重建
采用PIDC算法对DEG网络进行重构。例如,在PIDC中,我们检查了每一个基因三元组,\ (Source_1 \),\ (Source_2 \),目标.PIDC算法将信息划分为\ (Source_1 \)而且目标分为三类:冗余性、独特性和协同性。冗余信息是关于的那部分信息目标,要么\ (Source_1 \)或\ (Source_2 \)可以提供。唯一的信息来自唯一的信息\ (Source_1 \)或\ (Source_2 \)只有。协同信息是来自双方的信息的一部分\ (Source_1 \)而且\ (Source_2 \).然后,两者之间的直接函数关系\ (Source_1 \)而且目标是用唯一信息之间的平均比例来计算的吗\ (Source_1 \)而且目标对所有\ (Source_2 \)18.
我们使用了在NetworkInference.jl包中实现的PIDC算法18.分别采用贝叶斯块算法和最大似然算法作为离散算法和估计参数。它返回基因及其秩之间所有可能的边。最后,通过保留PIDC算法结果中排名最高的1%的边来定义一个DEG网络。
采用ELMER包进行DMC网络重构。ELMER网络重构主要有3个步骤:
- 1.
在HM450平台上识别远端甲基化探针,
- 2.
选择正常组和肿瘤组之间DNA甲基化水平显著不同(即高甲基化和低甲基化都有)的远端探针,
- 3.
连接假定的靶基因及其DMC探针。
该过程将返回具有显著(调整过的-\(p < 0.01\))探针的甲基化与基因的表达呈负相关。
基序和转录因子鉴定
为了识别通过与TF结合基序DNA甲基化来充当上游主调控因子的TF,我们执行了另外两个步骤:
- 1.
鉴定差异甲基化探针中的富集基序
- 2.
鉴定其表达与TF结合基序DNA甲基化相关的调控TF
我们使用TCGA完成了从DMC网络重建到TF识别的所有步骤。ELMER包中无监督模式下的管道功能。
网络集成
为了结合DMC和DEG网络,我们执行了一个图并操作。因此,集成网络中的顶点和边是来自DMC和DEG网络的顶点和边的并集。然后,我们提取有最多连通分量的子图或图的巨型分量。
网络分析
我们在集成网络上进行了群落识别、基因集富集分析和中间中心性测量。使用Leiden算法对网络进行社区检测10通过Python包leidenalg (https://github.com/vtraag/leidenalg).在这个函数中,我们使用CPMVertexPartition作为目标函数参数,0.0085作为分辨率参数的值。利用g:Profiler对各群落进行基因集富集分析69.我们使用京都基因和基因组百科全书、Reactome、WikiPathways和基因本体(分子功能、细胞成分和生物过程)作为g:Profiler的数据源。使用Graph.jl包计算中介中心性70.我们使用g:SCS算法来调整P在基因集富集分析中的应用价值。g:SCS方法是g:Profiler中用于计算多次测试校正的默认方法11.
为了分析社区间的交互,我们使用ForceAtlas2作为网络布局算法来可视化Gephi中的网络12,71.然后,我们计算了一个比率\ (cx (y) \),它度量了源社区之间连接的链接的比例x及目标社区y到源社区上的链接总数x没有连接到源社区本身(例如,社区间链接)。具体表述如下:
在哪里\ (L_x (y) \)连接社区的链接数是多少x和社区y.根据定义,\ (L_x (y) \)等于\ \ (L_y (x)).的分母\ (cx (y) \)是连接社区的链接数的总和x除社区外的其他社区x本身。
我们进行了网络随机化测试,以确定是否比例\ (cx (y) \)随机发生。网络随机化检验步骤如算法1所示
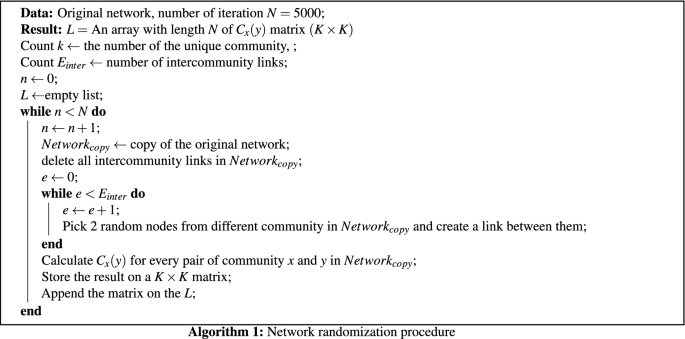
然后,我们对观察到的数据进行单样本t检验\ (C_ {x} (y) \)和\ (C_ {x} (y) \)从网络随机化过程。我们调整了P值使用邦费罗尼校正。
生存分析
我们根据中间中心性的十分位数将基因分成10个相等的组。然后,我们对每个基因进行单变量Kaplan-Meier生存分析。我们使用33百分位和67百分位作为分位数阈值来识别基因低表达和高表达的样本。我们使用TCGAbiolinks包中的tcgaanalyze_survivvalkm来执行生存分析。计算错误发现率(FDR)以校正多个假设检验,并且仅在FDR调整的情况下,结果才被接受为显著性\(p < 0.25\).我们统计了每组中显著基因的数量。通过Spearman相关检验,寻找显著基因数量与间性中心性之间的相关性。
在我们的研究中执行和复制所有分析的源代码可在GitHub存储库(https://github.com/yusri-dh/LUSC_integrated_network/).
数据可用性
我们下载了公开可用的LUSC数据集:美国国家癌症研究所(NCI)基因组数据共享(GDC) TCGAhttps://gdc.cancer.gov/access-data/gdc-data-portal采用TCGAbiolinks和ELMER封装。
参考文献
Lewis, d.r., Check, d.p., Caporaso, n.e., Travis, W. D. & Devesa, s.s.美国肺癌组织学类型趋势。癌症120, 2883 - 2892。https://doi.org/10.1002/cncr.28749(2014)。
库马尔,V.,阿巴斯,A. K. &阿斯特,J. C.。罗宾斯和科特兰疾病病理基础第十版。(Elsevier, Philadelphia, PA, 2021)。
网络,t.c.g.a.r.鳞状细胞肺癌的综合基因组特征。自然489, 519 - 525。https://doi.org/10.1038/nature11404(2012).
佩雷斯-莫雷诺,P., Brambilla, E., Thomas, R. & Soria, j . c .。肺鳞状细胞癌:分子亚型和治疗机会。中国。癌症Res。18, 2443 - 2451。https://doi.org/10.1158/1078-0432.ccr-11-2370(2012).
姚玲,沈海华,莱尔德,法纳姆,P. J.和伯曼,B. P.从癌症甲基化组推断调控元件景观和转录因子网络。基因组医学杂志。https://doi.org/10.1186/s13059-015-0668-3(2015)。
元,G。et al。NSD3组蛋白甲基化活性升高导致鳞状细胞肺癌。自然590, 504 - 508。https://doi.org/10.1038/s41586-020-03170-y(2021)。
桑切斯,R. & Mackenzie, S. A.白血病中差异甲基化和表达基因的生物标志物鉴定的综合网络分析。科学。代表。https://doi.org/10.1038/s41598-020-58123-2(2020)。
李娟,张强,陈志,徐东,王艳,一种基于网络的途径扩展方法,利用DNA甲基化和基因表达数据识别改变的途径。科学。代表。https://doi.org/10.1038/s41598-019-48372-1(2019)。
纳V。et al。人类基因调控网络中核心调控基因的自动识别。公共科学图书馆第一版。医学杂志。11, e1004504。https://doi.org/10.1371/journal.pcbi.1004504(2015)。
特拉格,V. A.沃特曼,L. &范埃克,N. J.《从鲁汶到莱顿:保证良好连接的社区》。科学。代表。https://doi.org/10.1038/s41598-019-41695-z(2019)。
Reimand, J., Kull, M., Peterson, H., Hansen, J. & Vilo, J. g: profiler -一个基于网络的工具集,用于从大规模实验中分析基因列表的功能。核酸测定。35, W193-W200。https://doi.org/10.1093/nar/gkm226(2007)。
Jacomy, M., Venturini, T., Heymann, S. & Bastian, M. ForceAtlas2,为Gephi软件设计的方便网络可视化的连续图布局算法。《公共科学图书馆•综合》9, e98679。https://doi.org/10.1371/journal.pone.0098679(2014)。
Pratapa, A., Jalihal, A. P., Law, J. N., Bharadwaj, A. & Murali, T. M.从单细胞转录组数据进行基因调控网络推理的基准算法。Nat方法。17, 147 - 154。https://doi.org/10.1038/s41592-019-0690-6(2020)。
科尔西斯先生。et al。原发性人类癌症的染色质可及性图景。科学https://doi.org/10.1126/science.aav1898(2018)。
Huynh-Thu, V. A., Irrthum, A., Wehenkel, L. & Geurts, P.使用基于树的方法从表达数据推断调节网络。《公共科学图书馆•综合》5, e12776。https://doi.org/10.1371/journal.pone.0012776(2010)。
金,S. Ppcor:用于快速计算半偏相关系数的R包。Commun。Stat .。方法22, 665-674(2015)。
高Papili, N., Ud-Dean, s.m.m., Gandrillon, O. & Gunawan, R.诚:从时间戳单细胞转录表达谱推断基因调控网络。生物信息学34, 258-266(2018)。
陈,t.e.,斯坦普夫,M. P. & Babtie, a.c.基因调控网络推断单细胞数据使用多元信息措施。细胞系统。5251 - 267. - e3。https://doi.org/10.1016/j.cels.2017.08.014(2017)。
马洪,s.s.m。et al。信息理论与信号转导系统:从分子信息处理到网络推理。Semin。细胞发育生物学。35, 98 - 108。https://doi.org/10.1016/j.semcdb.2014.06.011(2014)。
科尔多布斯基,文学硕士。et al。通过信息理论分析确定的儿童急性淋巴细胞白血病的遗传和表观遗传驱动因素。Nat,生物医学。Eng。5, 360 - 376。https://doi.org/10.1038/s41551-021-00703-2(2021)。
吴丹,张荣,Datta, S.揭示T细胞反应对SARS-CoV-2感染的长期保护。前面。麝猫。https://doi.org/10.3389/fgene.2022.871164(2022)。
洪绍培et al。单细胞转录组学揭示内分泌治疗的多步骤适应。Commun Nat。10, 3840年。https://doi.org/10.1038/s41467-019-11721-9(2019)。
席尔瓦,t.c.。et al。ELMER vol 2:从DNA甲基化和转录组谱重建基因调控网络的r/生物导体包。生物信息学35, 1974 - 1977。https://doi.org/10.1093/bioinformatics/bty902(2018)。
Ravi, N., Yang, M., Mylona, N., Wennerberg, J. & Paulsson, K.原发性间变性甲状腺癌的整体RNA表达和DNA甲基化模式。癌症https://doi.org/10.3390/cancers12030680(2020)。
黄,Y。et al。激活二价因子DLX5与主调节因子TP63合作促进鳞状细胞癌。核酸测定。49, 9246 - 9263。https://doi.org/10.1093/nar/gkab679(2021)。
Nassiri F。et al。脑膜瘤的临床应用综合分子分型。自然597, 119 - 125。https://doi.org/10.1038/s41586-021-03850-3(2021)。
科勒,F。et al。Hutchinson-Gilford早衰综合征中椎板相关结构域的表观遗传失调控。基因组医学。12, 46岁。https://doi.org/10.1186/s13073-020-00749-y(2020)。
哈纳汉,D. &温伯格,R. A.癌症的特征:下一代。细胞144, 646 - 674。https://doi.org/10.1016/j.cell.2011.02.013(2011)。
Kleczko, E. K., Kwak, J. W., Schenk, E. L. & Nemenoff, R. a .靶向补体通路作为肺癌的治疗策略。前面。Immunol。https://doi.org/10.3389/fimmu.2019.00954(2019)。
张荣,刘强,李涛,廖强,赵艳。补体系统在肿瘤微环境中的作用。癌细胞Int。https://doi.org/10.1186/s12935-019-1027-3(2019)。
Tagalakis, V。et al。非小细胞肺癌患者深静脉血栓形成的高风险:一项493例患者的队列研究j . Thorac。肿瘤防治杂志。2, 729 - 734。https://doi.org/10.1097/jto.0b013e31811ea275(2007)。
Castell, j.v., Donato, m.t. & Gómez-Lechón, m.j.肺中毒物的代谢和生物活化。体外细胞方法。Exp Toxicol。病理学研究。57, 189 - 204。https://doi.org/10.1016/j.etp.2005.05.008(2005)。
Ishitsuka, Y., Ogawa, T. & Roop, D.角化中的KEAP1/NRF2信号通路。抗氧化剂9, 751年。https://doi.org/10.3390/antiox9080751(2020)。
Müller, T. & Hengstermann, A. NRF2:预防吸烟依赖性肺病的朋友和敌人。化学。Toxicol >,25, 1805 - 1824。https://doi.org/10.1021/tx300145n(2012).
Niture, S. K. & Jaiswal, a.k. Nrf2蛋白上调抗凋亡蛋白Bcl-2并防止细胞凋亡。生物。化学。https://doi.org/10.1074/jbc.m111.312694(2012).
Traverso, N。et al。谷胱甘肽在癌症进展和化疗耐药中的作用。氧化物。地中海。细胞。Longev。1 - 10, 2013年。https://doi.org/10.1155/2013/972913(2013)。
徐,C。et al。萝卜硫素抑制7,12 -二甲基苯(a)蒽诱导的C57BL/6小鼠皮肤肿瘤是由核因子e2相关因子2介导的。癌症Res。66, 8293 - 8296。https://doi.org/10.1158/0008-5472.can-06-0300(2006)。
Gould, N. S., Min, E., Gauthier, S., Martin, R. J. & Day, B. J.肺谷胱甘肽对香烟烟雾暴露的适应性反应。和。Res。https://doi.org/10.1186/1465-9921-12-133(2011)。
马德森,d。et al。来自循环炎性单核细胞的肿瘤相关巨噬细胞通过细胞摄取降解胶原蛋白。细胞的代表。21, 3662 - 3671。https://doi.org/10.1016/j.celrep.2017.12.011(2017)。
德尤吉纳,e。et al。组织浸润性中性粒细胞是肿瘤微环境中诱导血管生成的MMP-9的主要体内来源。瘤形成16, 771 - 788。https://doi.org/10.1016/j.neo.2014.08.013(2014)。
Benoit, m.e., Clarke, e.v., Morgado, P., Fraser, d.a., Tenner, a.j.补体蛋白c1q指导巨噬细胞极化并限制凋亡细胞摄取过程中的炎性小体活性。j . Immunol。188, 5682 - 5693。https://doi.org/10.4049/jimmunol.1103760(2012).
李,L。et al。缺氧抑制肿瘤相关巨噬细胞中补体成分9的表达促进非小细胞肺癌进展细胞死亡发现。https://doi.org/10.1038/s41420-018-0064-3(2018)。
麦德勒,t.r.et al。补体C5a促进鳞状癌的发生并限制T细胞对化疗的反应。癌症细胞34561 - 578. - e6。https://doi.org/10.1016/j.ccell.2018.09.003(2018)。
阿林多夫。et al。c5a介导的白三烯b4扩增中性粒细胞趋化作用在抗肿瘤单克隆抗体和抗肿瘤单克隆抗体促进的肿瘤免疫治疗中至关重要\ \ upbeta \ ()葡聚糖。j . Immunol。174, 7050 - 7056。https://doi.org/10.4049/jimmunol.174.11.7050(2005)。
哈梅内伊,h.j.。et al。C5a调节IL-1\ \ upbeta \ ()尿酸一钠结晶性腹膜炎小鼠模型中白细胞的产生和募集。前面。杂志。https://doi.org/10.3389/fphar.2017.00010(2017)。
Guglietta, S。et al。c3ar依赖性NETosis诱导的凝血在小肠肿瘤发生过程中驱动原致中性粒细胞。Commun Nat。https://doi.org/10.1038/ncomms11037(2016)。
柯拉勒斯,L。et al。过敏性毒素C5a为肺癌的进展创造了有利的微环境。j . Immunol。189, 4674 - 4683。https://doi.org/10.4049/jimmunol.1201654(2012).
郭敬明,j.w.。et al。通过c3a受体途径激活补体改变CD4+ t淋巴细胞并介导肺癌进展。癌症Res。78, 143 - 156。https://doi.org/10.1158/0008-5472.can-17-0240(2017)。
郑浩,梅森,s.p., Barabási, a - l。蛋白质网络中的致死率和中心性。自然411, 41-42。https://doi.org/10.1038/35075138(2001).
何欣,张杰。为什么中枢在蛋白质网络中是必不可少的?公共科学图书馆麝猫。2, e88。https://doi.org/10.1371/journal.pgen.0020088(2006)。
Raman, K., Damaraju, N.和Joshi, G. K.蛋白质网络的组织结构:重新审视中心性-致命性假说。系统。Synth。医学杂志。8, 73 - 81。https://doi.org/10.1007/s11693-013-9123-5(2013)。
詹,P。et al。NCAPG2通过调节G2/M期促进肿瘤增殖,与肺腺癌不良预后相关。j .细胞。摩尔。地中海。21, 665 - 676。https://doi.org/10.1111/jcmm.13010(2016)。
金,E。et al。致癌长链非编码RNA psmg3 -反义1在肺鳞状细胞癌中的表达肿瘤防治杂志。列托人。https://doi.org/10.3892/ol.2021.13012(2021)。
西米诺Y。et al。FADD蛋白的释放反映了人类非小细胞肺癌的发展和侵袭性。Br。j .癌症106, 1989 - 1996。https://doi.org/10.1038/bjc.2012.196(2012).
阿兰达,j.f.。et al。MYADM通过erm依赖性调控ICAM-1表达来控制内皮屏障功能。摩尔。杂志。细胞24, 483 - 494。https://doi.org/10.1091/mbc.E11-11-0914(2013)。
太阳,L。et al。miR-182-3p/MYADM通过klf4/p21依赖机制参与肺动脉高压血管重塑。开展10, 5581 - 5599。https://doi.org/10.7150/thno.44687(2020)。
彭,B。et al。maldi-tof质谱测定生物标记物的蛋白质组学分析用于气管支气管结核后气管狭窄的诊断。其他实验。地中海。https://doi.org/10.3892/etm.2020.9495(2020)。
帕克,H. J.。et al。肺鳞状细胞癌角化与临床预后差相关。Tuberc。和。说。80, 179年。https://doi.org/10.4046/trd.2017.80.2.179(2017)。
黄。et al。癌症中tRNA及其衍生物的失调。J.临床经验。癌症Res。https://doi.org/10.1186/s13046-018-0745-z(2018)。
江,y y。et al。TP63, SOX2和KLF5建立了一个核心调节电路,控制食管鳞癌细胞系的表观遗传和转录模式。胃肠病学1591311 - 1327. - e19。https://doi.org/10.1053/j.gastro.2020.06.050(2020)。
龙,H. K. Blackledge, N. P. & Klose, R. J. ZF-CxxC结构域含蛋白,CpG岛和染色质连接。物化学。Soc。反式。41, 727 - 740。https://doi.org/10.1042/bst20130028(2013)。
金,H.-Y。et al。CXXC5是wnt/的负反馈调节器\ \ upbeta \ ()-catenin通路参与成骨细胞分化。细胞死亡不同。22, 912 - 920。https://doi.org/10.1038/cdd.2014.238(2015)。
燕,X。et al。CXXC5通过促进TGF-抑制肝细胞癌\ \ upbeta \ ()-诱导细胞周期阻滞和凋亡。细胞生物学。10, 48-59。https://doi.org/10.1093/jmcb/mjx042(2017)。
张,M。et al。CXXC finger 5蛋白是DNA损伤诱导的p53激活所必需的。科学。中国爵士。C生命科学。52, 528 - 538。https://doi.org/10.1007/s11427-009-0083-7(2009)。
高山,k。et al。FOXP1功能的综合分析揭示了前列腺癌的抑癌作用。摩尔。性。28, 2012 - 2024。https://doi.org/10.1210/me.2014-1171(2014)。
冯,J。et al。FoxP1的高表达与非小细胞肺癌患者生存率的提高有关。点。j .中国。病理学研究。138, 230 - 235。https://doi.org/10.1309/ajcpdhqfnyjz01yg(2012).
Colaprico,。et al。TCGAbiolinks:用于TCGA数据综合分析的r/生物导体包。核酸测定。44, e71-e71。https://doi.org/10.1093/nar/gkv1507(2015)。
Silva, t.c.等人(2016)TCGA工作流程:使用生物导体包分析癌症基因组学和表观基因组学数据。f1000研究5:1542,https://doi.org/10.12688/f1000research.8923.2
Raudvere U。et al。g:profiler:用于功能丰富分析和基因列表转换的web服务器(2019年更新)。核酸测定。47, W191-W198。https://doi.org/10.1093/nar/gkz369(2019)。
费尔班克斯,J。et al。Juliagraphs / /图表。jl:为julia编程语言优化的图形包。https://github.com/JuliaGraphs/Graphs.jl(2021)。
巴斯蒂安,M.,海曼,S. & Jacomy, M. Gephi:用于探索和操纵网络的开源软件(2009)。
作者信息
作者及隶属关系
贡献
Y.D.H.负责研究的概念化、数据管理、分析和可视化,并撰写手稿的原始草稿。K.K.负责研究的概念化、监督和编辑手稿。S.I.负责资金获取、项目管理、监督、稿件编辑等工作。所有作者都审阅了手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
Heryanto, Y.D, Katayama, K. & Imoto, S.分析肺鳞状细胞癌甲基化和基因表达谱的综合网络。Sci代表12, 15799(2022)。https://doi.org/10.1038/s41598-022-20232-5
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-022-20232-5