摘要gydF4y2Ba
与粘膜疾病相关的口腔微生物群,包括口腔鳞状细胞癌和口腔潜在恶性疾病,已在门和属水平上进行了广泛的分析。然而,口腔微生物群的细节在物种和操作分类单位(OTU)水平上仍然不清楚。我们的目的是确定口腔冲洗、病变和正常部位拭子样本中舌头粘膜异常患者的微生物群的差异。口腔标本取自10例口腔黏膜异常患者。基于Sanger法获得的16S rRNA基因序列,采用相似度为99.6%的OTU聚类,分析了微生物样品OTU和属水平的Alpha和beta多样性。在OTU水平上,病变的微生物区系多样性最小,但与正常部位和口腔冲洗样品的微生物区系不同。对应的OTUsgydF4y2Ba链球菌对象gydF4y2Ba而且gydF4y2Ba嗜血杆菌56gydF4y2Ba提示病变部位与正常部位微生物群差异的原因。在属水平上,这些微生物群之间没有观察到显著差异。综上所述,严格的otu水平微生物群分析可能能够区分粘膜异常患者的病变部位与正常部位。gydF4y2Ba
简介gydF4y2Ba
2020年,全球报告了377,713例口腔癌新患者,其中177,757例死亡gydF4y2Ba1gydF4y2Ba.2018年,有22,515名日本人被诊断出患有口腔癌和咽喉癌,2020年有7827名日本患者死于这些疾病(gydF4y2Bahttps://ganjoho.jp/reg_stat/statistics/data/dl/en.htmlgydF4y2Ba).口腔癌(OSCC)占口腔癌的90%以上gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.据报道,除了吸烟和饮酒,口腔细菌也与口腔癌有关gydF4y2Ba2gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.口腔细菌的参与也引起了从口腔潜在恶性疾病(OPMD)到OSCC的恶性变化的关注gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba.gydF4y2Ba
使用下一代DNA测序(NGS)分析了健康和疾病中的人类微生物群gydF4y2Ba9gydF4y2Ba.许多全身和口腔疾病中的口腔微生物群也已使用NGS进行了研究gydF4y2Ba10gydF4y2Ba.口腔黏膜疾病相关的口腔菌群分析已被广泛报道。在这些研究中,唾液gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,漱口样品/漱口水gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba口腔癌组织gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,拭子gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba以及各种临床标本gydF4y2Ba25gydF4y2Ba作为目标标本。这些研究大多使用16S rRNA基因作为靶基因,但用于PCR的引物组不同gydF4y2Ba11gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba25gydF4y2Ba.使用NGS,过滤和修剪后的核苷酸序列通常聚成相似度为97%或98%的操作分类单位(OTUs),并应用于门和属水平的微生物区系分析。一般来说,口腔细菌包括一个由许多密切相关的物种组成的属,例如gydF4y2Ba链球菌gydF4y2Ba;然而,口腔黏膜疾病中口腔微生物群的物种水平的细节尚未完全阐明。Segata等报道,口腔黏膜、角质化牙龈和硬腭中的微生物群与舌背和唾液中的微生物群不同gydF4y2Ba26gydF4y2Ba.虽然口腔黏膜癌中存在微生物群gydF4y2Ba23gydF4y2Ba还有牙龈鳞癌gydF4y2Ba25gydF4y2Ba已经使用NGS进行了分析,舌头微生物群的细节还没有完全分析,然而舌头是口腔癌最常见的部位gydF4y2Ba27gydF4y2Ba.gydF4y2Ba
在本研究中,为了阐明与口腔黏膜疾病相关的口腔微生物群的细节,我们获得了舌部口腔黏膜异常患者的拭子和口腔冲洗样本,并基于Sanger方法采用高精度16S rRNA基因测序对样本中的微生物群进行了分析。gydF4y2Ba
结果gydF4y2Ba
学科特点gydF4y2Ba
10例患者的特点及临床诊断见表gydF4y2Ba1gydF4y2Ba和补充图。gydF4y2BaS1gydF4y2Ba.共分析30个样品。患者的中位年龄(7男3女)为68岁(范围50-92岁)。根据世界卫生组织的分类,这些患者包括2例诊断为OSCC和8例诊断为OPMD。gydF4y2Ba
细菌细胞总数gydF4y2Ba
细菌细胞总数见补充图。gydF4y2BaS2gydF4y2Ba.拭子样本中细菌的平均数量为1.7 × 10gydF4y2Ba7gydF4y2Ba细胞/ml(从1.5 × 10gydF4y2Ba6gydF4y2Ba到3.2 × 10gydF4y2Ba7gydF4y2Ba细胞/ml), 8.3 × 10gydF4y2Ba6gydF4y2Ba细胞/ml(范围为5.2 × 10gydF4y2Ba5gydF4y2Ba到2.2 × 10gydF4y2Ba7gydF4y2Ba细胞/ml)(补充图;gydF4y2BaS2gydF4y2Baa).病变拭子的细菌细胞计数与正常部位无明显差异(p > 0.05)。gydF4y2BaS2gydF4y2Bab).口腔漱口液样品中细菌的平均数量为1.0 × 10gydF4y2Ba8gydF4y2Ba细胞/ml(范围3.5 × 10gydF4y2Ba7gydF4y2Ba到2.2 × 10gydF4y2Ba8gydF4y2Ba细胞/ml)(补充图。gydF4y2BaS2gydF4y2Bac)。gydF4y2Ba
OTU水平分析gydF4y2Ba
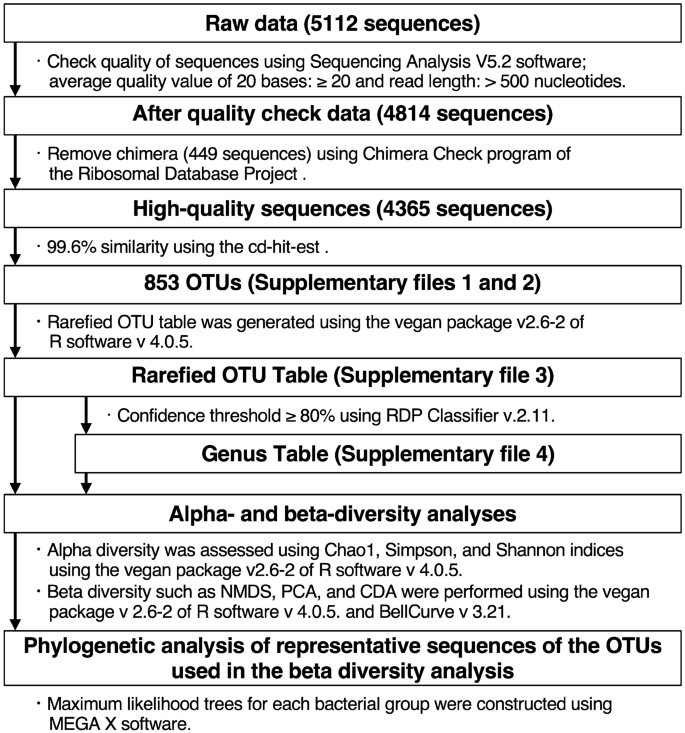
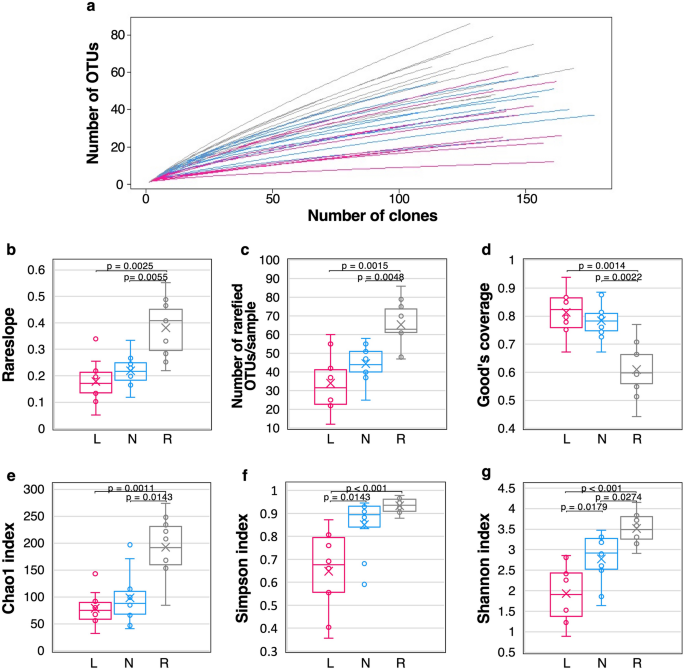
在本研究中,从30个样本中获得了4814个高度精确的序列reads(图。gydF4y2Ba1gydF4y2Ba).去除嵌合序列(449条)后,剩余4365条序列reads聚为853个OTUs,相似度为99.6% (Supplementary Files)gydF4y2BaS1gydF4y2Ba,gydF4y2BaS2gydF4y2Ba).拭子样品的稀疏曲线比口腔冲洗样品更接近饱和(图。gydF4y2Ba2gydF4y2Baa).所有样本中基于精细化OTU表的OTU相对丰度显示,OTU组成在个体患者和样本类型中存在较大差异(Supplementary File)gydF4y2BaS3gydF4y2Ba,补充图。gydF4y2BaS3gydF4y2Ba).拭子样本的罕见斜率值也显著低于口腔冲洗样本(p < 0.05)。gydF4y2Ba2gydF4y2Bab).病变、正常部位和口腔冲洗样本中检出的OTUs平均数量分别为34.0(12 ~ 60个OTUs)、44.5(25 ~ 58个OTUs)和65.4(47 ~ 86个OTUs)。gydF4y2Ba2gydF4y2Bac). Good对病变和正常部位的覆盖率估计平均为0.81(范围从0.67到0.94)和0.78(范围从0.67到0.88)(图。gydF4y2Ba2gydF4y2Ba分别d)。病变组和正常部位拭子组OTUs数量低于口腔冲洗组(p < 0.05),而病变组和正常部位拭子组Good 's覆盖估计值明显高于口腔冲洗组(p < 0.05)。gydF4y2Ba
OTU水平α多样性的稀疏性分析与比较。(gydF4y2Ba一个gydF4y2Ba)稀疏曲线。病变样本为粉色线,正常部位样本为蓝色线,口腔冲洗样本为灰色线。(gydF4y2BabgydF4y2Ba)稀坡,(gydF4y2BacgydF4y2Ba)精细化OTUs数量/样品,(gydF4y2BadgydF4y2Ba) Good的保险范围gydF4y2BaegydF4y2Ba) Chao1指数,(gydF4y2BafgydF4y2Ba)辛普森指数,及(gydF4y2BaggydF4y2Ba香农指数。(gydF4y2Bab-ggydF4y2Ba)箱形图显示了第25和第75百分位(分别为箱的底部和顶部)、中位数(中间水平线)、平均值(十字标记)以及不是异常值的最小值和最大值(顶部和底部须)。异常值定义为大于1.5×四分位间距(点)的值。使用Kruskal-Wallis检验评估三种样本类型之间的统计学意义。多重比较采用Steel-Dwass检验(bars, p < 0.05)。gydF4y2BalgydF4y2Ba病变,gydF4y2BaNgydF4y2Ba正常的网站,gydF4y2BaRgydF4y2Ba口腔冲洗,gydF4y2BaOTUgydF4y2Ba操作分类学单位。gydF4y2Ba
利用OTUs评价α-多样性的结果如图所示。gydF4y2Ba2gydF4y2Baeg。Chao1、Simpson和Shannon三项指标中,口腔冲洗样品的α-多样性显著高于病变部位和正常部位样品(p < 0.05)。病变拭子的Simpson指数和Shannon指数均低于正常拭子(p < 0.05)。这些结果清楚地表明,病变拭子的微生物群多样性低于正常部位拭子和口腔冲洗样本。gydF4y2Ba
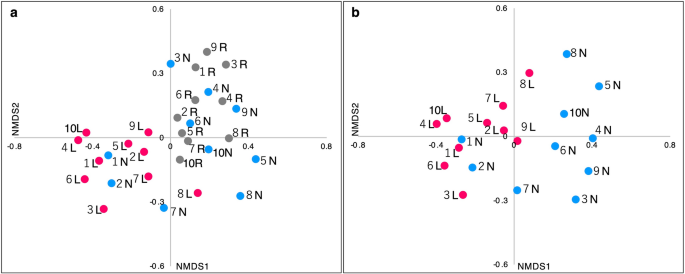
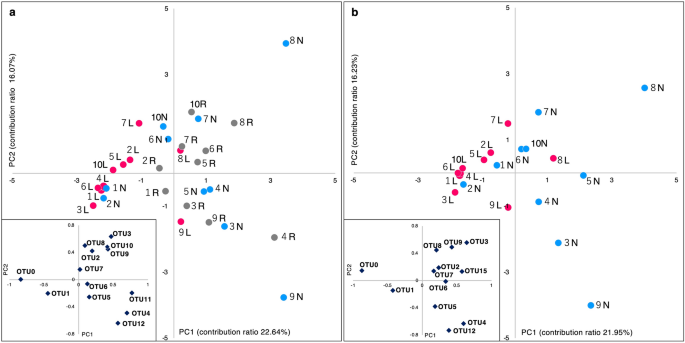
基于所有样本OTUs的β-多样性采用三种多变量分析进行评估。使用Bray-Curtis不相似指数的非度量多维尺度(NMDS)分析显示,病变拭子上的斑点倾向于独立于正常部位拭子和口腔冲洗样本(图2)。gydF4y2Ba3.gydF4y2Baa、b)。在主成分分析(PCA)结果(图。gydF4y2Ba4gydF4y2Ba),正常部位和口腔冲洗样品的斑点沿第一主成分(PC1)正方向分布(图。gydF4y2Ba4gydF4y2Baa).病变拭子上的斑点则呈PC1阴性方向分散。由于其中一个具有代表性的特征向量在OTU 0时对PC1的负方向有较大的影响,其对应的细菌可能是区分病变菌群的关键因素。仅比较拭子样本(图。gydF4y2Ba4gydF4y2Bab),病变部位与正常部位的斑点也被清晰地分开,并提示OTU 0作为特征向量在区分中的强作用。gydF4y2Ba
基于OTU水平的主成分分析。(gydF4y2Ba一个gydF4y2Ba)三种样本类型。(gydF4y2BabgydF4y2Ba)两份拭子样本。横轴为PC1,纵轴为PC2。特征向量在下面的方框中显示。其他的OTUs相对丰度< 1%在(gydF4y2Ba一个gydF4y2Ba)所有样本的总序列和(gydF4y2BabgydF4y2Ba)病变部位和正常部位的总序列。每个点对应一个样本。病变样本为粉红点,正常部位样本为蓝点,口腔冲洗样本为灰点。数据标签显示了患者编号。gydF4y2BalgydF4y2Ba病变,gydF4y2BaNgydF4y2Ba正常的网站,gydF4y2BaRgydF4y2Ba口腔冲洗,gydF4y2BaOTUgydF4y2Ba操作分类单位,gydF4y2BaPC1gydF4y2Ba第一个主成分,gydF4y2BaPC2gydF4y2Ba第二个主成分。gydF4y2Ba
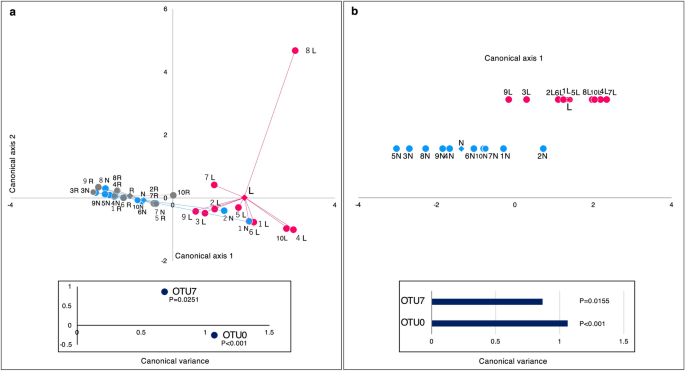
为了检测能有效区分病变拭子与其他样本的因素,将典型判别分析(canonical discriminant analysis, CDA)应用于这些样本(图。gydF4y2Ba5gydF4y2Ba).OTUs 0和7作为CDA中区分病变拭子与其他样本的鉴别因素的作用如图所示。gydF4y2Ba5gydF4y2BaA,b (p < 0.05)。这些结果表明,病变样品与正常部位和口腔冲洗样品含有不同的微生物群。此外,我们发现OTUs 0和7对应的细菌种类可能有助于区分病变和正常部位。利用RDPII网站上的分类器对OTUs 0 ~ 12和15的代表性序列进行属估计后(补充表gydF4y2BaS1gydF4y2Ba),基于每个属的16S rRNA基因序列进行系统发育分析(补充图。gydF4y2BaS4gydF4y2Ba- - - - - -gydF4y2BaS10gydF4y2Ba).ML树显示OTUs 0和7亲缘关系接近gydF4y2Ba链球菌对象gydF4y2Ba而且gydF4y2Ba嗜血杆菌56,gydF4y2Ba分别。gydF4y2Ba
基于精细化OTU水平的典型判别分析。(gydF4y2Ba一个gydF4y2Ba)三种样本类型。(gydF4y2BabgydF4y2Ba)两份拭子样本。每个点表示每个样本基于各自典型变量的典型得分(CS)。x轴是规范轴1,y轴是规范轴2。每个点对应一个样本。病变部位为粉红点,正常部位为蓝点,口腔冲洗样品为灰点,每个重心为菱形。数据标签显示患者编号。gydF4y2BalgydF4y2Ba病变,gydF4y2BaNgydF4y2Ba正常的网站,gydF4y2BaRgydF4y2Ba口腔冲洗。其他的OTUs相对丰度< 1%在(gydF4y2Ba一个gydF4y2Ba)所有样本的总序列和(gydF4y2BabgydF4y2Ba)病变部位和正常部位的总序列。gydF4y2BaOTUgydF4y2Ba操作分类学单位。正则判别函数p < 0.05。gydF4y2Ba
属级分析gydF4y2Ba
在属级分析中,这三个属:gydF4y2Ba链球菌gydF4y2Ba(57.52%),gydF4y2BaGemellagydF4y2Ba(8.76%)和gydF4y2Ba奈瑟氏菌属gydF4y2Ba(6.37%)占总序列的72.65% (Supplementary File .gydF4y2BaS4gydF4y2Ba,补充图。gydF4y2BaS11gydF4y2Ba).然而,属gydF4y2Ba梭菌属gydF4y2Ba(2.98%)和gydF4y2Ba普氏菌gydF4y2Ba(2.45%),其中包括几种已知的牙周病原体,占本研究总序列的不到3%。在属水平上的α-多样性分析显示,病变拭子的Chao1指数显著低于口腔冲洗样品(p < 0.05)。gydF4y2BaS12gydF4y2Ba).在NMDS和基于属级的PCA结果中,这些样品之间没有显著差异(补充图)。gydF4y2Ba向gydF4y2Ba,gydF4y2BaS14系列gydF4y2Ba).CDA结果显示了属的贡献gydF4y2Ba奈瑟氏菌属、韦永氏球菌属gydF4y2Ba而且gydF4y2BaSphingomonasgydF4y2Ba(p < 0.05)。gydF4y2BaS15gydF4y2Baa).属gydF4y2Ba韦永氏球菌属gydF4y2Ba作为区分病变与正常菌群的因素之一(p < 0.05)(补充图。gydF4y2BaS15gydF4y2Bab).然而,没有发现病变样本的特异性因素。gydF4y2Ba
讨论gydF4y2Ba
本研究首次采用基于Sanger测序和严格OTU聚类的高精度16S rRNA基因序列,分析了舌头可见口腔黏膜异常(包括OSCC和OPMD)的微生物群,相似性接近100%。结果显示,舌部病变的拭子样本、舌部正常部位的拭子样本和可见口腔黏膜异常患者的口腔冲洗样本中的微生物群存在显著差异。gydF4y2Ba
许多基于NGS的关于口腔微生物群的研究都是使用唾液或口腔冲洗样本进行的gydF4y2Ba7gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba.基于本研究中的OTU水平,口腔冲洗样品的微生物区系与病变拭子样品的微生物区系不同。这说明口腔冲洗样品的菌群不能代表病变,口腔冲洗样品不适合调查病变菌群的因素。先前的研究表明OSCC和OPMD病变部位与gydF4y2Ba梭菌属gydF4y2Ba18gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2BaAlloprevotellagydF4y2Ba23gydF4y2Ba,gydF4y2BaPorphyromonasgydF4y2Ba18gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba普氏菌gydF4y2Ba17gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba棒状杆菌属gydF4y2Ba17gydF4y2Ba,gydF4y2Ba假单胞菌gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba消化链球菌属gydF4y2Ba22gydF4y2Ba,与正常部位配合gydF4y2Ba链球菌gydF4y2Ba18gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba而且gydF4y2Ba韦永氏球菌属gydF4y2Ba20.gydF4y2Ba.大多数研究都在属和OTU水平上进行分析(相似度几乎为97%或98%),但每项研究的结果和结论都不相同。因此,预测OSCC和OPMC的细菌因子尚未完全阐明。gydF4y2Ba
在本研究中,属gydF4y2Ba链球菌gydF4y2Ba在所有三种样本类型中都是最主要的。这与健康受试者唾液研究的结果一致gydF4y2Ba28gydF4y2BaOSCC患者的唾液和口腔冲洗样本gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba以及OSCC患者的拭子gydF4y2Ba23gydF4y2Ba.众所周知,有许多不同种类的细菌属于该属gydF4y2Ba链球菌gydF4y2Ba口腔中的16S rRNA基因序列非常相似。识别这些细菌种类并详细分析微生物群是不可能用相似度为97%和99%的聚类OTUs进行的gydF4y2Ba29gydF4y2Ba.在以往使用NGS技术的研究中,大多数都使用了相似度为97%或98%的OTUs聚类,且分析仅限于属水平。V3-V5区域比V3-V4区域在物种水平上更好地识别细菌,其中一个例子是gydF4y2Ba链球菌gydF4y2Ba物种gydF4y2Ba30.gydF4y2Ba.由于Sanger方法可以读取相对较长的序列,并且比其他NGS方法读取更准确gydF4y2Ba31gydF4y2Ba,该方法目前适用于获得约600 bp的V3-V5区域的高精度碱基序列。为了了解口腔黏膜疾病(如OSCC、OPMD)与细菌群落之间的相关性,有必要使用高精度的16S rRNA基因序列,在严格的OTUs基础上阐明口腔微生物区系。gydF4y2Ba
在之前的一项研究中,我们对Sanger法测定的高精度核苷酸序列进行了OTU分析,相似性为99.6%,并证明了其在物种或菌株水平上区分鼻分泌物中微生物群的能力gydF4y2Ba32gydF4y2Ba.该分析方案应用于口腔菌群研究,发现病变部位菌群α-多样性低于正常部位,并发现口腔冲洗样品。这一结果与以往基于NGS的研究结果相似gydF4y2Ba12gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,但与赵等的结果不同。gydF4y2Ba22gydF4y2Ba.本研究中特别有趣的是,在β多样性分析中使用严格的OTUs (NMDS, PCA和CDA)可以区分病变和正常部位,并确定了独特的细菌因子候选。与属的一个种相对应的OTUgydF4y2Ba链球菌gydF4y2Ba,推测为gydF4y2Ba美国的对象gydF4y2Ba(OTU 0),对PCA和CDA的病灶收敛均有显著影响。OTU的显著效应对应于gydF4y2Bah . 56gydF4y2Ba(OTU 7)在病变中也被建议。根据以往NGS的研究结果,该属gydF4y2Ba链球菌gydF4y2Ba而且gydF4y2Ba嗜血杆菌gydF4y2Ba在正常部位比病变部位更常见,这与本研究的结果不一致gydF4y2Ba18gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba.在健康个体中,gydF4y2Ba美国的对象gydF4y2Ba有报道显示舌头的特定部位定植,但很少牙龈下斑块gydF4y2Ba33gydF4y2Ba.相比之下,gydF4y2Ba美国的对象gydF4y2Ba通过对舌苔样本的宏基因组分析,是否被认为是胃炎的潜在生物标志物,包括癌前病变gydF4y2Ba34gydF4y2Ba.OSCC等粘膜疾病病变与OPMD的关系有待进一步分析;然而,gydF4y2Ba美国的对象gydF4y2Ba可能是这些疾病的前兆。虽然在NGS分析中通常使用属水平分析,但本研究中属水平的结果并不能清楚地描述病变和正常部位的微生物区系。本研究结果与其他NGS研究的差异不仅在于样本类型和靶16S rRNA基因可变区域的差异,还在于OTU聚类相似性。因此,在对获得的OTU进行细菌分类之前,可以通过分析OTU序列来确定参与菌群收敛的重要因素。gydF4y2Ba
在某些病例中,如患者1和2,病变和正常部位β-多样性差异异常不明显。在这些病例中,病变和正常部位之间细菌细胞计数的差异可能会影响结果。据报道,细菌细胞数量影响疾病的发病gydF4y2Ba35gydF4y2Ba.病变与细菌细胞计数之间的关系需要进一步研究。另一种可能是,患者1和2可能容易在口腔中出现多个病变,尽管无法从视觉上识别。gydF4y2Ba
这项研究有一些局限性。首先,用棉签擦拭的区域为1厘米gydF4y2Ba2gydF4y2Ba,使得难以收集较小的病变样本。其次,本研究分析的克隆数量高达192个/份样品,该方法可能无法检测小群体的细菌种类。事实上,稀疏分析表明样本量不足以覆盖整个菌群。然而,由于我们的主要目的是评估口腔样品中微生物群的确切差异,这种影响可能并不显著。第三,由于样本量有限,仅10例患者,无法对OSCC和OPMD的菌群进行统计学比较。在本研究中,尚无法确定病变部位口腔微生物群的变化是否导致了癌症的发展,或者其他因素导致的口腔黏膜恶性转化是否改变了病变部位的微生物群。gydF4y2Ba
综上所述,基于OTU水平的分析揭示了口腔病变菌群的特征;然而,基于属水平的分析无法区分病变部位的微生物群与正常部位的微生物群。这些结果清楚地表明,在比属水平(如种或OTU水平)更高的分辨率下调查细菌谱系,可能对区分口腔部位样本很重要。gydF4y2Ba
方法gydF4y2Ba
参与者人数gydF4y2Ba
采用单中心前瞻性观察研究。纳入了2018年至2020年在日本职业与环境卫生大学医院牙科和口腔外科就诊的20岁及以上舌侧边界有明显口腔粘膜异常的患者。在入组时收集性别、年龄等临床信息。样本采集于患者活检和抗生素治疗前。gydF4y2Ba
样品收集gydF4y2Ba
为了消除由于个体差异造成的采样误差,棉签和口腔冲洗样本由同一名牙医收集。从肉眼可见病变的粘膜表面和舌上解剖位置相反的正常部位采集拭子。使用1平方厘米的l型卡尺从特定区域(约1厘米)采集拭子样本gydF4y2Ba2gydF4y2Ba)显示病变部位及正常部位。采样点表面干燥,每1 cm干燥一定面积gydF4y2Ba2gydF4y2Ba用无菌棉签擦拭10次,保存于15 ml无菌管中。然后将口腔冲洗样品用5ml消毒蒸馏水冲洗10 s,收集于50ml无菌管中。所有样品立即在4°C保存,直到下一个步骤(1 d内)。在下一个步骤之前,为了在棉签上制备细菌细胞悬浮液,将1 ml磷酸盐缓冲生理盐水加入到15 ml试管中的棉签中,并旋转15 s。随后,使用布兰森超声波洗衣机(Branson超声波公司)对其进行超声处理5分钟。gydF4y2Ba
道德声明gydF4y2Ba
这项研究得到了日本职业与环境卫生大学医学研究伦理委员会的批准(UOEHCRB21-119)。本研究所有方法均按照相关指南和规定进行。所有患者均获得知情的书面同意。gydF4y2Ba
数字化的染色gydF4y2Ba
如前所述,使用荧光染色方法对样品中的所有细菌细胞进行计数gydF4y2Ba36gydF4y2Ba.用0.1 M磷酸盐缓冲液[pH 8.5], 5% NaCl, 0.5 mM EDTA钠,分别从拭子样品(10 μl)和口腔冲洗样品(50 μl)中取细菌悬液(0.1 M磷酸盐缓冲液[pH 8.5])充至1000 μl。然后,在1 ml溶液中加入50 μl (2.0 mg/ml)的溴化乙锭(EtBr)溶液。染色20min后,用0.2 μm孔径过滤器过滤细菌细胞,用2ml无菌磷酸盐缓冲盐水(PBS)冲洗。使用Olympus BX40外照荧光显微镜(Olympus, Tokyo, Japan)进行显微镜检查,并统计滤镜上30个场的细菌数量。计算从拭子和口腔冲洗样品制备的每1毫升细菌溶液中的细菌计数。gydF4y2Ba
DNA提取gydF4y2Ba
取棉签上的细菌细胞悬液640 μl和口腔冲洗样品400 μl,用PBS充至1440 μl,然后分别加入30% SDS(终浓度3.0%)溶液160 μl。治疗后与Astrason模型XL2020(美国纽约法明岱尔MISONIX Inc .)一次15秒和三次总共45 s (20 kHz), 700μl整除与400μl TE-saturated混合酚(Nacalai Tesque,京都,日本),在15000转离心5分钟。上层清液中提取的DNA与PBS和TE缓冲洗两次和集中(最终体积大约40μl在TE缓冲)使用一个Amicon超- 100 K滤波器(美国默克密理博有限公司)。用上述步骤从640 μl PBS中提取DNA作为阴性对照模板。gydF4y2Ba
聚合酶链反应gydF4y2Ba
使用通用引物E341F和E907R扩增部分16S rRNA基因(约580 bp,包括V3-V5可变区)gydF4y2Ba37gydF4y2Ba和AmpliTaq金DNA聚合酶LD(应用生物系统公司,美国),如前所述gydF4y2Ba36gydF4y2Ba.用2%琼脂糖凝胶电泳证实,从标本中提取的模板DNA中只有一条DNA条带,而从阴性对照模板中没有DNA条带。gydF4y2Ba
克隆文库构建及核苷酸测序分析gydF4y2Ba
确认PCR产物后,使用TOPO TA克隆试剂盒(Invitrogen, USA)根据制造商说明书构建克隆文库。从每个样品中随机选取192个菌落转入PCR板孔中,测序分析如前所述gydF4y2Ba36gydF4y2Ba.使用AmpliTaq Gold 360 DNA聚合酶(Applied Biosystems, USA)和引物组(M13Forward: 5ʹ-GTAAAACGACGGCCAG-3ʹ和M13Reverse: 5ʹ-CAGGAAACAGCTATGAC-3ʹ)对克隆载体(pCR 4 TOPO)中的插入物进行扩增。使用ExoSAP-IT (GE Healthcare UK Ltd., England, UK)去除未反应的引物和脱氧核糖核酸三磷酸,将PCR混合物(1 μl)使用BigDye Terminator Cycle sequencing Kit v3.1 (Applied Biosystems)进行测序反应。核苷酸序列是使用3130xl遗传分析仪(应用生物系统)确定的。使用测序分析V5.2软件(应用生物系统公司)评估序列读取的质量。gydF4y2Ba38gydF4y2Ba.经过质量检查(20个碱基的平均质量值≥20,读取长度为> 500个核苷酸),将引物和载体的序列从剩下的序列中剪去。此外,包含模糊碱基的序列被排除。使用核糖体数据库项目(RDP)的Check Chimera程序对其余高度精确的序列(约550 bp)进行嵌合体筛选。gydF4y2Ba39gydF4y2Ba.所有通过检测的序列均在日本DNA数据库中注册(登录号:LC672676-LC677040)。然后,将高度精确的序列聚成OTUsgydF4y2Ba40gydF4y2Ba使用cd-hit-estgydF4y2Ba41gydF4y2Ba以99.6%序列相似度作为阈值。处理后,使用R软件v 4.0.5的纯素包v2.6-2生成精细化OTU表。OTUs的代表性序列用于下面描述的细菌组成分析。gydF4y2Ba
对于属级分析,使用RDP Classifier v.2.11对每个OTU的代表序列进行属级分类,阈值为80%gydF4y2Ba42gydF4y2Ba.属水平低于80%的序列被归类为“未分类细菌”,处于下一个最高分类等级。占总序列数1%以下的OTUs和属分别被归为“其他”(补充表)gydF4y2BaS2gydF4y2Ba,gydF4y2BaS3gydF4y2Ba,gydF4y2BaS4gydF4y2Ba,gydF4y2BaS5gydF4y2Ba).gydF4y2Ba
数据分析gydF4y2Ba
本研究的所有数据分析均使用R软件v 4.0.5和BellCurve v 3.21进行。使用纯素包v2.6-2生成基于OTUs的稀疏曲线。OTUs的数量和Good的覆盖率估计gydF4y2Ba43gydF4y2Ba,以精细化OTUs表为基础,计算各样本数据集的多样性覆盖率。基于OTU和属水平的Alpha多样性使用Chao1、Simpson和Shannon指数使用素食包v2.6-2进行评估。为了评估病变拭子样本、正常拭子样本和口腔冲洗样本之间的β-多样性,采用BellCurve v 3.21进行NMDS、PCA和CDA等多变量分析。NMDS基于Bray-Curtis不相似度的距离矩阵进行分析,使用OTU或属在每个样本中的相对丰度,使用纯素包v 2.6-2。同样地,主成分分析使用每个样品中OTU或属的相对丰度。此外,进行逐步CDA以确定病变样本中显著的OTUs或属因子。病变拭子与正常拭子之间的比较还使用多变量分析进行了验证。gydF4y2Ba
统计分析gydF4y2Ba
使用Mann-Whitney法比较样品间的细菌细胞计数gydF4y2BaUgydF4y2Ba测试。采用Kruskal-Wallis检验评价OTU数、Good’s覆盖率、rareslope和α-多样性。采用钢屑试验对三种样品进行比较。p < 0.05为差异有统计学意义。本研究中所有统计分析均使用BellCurve软件进行。gydF4y2Ba
系统发育分析gydF4y2Ba
多变量分析中使用的OTUs的代表性序列的细菌谱系使用系统发育分析进行估计。利用RDPII网站上的分类器软件估计与每个序列密切相关的细菌谱系,并从RDP数据库中收集该属所含类型菌株的序列。使用MEGA X软件构建每个细菌群的最大似然树。为每个菌群(属)收集的序列使用MUSCLE算法多次对齐。每个系统发育分析的最佳模型是使用查找最佳DNA模型算法选择的。500个引导带的引导支持值显示为系统发生树中每个部分的百分比。gydF4y2Ba
数据可用性gydF4y2Ba
本研究分析的所有序列已在日本DNA数据库中注册(登录号:LC672676-LC677040)。在此研究过程中产生的所有数据都包含在这篇发表的文章及其补充信息文件中。gydF4y2Ba
参考文献gydF4y2Ba
唱,H。gydF4y2Baet al。gydF4y2Ba2020年全球癌症统计:GLOBOCAN估计了185个国家的36种癌症的发病率和死亡率。gydF4y2BaCA Cancer J. Clin。gydF4y2Ba71gydF4y2Ba, 209 - 249。gydF4y2Bahttps://doi.org/10.3322/caac.21660gydF4y2Ba(2021)。gydF4y2Ba
Johnson, n.w., Jayasekara, P. & Amarasinghe, a.a.口腔鳞状细胞癌和前驱病变:流行病学和病因学。gydF4y2Ba牙周病学2000gydF4y2Ba57gydF4y2Ba, 19-37。gydF4y2Bahttps://doi.org/10.1111/j.1600-0757.2011.00401.xgydF4y2Ba(2011)。gydF4y2Ba
Rischin, D, Ferris, R. L. & Le, Q. T.头颈部癌症进展概述。gydF4y2Baj .中国。肿瘤防治杂志。gydF4y2Ba33gydF4y2Ba, 3225 - 3226。gydF4y2Bahttps://doi.org/10.1200/JCO.2015.63.6761gydF4y2Ba(2015)。gydF4y2Ba
蒙特罗,P. H. &帕特尔,S. G.口腔癌症。gydF4y2BaSurg.杂志。中国。n。gydF4y2Ba24gydF4y2Ba, 491 - 508。gydF4y2Bahttps://doi.org/10.1016/j.soc.2015.03.006gydF4y2Ba(2015)。gydF4y2Ba
Pushalkar, S。gydF4y2Baet al。gydF4y2Ba口腔鳞状细胞癌患者肿瘤与非肿瘤组织中口腔微生物群的比较。gydF4y2BaBMC Microbiol。gydF4y2Ba12gydF4y2Ba, 144年。gydF4y2Bahttps://doi.org/10.1186/1471-2180-12-144gydF4y2Ba(2012)。gydF4y2Ba
希利,C. M. &莫兰,G. P.微生物组和口腔癌:问题多于答案。gydF4y2Ba口腔肿瘤防治杂志。gydF4y2Ba89gydF4y2Ba, 30-33。gydF4y2Bahttps://doi.org/10.1016/j.oraloncology.2018.12.003gydF4y2Ba(2019)。gydF4y2Ba
Gopinath D。gydF4y2Baet al。gydF4y2Ba口腔白斑中的唾液细菌转移类似于口腔癌细菌组的非生物性。gydF4y2Ba口腔微生物。gydF4y2Ba13gydF4y2Ba, 1857998。gydF4y2Bahttps://doi.org/10.1080/20002297.2020.1857998gydF4y2Ba(2020)。gydF4y2Ba
Pietrobon G。gydF4y2Baet al。gydF4y2Ba口腔白斑与口腔微生物群:一项综合综述。gydF4y2Ba癌症gydF4y2Ba13gydF4y2Ba, 4439年。gydF4y2Bahttps://doi.org/10.3390/cancers13174439gydF4y2Ba(2021)。gydF4y2Ba
吉尔伯特,j.a.。gydF4y2Baet al。gydF4y2Ba目前对人类微生物群的了解。gydF4y2BaNat,地中海。gydF4y2Ba24gydF4y2Ba, 392 - 400。gydF4y2Bahttps://doi.org/10.1038/nm.4517gydF4y2Ba(2018).gydF4y2Ba
威利斯,J. R. & Gabaldón, T.人类口腔微生物组在健康和疾病:从序列到生态系统。gydF4y2Ba微生物gydF4y2Ba8gydF4y2Ba, 308年。gydF4y2Bahttps://doi.org/10.3390/microorganisms8020308gydF4y2Ba(2020)。gydF4y2Ba
李,W. H。gydF4y2Baet al。gydF4y2Ba口腔微生物群的细菌改变及其与口腔癌的关系。gydF4y2Ba科学。代表。gydF4y2Ba7gydF4y2Ba, 16540年。gydF4y2Bahttps://doi.org/10.1038/s41598-017-16418-xgydF4y2Ba(2017)。gydF4y2Ba
Guerrero-Preston, R。gydF4y2Baet al。gydF4y2Ba16S rRNA扩增子测序鉴定与口腔癌、人乳头状瘤病毒感染和手术治疗相关的微生物群。gydF4y2BaOncotargetgydF4y2Ba7gydF4y2Ba, 51320 - 51334。gydF4y2Bahttps://doi.org/10.18632/oncotarget.9710gydF4y2Ba(2016)。gydF4y2Ba
胡晓霞,张强,华宏,陈芳。口腔白斑和口腔癌患者唾液微生物群的变化。gydF4y2Ba口腔肿瘤防治杂志。gydF4y2Ba56gydF4y2Ba, e6-e8。gydF4y2Bahttps://doi.org/10.1016/j.oraloncology.2016.03.007gydF4y2Ba(2016)。gydF4y2Ba
甘利,我。gydF4y2Baet al。gydF4y2Ba牙周病原体是口腔鳞状细胞癌的危险因素,与烟草、酒精和人乳头瘤病毒无关。gydF4y2BaInt。j .癌症gydF4y2Ba145gydF4y2Ba, 775 - 784。gydF4y2Bahttps://doi.org/10.1002/ijc.32152gydF4y2Ba(2019)。gydF4y2Ba
Lim Y。gydF4y2Baet al。gydF4y2Ba口腔微生物组生物标记物面板在预测口腔和口咽癌方面的表现。gydF4y2Ba前面。细胞的感染。Microbiol。gydF4y2Ba8gydF4y2Ba, 267年。gydF4y2Bahttps://doi.org/10.3389/fcimb.2018.00267gydF4y2Ba(2018).gydF4y2Ba
杨传勇。gydF4y2Baet al。gydF4y2Ba与口腔鳞状细胞癌分期相关的口腔微生物群落动态。gydF4y2Ba前面。Microbiol。gydF4y2Ba9gydF4y2Ba, 862年。gydF4y2Bahttps://doi.org/10.3389/fmicb.2018.00862gydF4y2Ba(2018).gydF4y2Ba
Sarkar, P。gydF4y2Baet al。gydF4y2Ba口腔鳞状细胞癌发展过程中口腔微生物群失调。gydF4y2Ba前面。肿瘤防治杂志。gydF4y2Ba11gydF4y2Ba, 614448年。gydF4y2Bahttps://doi.org/10.3389/fonc.2021.614448gydF4y2Ba(2021)。gydF4y2Ba
Al-Hebshi, N。gydF4y2Baet al。gydF4y2Ba炎症菌群特征gydF4y2Ba梭菌属nucleatumgydF4y2Ba而且gydF4y2Ba铜绿假单胞菌gydF4y2Ba与口腔鳞状细胞癌有关。gydF4y2Ba科学。代表。gydF4y2Ba7gydF4y2Ba, 1834年。gydF4y2Bahttps://doi.org/10.1038/s41598-017-02079-3gydF4y2Ba(2017)。gydF4y2Ba
佩蕾娜米。gydF4y2Baet al。gydF4y2Ba炎症菌群与口腔鳞状细胞癌。gydF4y2Baj .凹痕。Res。gydF4y2Ba97gydF4y2Ba, 725 - 732。gydF4y2Bahttps://doi.org/10.1177/0022034518767118gydF4y2Ba(2018).gydF4y2Ba
杨,柯。gydF4y2Baet al。gydF4y2Ba口腔鳞状细胞癌患者组织对和唾液样本的口腔微生物群分析——一项初步研究。gydF4y2Ba前面。Microbiol。gydF4y2Ba12gydF4y2Ba, 719601年。gydF4y2Bahttps://doi.org/10.3389/fmicb.2021.719601gydF4y2Ba(2021)。gydF4y2Ba
阿米尔,A,加尔文,S,希利,C. M.和莫兰,G. P.潜在恶性口腔白斑的微生物组显示富集gydF4y2Ba梭杆菌,细螺旋体,弯曲杆菌gydF4y2Ba而且gydF4y2Ba罗思氏菌属gydF4y2Ba物种。gydF4y2Ba前面。Microbiol。gydF4y2Ba8gydF4y2Ba, 2391年。gydF4y2Bahttps://doi.org/10.3389/fmicb.2017.02391gydF4y2Ba(2017)。gydF4y2Ba
赵,H。gydF4y2Baet al。gydF4y2Ba与口腔癌相关的口腔微生物群变异。gydF4y2Ba科学。代表。gydF4y2Ba7gydF4y2Ba, 11773年。gydF4y2Bahttps://doi.org/10.1038/s41598-017-11779-9gydF4y2Ba(2017)。gydF4y2Ba
张玲,刘燕,郑海娟,张春萍。口腔微生物群对口腔癌的影响。gydF4y2Ba前面。细胞的感染。Microbiol。gydF4y2Ba9gydF4y2Ba, 476年。gydF4y2Bahttps://doi.org/10.3389/fcimb.2019.00476gydF4y2Ba(2019)。gydF4y2Ba
约斯特,S。gydF4y2Baet al。gydF4y2Ba经元转录组分析发现口腔鳞状细胞癌中口腔微生物组的毒力增加。gydF4y2BaInt。口腔科学。gydF4y2Ba10gydF4y2Ba, 32。gydF4y2Bahttps://doi.org/10.1038/s41368-018-0037-7gydF4y2Ba(2018).gydF4y2Ba
李,Y。gydF4y2Baet al。gydF4y2Ba牙龈鳞状细胞癌与牙周炎之间口腔微生物群的组成与功能。gydF4y2Ba口腔肿瘤防治杂志。gydF4y2Ba107gydF4y2Ba, 104710年。gydF4y2Bahttps://doi.org/10.1016/j.oraloncology.2020.104710gydF4y2Ba(2020)。gydF4y2Ba
Segata, N。gydF4y2Baet al。gydF4y2Ba基于7个口腔表面、扁桃体、喉咙和粪便样本的成人消化道细菌微生物组组成。gydF4y2Ba基因组医学杂志。gydF4y2Ba13gydF4y2Ba, R42。gydF4y2Bahttps://doi.org/10.1186/gb-2012-13-6-r42gydF4y2Ba(2012)。gydF4y2Ba
Siegel, R. L., Miller, K. D., Fuchs, H. E. & Jemal, A.癌症统计,2021。gydF4y2BaCA Cancer J. Clin。gydF4y2Ba71gydF4y2Ba, 7-33。gydF4y2Bahttps://doi.org/10.3322/caac.21654gydF4y2Ba(2021)。gydF4y2Ba
秋山,T。gydF4y2Baet al。gydF4y2Ba一种综合分析口腔菌群的PCR新方法的建立及其在牙源性感染患者中的应用。gydF4y2Ba口腔外科,口腔医学,口腔病理。口服Radiol。Endod。gydF4y2Ba109gydF4y2Ba, 669 - 676。gydF4y2Bahttps://doi.org/10.1016/j.tripleo.2009.10.045gydF4y2Ba(2010)。gydF4y2Ba
Ikegami, H。gydF4y2Baet al。gydF4y2Ba利用新一代测序技术对呼吸道感染患者标本微生物区系分析的改进。gydF4y2Ba科学。代表。gydF4y2Ba11gydF4y2Ba, 19534年。gydF4y2Bahttps://doi.org/10.1038/s41598-021-98985-8gydF4y2Ba(2021)。gydF4y2Ba
Yatera, K., Noguchi, S. & Mukae, H.下呼吸道中的微生物群。gydF4y2Ba和。Investig。gydF4y2Ba56gydF4y2Ba, 432 - 439。gydF4y2Bahttps://doi.org/10.1016/j.resinv.2018.08.003gydF4y2Ba(2018).gydF4y2Ba
谢涛,季勇。下一代DNA测序。gydF4y2Ba生物科技Nat。》。gydF4y2Ba26gydF4y2Ba, 1135-1145(2008)。gydF4y2Ba
Haro, K., Ogawa, M., Saito, M., Kusuhara, K. & Fukuda, K.基于高精度16S rRNA基因测序分析儿童鼻分泌物的细菌组成。gydF4y2Ba科学。代表。gydF4y2Ba10gydF4y2Ba, 20193年。gydF4y2Bahttps://doi.org/10.1038/s41598-020-77271-zgydF4y2Ba(2020)。gydF4y2Ba
Belstrøm, D。gydF4y2Baet al。gydF4y2Ba显性的转录活性gydF4y2Ba链球菌gydF4y2Ba口腔多个部位的种类与牙周状况有关。gydF4y2Ba前面。细胞的感染。Microbiol。gydF4y2Ba11gydF4y2Ba, 752664年。gydF4y2Bahttps://doi.org/10.3389/fcimb.2021.752664gydF4y2Ba(2021)。gydF4y2Ba
崔,J。gydF4y2Baet al。gydF4y2Ba舌苔微生物群作为胃炎包括癌前级联的潜在生物标志物。gydF4y2Ba蛋白细胞gydF4y2Ba10gydF4y2Ba, 496 - 509。gydF4y2Bahttps://doi.org/10.1007/s13238-018-0596-6gydF4y2Ba(2019)。gydF4y2Ba
Vandeputte D。gydF4y2Baet al。gydF4y2Ba定量微生物组分析将肠道群落变化与微生物负荷联系起来。gydF4y2Ba自然gydF4y2Ba551gydF4y2Ba, 507 - 511。gydF4y2Bahttps://doi.org/10.1038/nature24460gydF4y2Ba(2017)。gydF4y2Ba
Kawanami, T。gydF4y2Baet al。gydF4y2Ba厌氧菌的更高意义:细菌性胸膜炎的克隆文库分析。gydF4y2Ba胸部gydF4y2Ba139gydF4y2Ba, 600 - 608。gydF4y2Bahttps://doi.org/10.1378/chest.10-0460gydF4y2Ba(2011)。gydF4y2Ba
Lane, d.j. 16S/23S rRNA测序。在gydF4y2Ba细菌系统核酸技术gydF4y2Ba(eds Stackebrandt, E. & Goodfellow, M.) (Wiley, 1991)。gydF4y2Ba
另,K。gydF4y2Baet al。gydF4y2Ba吸入性肺炎机械通气时菌群动态变化。gydF4y2BaBMC Pulm。地中海。gydF4y2Ba19gydF4y2Ba, 260年。gydF4y2Bahttps://doi.org/10.1186/s12890-019-1021-5gydF4y2Ba(2019)。gydF4y2Ba
埃德加,R. C.,哈斯,B. J.,克莱门特,J. C.,昆斯,C.和奈特,R. UCHIME提高了嵌合体检测的灵敏度和速度。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 2194 - 2200。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btr381gydF4y2Ba(2011)。gydF4y2Ba
Blaxter, M。gydF4y2Baet al。gydF4y2Ba使用DNA条形码数据定义操作分类单元。gydF4y2Ba费罗斯。反式。r . Soc。Lond。BgydF4y2Ba360gydF4y2Ba, 1935 - 1943。gydF4y2Bahttps://doi.org/10.1098/rstb.2005.1725gydF4y2Ba(2005)。gydF4y2Ba
黄勇,牛斌,高勇,付林,李伟,CD-HIT套件:用于聚类和比较生物序列的web服务器。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 680 - 682。gydF4y2Bahttps://doi.org/10.1093/bioinformatics/btq003gydF4y2Ba(2010)。gydF4y2Ba
王琪,王志强,王志强,王志强,王志强。基于朴素贝叶斯分类器的rRNA序列快速分类。gydF4y2Ba达成。环绕。Microbiol。gydF4y2Ba73gydF4y2Ba, 5261 - 5267。gydF4y2Bahttps://doi.org/10.1128/AEM.00062-07gydF4y2Ba(2007)。gydF4y2Ba
很好,I. J.种群频率和种群参数的估计。gydF4y2Ba生物统计学gydF4y2Ba40gydF4y2Ba, 237 - 264。gydF4y2Bahttps://doi.org/10.1093/biomet/40.3-4.237gydF4y2Ba(1953)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
Y.S.设计了这项研究,参与了样本收集、数据采集和解释、统计分析,并起草了手稿。K.H.对数据解释和统计分析做出了贡献。M.O, A.M.和m.s有助于数据解释。K.F.设计了研究,监督数据收集,对数据分析和统计分析做出了贡献。所有作者都审阅了手稿并批准其出版。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
小泽,Y,哈罗,K,小川,M。gydF4y2Baet al。gydF4y2Ba口腔冲洗、病变和舌粘膜异常患者正常部位样品微生物群的差异。gydF4y2BaSci代表gydF4y2Ba12gydF4y2Ba, 16839(2022)。https://doi.org/10.1038/s41598-022-21031-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41598-022-21031-8gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba