文摘
疟疾由疟原虫引起的,是一个主要的全球卫生挑战。全基因组测序(WGS)恶性疟原虫和间日疟原虫基因组提供洞察寄生虫遗传多样性,传播模式,可以通知临床决策和监督的目的。测序技术的进步正在帮助生成及时和大基因组数据集,应用人工智能的前景分析技术(如机器学习)支持编程疟疾控制和消除。在这里,我们评估潜在的应用深度学习卷积神经网络方法预测感染的地理起源(大陆,国家,GPS位置)使用WGS数据恶性疟原虫(n = 5957;27个国家),间日疟原虫(n = 659;13个国家)隔离。使用了高质量的全基因组单核苷酸多态性(snp) (恶性疟原虫:750 k,间日疟原虫:588 k),人口结构和祖先的分析揭示了在国家级集群。当预测位置两个物种、分类(回归相比)方法距离最低错误,国家层面和> 90%的准确率。我们的工作展示了机器学习方法的效用geo-classification疟疾寄生虫。与此前WGS数据生成更多malaria-affected跨区域、机器学习方法的性能geo-classification将改善,从而支持疾病控制活动。
介绍
疟疾造成的疟原虫寄生虫和由按蚊传播的,仍是一个紧迫的全球卫生问题,死亡率和发病率负担严重集中在不到五岁的儿童。的发病率和死亡率的影响恶性疟原虫疟疾主要是集中在撒哈拉沙漠以南的非洲地区,而负担的间日疟原虫是觉得在亚洲和南美洲最严重1。之间的复杂协同进化的历史疟原虫寄生虫,人类,按蚊是包含在每个生物的基因组中,和基因组的工具和数据的关键重要性理解疟疾的基本遗传基础,其地理空间分布和控制策略来消除它。有一个快速增长的数量恶性疟原虫和间日疟原虫分离DNA发生了全基因组测序(WGS),在基因组技术的持续进步可能加速到及时生成数据集从临床和监测血液样本告知疾病流行病学和控制。
富人WGS数据中包含的信息可以用来推断传输模式,耐药性检测,支持更广泛的疟疾控制项目和消除策略2,3。WGS数据结合人口基因组方法可以检测选择性扫描与耐药性和推断的地理起源相关的感染,包括导入或如果找到感染耐药,并相应地调整治疗是否应该。众所周知,疟疾寄生虫主要基于地理人口结构4,5。几个有用的分子条形码对物种形成和地理已经开发出来2,3,但通常这些条形码没有使用整个基因组的高维数据和相关的计算成本3。然而,机器学习(人工智能的一个分支,它),合并和分析的能力很大,高维数据集在一个有效的方式,似乎可能适合geo-predicting使用WGS数据。机器学习可以应用分类,涉及预测一个标签(如国家、大陆地区)和回归,包括预测数量(例如,经度或纬度)。
机器学习应用有效地在疟疾研究各种各样的问题,包括进化选择的检测与耐药有关6,7寄生虫的分类和检测红细胞8,9,10,11和抗疟药物发现12。深度学习是机器学习的一个子集算法提取目标和学习一系列分层表示,通常利用大量数据。深度学习的应用,特别是神经网络,在群体遗传学探索过13,14,包括其他病原体15,16。开拓性工作也表明,机器学习,包括深度学习卷积神经网络(cnn),可以用来预测地理位置从人类,蚊子恶性疟原虫遗传变异17、构建方法和使用大型基因分型芯片或WGS人口结构评估18,19。在这里,我们的目标是进一步扩大应用的geo-prediction疟原虫通过使用一个非常大的数据集的隔离全球采购,(恶性疟原虫n = 5957, 27个国家;间日疟原虫,13个国家,n = 659) 11个地区(东南亚(海),南部海(SSEA)、南亚、南美、西非、非洲中部,南部非洲中部,东部非洲,非洲之角,南部非洲,大洋洲)。我们探索潜在的常规机器学习方法旨在学习表示从序列和地理数据,以及深度学习方法,旨在学习和提取层分层表示的SNP组合与地理位置有关。我们比较四个一般应用方法,包括分类方法,预测位置,随后插入特定的坐标,以及比较性能跨地域(国家)都包括在这些观察和排除他们从训练集用于开发模型。
材料和方法
原始测序数据的处理
公开原始Illumina公司(> 150个基点)配对序列数据先前发表的研究恶性疟原虫和间日疟原虫从ENA存储库下载(见S1表和S2加入数据表),并伴随着元数据包括取样的位置(见S1表和S2纬度和经度坐标表)。数据包括公共原始序列和GPS数据MalariaGEN项目(www.malariagen.net)。生WGS数据恶性疟原虫(n = 5957)间日疟原虫(n = 659)是一致的Pf3D7(v3)和PvP01(v1)参考基因组,分别使用bwa-mem软件(v0.7.12)使用默认参数设置(例如,关于不匹配和顺序阅读剪报处罚;看到http://bio-bwa.sourceforge.net/bwa.shtml)。fixmate samtools (v1.9)功能和markdup产生的BAM文件调用一组潜在的变体20.。变体质量控制、校准进行了评估使用GATK的BaseRecalibrator和ApplyBQSR功能,基准测试了已知的高质量变异遗传交叉恶性疟原虫5,21和之前策划数据集间日疟原虫20.。修订组单核苷酸多态性和插入/删除(indels)被称为与GATK HaplotypeCaller(4.1.4.1版)使用选项erc GVCF5,22。变量被分配一个质量分数使用GATK变体质量分数调整(VQSR),和那些VQSLOD分数< 0,代表变异比真实更有可能是假的,被过滤掉7,22。此外,单核苷酸多态性被移除,如果他们失去了10%以上的等位基因7,22,生成的数据集组成的寄生虫的基因组恶性疟原虫(5957隔离,750 k SNPs)和间日疟原虫(659隔离,588 k SNPs)。人口结构是由主成分分析(PCA)的孤立国民党之间的差异。同时,掺合料分析23进行跨地理了解祖先群体的构成,在最优数量的组(K)成立使用交叉验证值介于1和20。这种交叉验证分析导致10祖先群体恶性疟原虫和间日疟原虫(K = 10)。
统计模型和性能
使用机器学习(ML)和深度学习(DL)统计模型,目标是在一个位置使用单核苷酸多态性预测地理来源(GPS),国家和地区解决。我们应用两个标准模型分类在国家和地区层面上:(1)惩罚多项逻辑回归分类器(LOG-C;毫升);(2)CNN (CNN-C;DL)。随后,我们使用了预测概率放在不同的位置来执行这些位置之间的加权插值和GPS坐标水平做出预测。
特别是,最后预测位置(经度和纬度)是由分类器的加权平均预测,把重量放在哪里的概率的模型在每个位置。
我们还应用GPS坐标的两个回归模型预测:(3)惩罚线性回归模型(LIN-R;毫升);(iv) CNN (CNN-R;DL)。LOG-C和LIN-R模型的正则化强度C调在L1点球(套索)和实现sklearn Python包(https://scikit-learn.org)。惩罚参数调谐使用交叉验证(见下文,S3表)。深度学习CNN架构实施使用Keras库(2.2.4版)24在Python中。CNN模型有一个架构soft-max预测层通过辍学、正规化25为了防止过度拟合和支持可转让性。4的主要模型有一个卷积层过滤器,与各自的过滤器的大小(40岁,9)后跟两个退学和致密层与ReLu激活(类似于17),应用随机梯度下降算法的优化。我们的模型训练和验证1000时代。模型的参数化是总结(S3表)。我们创建了一个数据集分层三倍分裂(80%的训练,10%的验证,测试10%)模型,并使用旨在验证数据集参数(S3表)。LOG-C和LIN-R模型旨在(分层、4倍)的正则化强度C L1点球。报告的分数(准确性、平均加权距离误差)的计算是通过合作测试集(见所做出预测S3最后一个参数设置表)。此外,我们进行了一次“leave-one-geography-out”,每一个地理的训练数据集反过来,省略了与模型训练其余地域上,理解普遍性对以前看不见的地方26。
分类精度确定后分配预测经度和纬度对个别国家。分类模型,平均(加权)距离误差计算使用半正矢方法允许(角)沿着球面距离计算,根据不同的实际和估计的位置。后者是由分类器的加权平均预测,把重量放在哪里的概率的模型在每个位置。标签的准确计算是基于预测与测试数据。特别是,用天真的预测基于基线精度最常见的国家将为18.8%恶性疟原虫(柬埔寨)和24.3%间日疟原虫(泰国)。回归模型的误差计算使用半正矢方法基于预测之间的差异和实际使用角距离纬度和经度。
结果
疟疾分离序列数据和人口结构
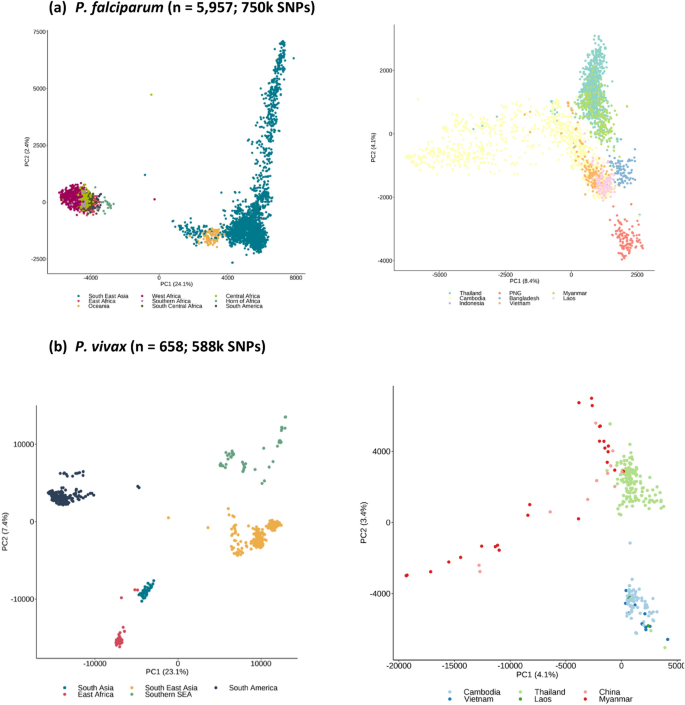
原始WGS数据伴随地理起源信息是可用在公共领域恶性疟原虫27个国家(n = 5957)间日疟原虫13个国家(n = 659)(表1),它代表每个寄生虫的全球分布。大多数恶性疟原虫隔离是来自海(2648 44.5%),其次是西非(2042 34.3%)和东非(451年7.6%)。同时,对于间日疟原虫,隔离大部分是来自海(282 42.9%)其次是南美洲(220 33.4%)和SSEA(48)(表1)。分别通过分析每个物种,发现高质量的全基因组单核苷酸多态性在隔离(恶性疟原虫750 k单核苷酸多态性,间日疟原虫588 k SNPs)。大多数snp微小等位基因频率较低(SNPs加< 1%:恶性疟原虫94.6%,间日疟原虫77.6%)(S1图)。大多数单核苷酸多态性基因的地区(恶性疟原虫76.5%,间日疟原虫54.3%),高比例的非同义(NS)氨基酸的变化(恶性疟原虫63.0%,间日疟原虫42.5%)。之间的遗传多样性恶性疟原虫隔离相对均匀在27个国家(SNPπ:中位数0.037,0.027 - -0.053),并降低幅度比间日疟原虫的数据是来自13个国家(SNPπ:中位数0.056,范围0.037 - -0.066)(表1)。
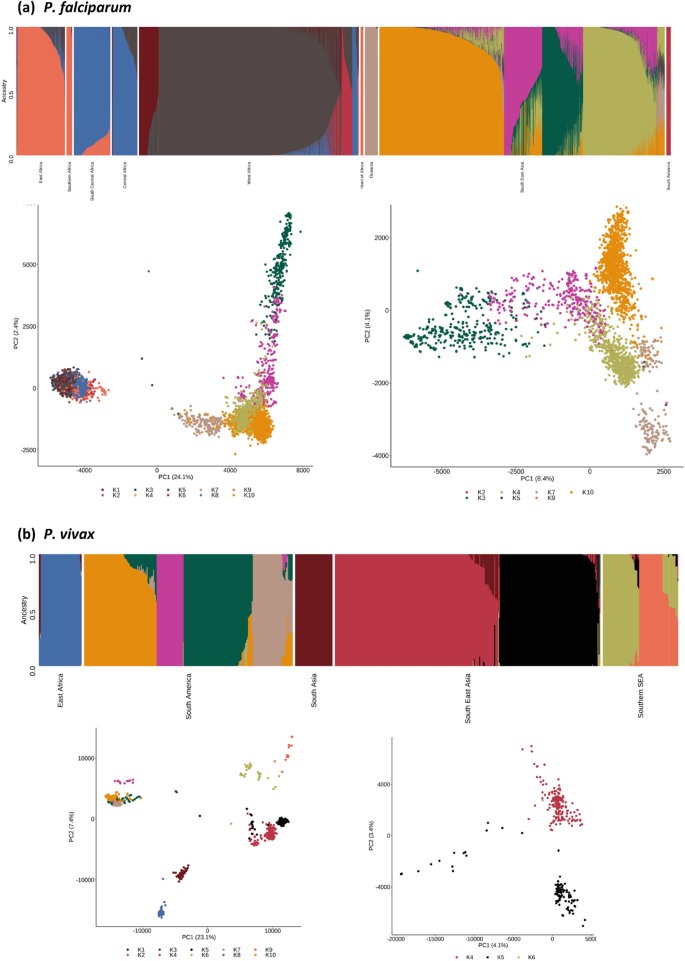
无监督聚类方法应用于每一个物种的全基因组单核苷酸多态性,揭示他们的人口结构和关联的程度(伪)祖先的模式。主成分分析(PCA)的恶性疟原虫和间日疟原虫由大陆隔离显示预期的分离,明确证据的人口结构在区域和国家层面(无花果。1)。人口结构和祖先使用掺合料的分析软件23确定数量的祖先组(恶性疟原虫K = 10,间日疟原虫K = 10),为每个隔离及其相对丰度估计(无花果。2)。为恶性疟原虫占主导地位,有祖传的组织在地区和大陆(非洲4,海4、大洋洲,南美洲1),一些证据的混合血统(例如,海洋隔离3祖先群体),但一般的一致性在国家。为间日疟原虫,占主导地位的祖先群体的数字区域不同于恶性疟原虫(南美洲4、海洋2,SSEA 2,东非,南亚1),由于采样和疟原虫物种地方的特性差异,如缺乏附近间日疟原虫在非洲。总的来说,有更多的同质性祖先的团体间日疟原虫隔离,一些团体广泛与邻国(比较图。1)。这些分析证实,spatial-genomic使用WGS数据聚类和分类是可行的。
geo-classification模型的应用
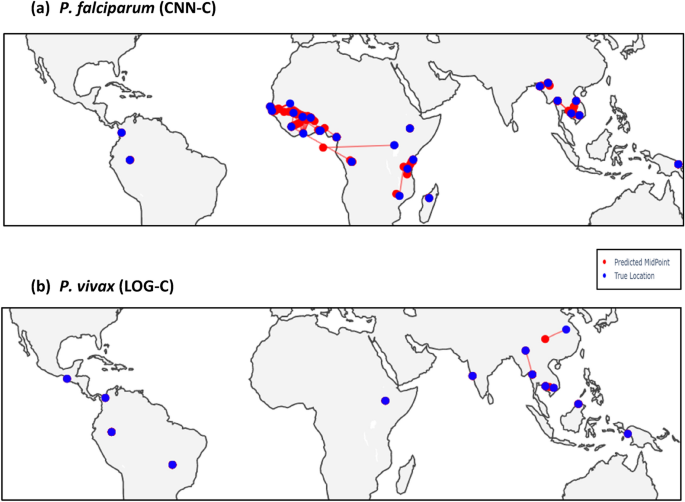
为恶性疟原虫,分类方法的预测性能(LOG-C CNN-C)是比回归模型(LIN-R CNN-R)在区域(表2)和遍及全国的(表3)分析(平均距离(公里):错误470年LIN-R LOG-C 93年CNN-R 245年CNN-C 77)。地点包括在训练数据集,分类模型的性能接近100%在地区层面上,和接近90%在国家层面(S4表,S5表)。最贫穷的性能模型是对非洲人口,例如,CNN-C的平均距离误差是在西非(267公里)和东部非洲国家(117公里,尤其是肯尼亚和乌干达),以及马拉维(530公里)(表3),相对于其他地区。这个观察是一致的复杂的祖先在非洲人口(无花果。2),以及另一个深度学习分析17。正如预期的那样,我们国家没有在数据使用的培训模型预测,错误的距离(公里)至少~ 5倍大(1983年CNN-R LIN-R 2246, LOG-C 1848, CNN-C 1540),与最贫穷的预测秘鲁(表4)。表现最好的模型在此设置是CNN-C分类器(无花果。3)。
为间日疟原虫分类方法的预测性能(LOG-C CNN-C)也优越而回归模型(LIN-R CNN-R)跨区域(表2)和遍及全国的(表3)分析(平均距离(公里):错误890年LIN-R LOG-C 33岁CNN-R 819年CNN-C 36)(表3)。地点包括在训练数据集,分类模型的性能接近100%在区域和国家层面上,与邻国中国和缅甸(最穷的性能S4表,S5表)。(意味着)距离误差的不习惯的国家发展模式是独特的大(公里:LIN-R 1481, 2508年LOG-C CNN-R 2512年CNN-C 2405),与最贫穷的预测埃塞俄比亚和秘鲁(表4)。表现最好的模型在此设置是一个LIN-R回归(无花果。3)。
讨论
WGS数据疟原虫寄生虫可以检测进口感染、耐药和传播模式,从而协助决策在临床和疟疾控制设置。与WGS赢得人心的实现卫生系统,有机会实现统计学习方法协助监测活动。一个清晰的用例包括隔离的地理起源的决心,见解的基础上从以前的工作表明,基因组数据可以用于通过地理集群寄生虫2,3,4,5。我们的工作表明,机器学习方法,特别是关注分类(例如,深度学习cnn),有可能准确预测地理位置在GPS和国家层面的决议。正如所料,性能更强的孤立的地理起源已经表示在国家层面的数据集,证明WGS需要实现更广泛的遗传多样性填补国家空白。是最弱的预测恶性疟原虫在非洲西部和东部,共同的血统,混合感染,运动的人、耐药性和疟疾地方的特性可以复杂的遗传多样性分析。距离错误类似于前一个机器学习的分析恶性疟原虫(中位数< 20公里),它实现了一个深度学习的方法在一个较小的数据集17。CNN的分类方法似乎表现良好在疟原虫物种中,实施措施,以减少过度学习的影响,及其与更大的性能可能会提高隔离取样和WGS数据。
当我们实现了一组有限的机器学习方法,还有测试范围的选择方法(例如,梯度增加了树木,支持向量机)16或进一步优化我们的模型parametrisations(除了默认设置)来提高性能。例如,尽管L1-penalized回归方法通常很竞争,稳定选择套索的顶部通常导致改进27。此外,生成的模型是白盒,导致一组可判断的snp。cnn是最利用深度学习网络类型,和以超越替代方法28。然而,CNN模型的一个限制是他们的“黑盒”性质,几层组成的一个复杂的体系结构,在我们的上下文(和其他人17)难以建立(组合)单核苷酸多态性是地理分析的信息。其他研究已经使用人口基因组方法确定信息SNPs,重点应用pcr检测或资源贫乏国家的扩增子测序2,3。我们提供计算机代码实现模型,协助未来评估仿真或实证研究。未来工作的重点应该放在开发一个在线“geo-locator”工具,揭示了一个位置的预测,可以评估其合理性与实际位置,如果已知,反馈到模型构建和学习过程。这样一个框架也可以扩展集成显式的抗药性标记29日为疟疾病媒,以及基因组数据17,使用序列生成的便携式和现场部署的测序平台(例如,牛津纳米孔技术的奴才)。这些工具将直接在流行国家疟疾控制项目的价值,包括那些正在实施消除那些想要区分本地收购或进口的活动感染。它还将帮助这些国家疟疾负担较低,包括通过进口寄生虫的检测可能威胁消灭疟疾的目标。
总之,我们的研究表明,机器学习方法可以发挥有益的作用在确定的地理起源WGS隔离,从而为控制和监测活动提供重要的见解。此外,这样的方法将可伸缩当WGS常规和成本有效,导致环境越来越“大数据”是用于决策。这个“学习”系统的效用将会改善随着时间的推移,基础方法和模型性能改善与更多的数据变得可用,他们中实现信息化工具来协助监测和临床决策。这个实用程序强调的好处使测序数据和地理信息公开有关全球数据库更及时的方式理解感染动态,这也被证明了的优点COVID-19危机。
结论
测序技术的进步正在实时genomics-informed监测和临床管理成为现实。结果大基因组数据集,我们的研究表明,机器学习方法,人工智能的一个子集,可以准确地预测疟疾寄生虫的地理来源从序列数据。与更大的地理范围和信息基础设施,这种方法将改善性能和协助疟疾控制和消除活动。
数据可用性
来自欧洲的原始WGS数据可用核苷酸存档(ENA)(见S1表和S2表为项目加入数字)。可从计算代码和机器学习模型https://github.com/WDee/GeoComparison。
引用
世界卫生组织。世界疟疾报告》(2020)。
普雷斯顿,m D。et al。organellar基因组多态性的条码识别恶性疟原虫株的地理起源。Commun Nat。51 - 7 (2014)。
DiezBenavente E。et al。分子条形码通知的地理起源和传播动力学间日疟原虫疟疾。公共科学图书馆麝猫。16e1008576 (2020)。
商业部副部长Diez Benavente上周E。et al。独特的遗传结构和选择模式从南亚和非洲东部间日疟原虫。Commun Nat。121 - 11 (2021)。
Samad H。et al。Imputation-based群体遗传学分析恶性疟原虫疟疾寄生虫。公共科学图书馆麝猫。11e1005131 (2015)。
Pybus M。et al。层次提高:机器学习框架努力检测和分类有选择性的在人群中扫。生物信息学31日493 (2015)。
Deelder, W。et al。使用深度学习在疟原虫序列数据识别最近积极的选择。颧骨。J。20.1 - 9 (2021)。
全,Q。,Wang, J. & Liu, L. An effective convolutional neural network for classifying red blood cells in malaria diseases.Interdiscip。科学。第一版。生命科学。12,217 - 225 (2020)。
梁,Z。et al。疟疾CNN-based图像分析诊断。:Proc - 2016 IEEE Int。Conf Bioinforma。生物医学。BIBM 2016493 - 496 (2017)。https://doi.org/10.1109/BIBM.2016.7822567。
Poostchi, M。,Silamut, K., Maude, R. J., Jaeger, S. & Thoma, G. Image analysis and machine learning for detecting malaria.Transl。Res。194年36-55 (2018)。
Fuhad, k . m . F。et al。基于深度学习的自动疟疾寄生虫检测血涂片及其基于智能手机的应用程序。诊断10329 (2020)。
七巧板,b . J。et al。深入学习驱动研究药物发现:解决疟疾。公共科学图书馆第一版。医学杂志。16e1007025 (2020)。
来自um L。,Brandvain, Y. & Schrider, D. R. The unreasonable effectiveness of convolutional neural networks in population genetic inference.摩尔。杂志。另一个星球。36,220 - 238 (2019)。
桑切斯,T。卷曲,J。,Charpiat, G. & Jay, F. Deep learning for population size history inference: design, comparison and combination with approximate Bayesian computation.bioRxivhttps://doi.org/10.1101/2020.01.20.910539(2020)。
Deelder, W。et al。机器学习预测准确的结核分枝杆菌耐药性从全基因组测序数据。前面。麝猫。10922 (2019)。
Libiseller-Egger, J。费兰,J。,Campino, S., Mohareb, F. & Clark, T. G. Robust detection of point mutations involved in multidrug-resistant mycobacterium tuberculosis in the presence of co-occurrent resistance markers.公共科学图书馆第一版。医学杂志。16e1008518 (2020)。
电池,c·J。,Ralph, P. L. & Kern, A. D. Predicting geographic location from genetic variation with deep neural networks.Elife922页(2020)。
第5期,G。,Jónsson, H., Hinge, A., Manchih, N. & Orlando, L. Accurate continuous geographic assignment from low- to high-density SNP data.生物信息学32,1106 - 1108 (2016)。
Bhaskar,。,Javanmard, A., Courtade, T. A., Tse, D. & Valencia, A. Novel probabilistic models of spatial genetic ancestry with applications to stratification correction in genome-wide association studies.生物信息学33,879 - 885 (2017)。
李,h .改善SNP发现基地对齐质量。生物信息学27,1157 - 1158 (2011)。
英里,一个。et al。Indels、结构变化和复合驱动恶性疟原虫基因组多样性。基因组Res。26,1288 - 1299 (2016)。
Benavente, e D。et al。基因变异在间日疟原虫疟疾揭示区域选择性压力下。《公共科学图书馆•综合》12e0177134 (2017)。
亚历山大·d·H。,Novembre, J. & Lange, K. Fast model-based estimation of ancestry in unrelated individuals.基因组Res。19,1655 - 1664 (2009)。
Chollet F。,&others. Keras. GitHub. Retrieved fromhttps://github.com/fchollet/keras(2015)。
斯利瓦斯塔瓦,N。辛顿,G。,Krizhevsky, A. & Salakhutdinov, R. Dropout: A simple way to prevent neural networks from overfitting.j·马赫。学习。Res。15,1929 - 1958 (2014)。
Mordelet f &绿色,j.p. ProDiGe:疾病基因和多任务的优先级机器学习从积极和未标记示例。BMC Bioinform。121 - 15,(2011)。
马希,p . & Tournoud m .预测细菌耐药性选择使用k-mers从全基因组序列与稳定。BMC Bioinform。191 - 11 (2018)。
Alzubaidi, L。et al。深度学习的回顾:概念、CNN架构挑战,应用,未来的发展方向。j .大数据81 - 74 (2021)。
Turkiewicz,。et al。遗传多样性的恶性疟原虫GTP-cyclohydrolase 1,二氢叶酸还原酶和dihydropteroate合成酶基因揭示磺胺多辛-乙胺嘧啶抗疟药物耐药性的新见解。公共科学图书馆麝猫。16e1009268 (2020)。
确认
TGC由英国医学研究理事会资助(批准号先生/ M01360X / 1 / N010469/1先生/ R025576/1先生/ R020973/1先生和先生/ X005895/1)资助。SC是由BloomsburySET和英国医学研究理事会资助(先生/ M01360X / 1 / R025576/1先生/ R020973/1先生和先生/ X005895/1)。我们感谢阿列克谢尚未提供支持Python代码。
作者信息
作者和联系
贡献
W.D.,S.C., L.P., and T.G.C. conceived and designed the study. E.M. and J.E.P. performed the bioinformatic processing of the raw sequencing data. W.D. and E.M. performed the population genetic and statistical analysis, under the supervision of S.C., L.P. and T.G.C. W.D. wrote the first draft of the manuscript. All authors commented on and edited the manuscript and approved the final version. W.D. and T.G.C. compiled the final manuscript.
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
出版商的注意
施普林格自然保持中立在发表关于司法主权地图和所属机构。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
Deelder, W。Manko E。,Phelan, J.E.et al。地理分类疟原虫通过机器学习应用到整个基因组序列数据。Sci代表1221150 (2022)。https://doi.org/10.1038/s41598 - 022 - 25568 - 6
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598 - 022 - 25568 - 6