摘要
PmlR2是一类LitR/CarH家族转录调节因子,PmSB-LOV是一种“短”lov型蓝光感受器,相邻编码在其中假单胞菌mendocinaNBRC 14162。一种“短”lov型光感受器的效应蛋白假单胞菌目前尚未确认。在这里,我们发现PmlR2是PmSB-LOV的效应蛋白。转录分析显示,附近基因的表达pmlR2它的同源基因,pmlR1,是对光照的反应。体外dna -蛋白质结合分析表明,重组PmlR2直接结合到光诱导基因的启动子区。此外,PmSB-LOV表现出典型的lov型光诱导光谱变化。凝胶过滤色谱表明,发光的PmSB-LOV与PmlR2直接相关,而非发光的蛋白质不相互作用。PmSB-LOV结合后PmlR2功能的抑制通过表面等离子体共振验证:PmlR2的dna结合能力在蓝光照射的PmSB-LOV存在下被特异性抑制。体外转录实验显示,在发光的PmSB-LOV存在时,PmlR2抑制因子活性的剂量依赖性降低。总的来说,有证据表明PmlR2的dna结合活性被其与蓝光激活的PmSB-LOV的直接结合所抑制,从而通过RNA聚合酶实现光诱导启动子的转录。
简介
LitR(光诱导转录;/CarH蛋白家族是含有c端维生素B的mre型转录调控因子12绑定域名。LitR/CarH在几种细菌的光诱导类胡萝卜素生产中起着核心作用,包括Myxococcus克桑托斯1;一种革兰氏阴性的滑翔细菌,色链霉菌A3 (2)2;革兰氏阳性抗生素生产商,芽孢杆菌megateriumQM B15513.;低gc内孢子形成革兰氏阳性细菌;而且栖热菌属酸奶HB274是一种嗜热的革兰氏阴性细菌。在此之前,我们揭示了litr介导的光诱导转录的分子机制b . megateriumQM B15513..的LitRb . megaterium与腺苷B有关12(AdoB12),并作为负转录调节因子。光照下,LitR蛋白被AdoB光解灭活12,由于Co-C键的光敏性,阻止了dna结合活性3..类似的光响应机制涉及LitR已在t .酸奶1而且Myxococcus克桑托斯5.这些研究表明,LitR/CarH家族成员在系统遗传多样性的非光养细菌中作为光依赖调节因子,主要参与光诱导类胡萝卜素的产生1,6,7.
多个基因组序列的完成揭示了LitR同源物在不同的细菌基因组中广泛分布,包括革兰氏阳性和革兰氏阴性细菌8.最近,我们报道了基于c端结构域氨基酸序列的系统发育分析,至少有5类LitR/CarH家族蛋白(指定class I - V LitR)8,与n端HTH dna结合域相反,它们已经分化。通常,mre型调控蛋白在n端具有hth - dna结合结构域,在c端具有效应子结合结构域。如上所述,I类litr是adb12基于光感调节器。II类litr主要见于属内假单胞菌只保留4螺旋束结构域,即两个B中的一个12-binding域,这意味着它们没有能力与B . binding域相关联129.III、IV、V类litr主要分布于本属洋葱8,弧菌,微球菌的,分别。尽管III类LitR的c端与功能域没有相似之处,但重组蛋白的吸收光谱发生了依赖于光的变化,这表明III类LitR具有一种新的光敏感机制8.IV类和V类升不含B12绑定域,其功能未知。这些结果表明,非光养细菌中的LitR/CarH家族蛋白具有对光的感知和反应能力,具有不同的光感机制。
光、氧或电压(LOV)蛋白含有黄素单核苷酸(FMN)发色团,在植物、真菌和细菌中多种蓝光依赖性细胞功能的调节中发挥重要作用10.细菌LOV蛋白主要根据效应结构域的存在与否分为两类,效应结构域将光信号传递给其他结构域或蛋白,如转录调控因子或激酶11.在保留效应域的LOV蛋白中,研究了光感受器结构域和效应域之间的关系12,13,14,15.缺乏效应子结构域的LOV蛋白被称为“短”LOV。以前也研究过三种“短”细菌lov的光化学:PpSB1-LOV16,17,18和PpSB2-LOV18,19的p . putidaKT2440, RsLOV的Rhodobacter sphaeroides这是一种紫色的革兰氏阴性光合细菌20.的DsLOVDinoroseobacter shibae是一种海洋光养细菌21.然而,目前还没有确定这些“短”恋人的绑定伙伴。
假单胞菌putidaKT2440是一种革兰氏阴性土壤细菌,具有多个LitR同源物(PplR1-PplR3),被指定为II类LitR9.我们最近报道了三个pplR这些基因协同参与19个光诱导基因的表达,包括编码PplR1的基因,PhrB(编码DNA光解酶),UfaM(含呋喃脂肪酸合成酶),FolE (GTP环水解酶I), CryB(隐花色素样蛋白),以及多个没有注释/已知功能的基因9.基因敲除的ppSB1-LOV而且ppSB2-LOV在黑暗和光照条件下,ppsb - lov都能引起PplR依赖基因的低水平转录,这表明ppsb - lov通过一种未知机制控制PplR活性9.
我们之前的研究表明ppSB-LOV基因控制pplr依赖的光诱导转录9;然而,这一机制的体外分析面临局限性。例如,重建蛋白质-蛋白质相互作用受到制备可溶性和无标记的PplR重组蛋白的困难的阻碍。然而,我们预计,进一步阐明由“短”LOV蛋白和MerR家族蛋白介导的光调节机制将为细菌光反应机制提供新的见解。在这里,我们发现重组蛋白来源于假单胞菌mendocinaNBRC 14162可稳定纯化为可溶性蛋白,并研究了PmlR2 (假单胞菌mendocina光诱导转录调控因子),PplR的同源物,PmSB-LOV (假单胞菌mendocinaSensory box-LOV),是PpSB-LOV的同源物。本研究表明PmlR2的dna结合活性受PmSB-LOV通过光依赖性蛋白-蛋白相互作用负向控制。我们提供了第一个生物化学证据,表明一个mre型转录调节因子是细菌中“短”LOV型蓝光感受器的效应蛋白。
结果
pmlR而且pmSB-LOVmendocina假单胞菌NBRC 14162基因
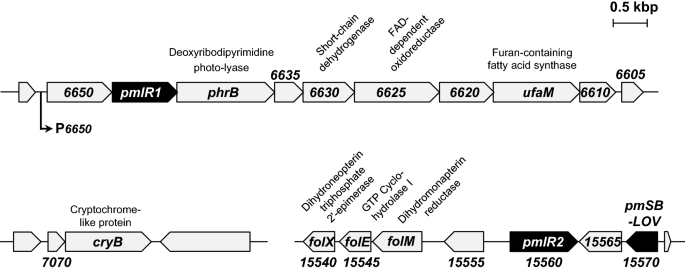
p . mendocinaNBRC 14162是一种革兰氏阴性菌,以乙醇为碳源富集土壤分离得到22.数字1显示了两个基因组组织的示意图pplR同源染色体(指定pmlR1而且pmlR2的同系词ppSB-LOV(指定pmSB-LOV)p . mendocinaNBRC14162。
的pmlR1该区域包含编码脱氧核糖二嘧啶光解酶(PhrB)、短链脱氢酶(PME6630)、fad依赖性氧化还原酶(PME6625)、含呋喃脂肪酸合成酶(UfaM)和假设蛋白质(PME6650、PME6635、PME6620和PME6610)的基因。的pmlR1地区是高度保守的假单胞菌。以及其他近亲细菌9.的pmlR2地区和pmlR1这种细菌中的簇在不同的位点上编码,类似于基因组测序p . mendocinaNK-0123和ymp24.的pmlR2该区域包括编码PmSB-LOV、二氢蝶呤还原酶(FolM)、GTP环水解酶I (FolE)、三磷酸二氢蝶呤2′-外素酶(FolX)和一个假设蛋白(PME15555)的基因。FolM、FolE和FolX可能包含一个操纵子,参与叶酸和蝶呤的生物合成25.基因簇,由pmlR2,PME15565,pmSB-LOV基因,是保守在染色体上的假单胞菌spp。
的pmlR2该基因编码321个氨基酸的蛋白质(计算分子量为35.82 kDa),与的PplR3、PplR2和PplR1的同源性分别为56%、39%和34%p . putida,分别(图;S1相比之下,PmlR1 (318 aa, 35.56 kDa)与PplR1、PplR3和PplR2的氨基酸同源性分别为45%、36%和28%。PmlR2与PmlR1在整个序列中具有较低的同源性(26.4%),但其n端mre型HTH dna结合域具有70%的相似性。这种HTH结构域的高度相似性在PplR蛋白中也被观察到。S1A). Pfam搜索显示PmlR1和PmlR2的内部区域,Arg残留106对参数179(PmlR1)和Trp113将低浓缩铀161(PmlR2),与B有相似之处12-binding_2域(PF02607E-值分别为1.1e-05和3.5e-07。两种PmlR蛋白的c端与已知功能蛋白结构域没有相似之处。一般有两个域,B12绑定域和B12-binding_2域,是与B关联所必需的12这表明pmlr没有能力与B形成络合物12.
PmSB-LOV由148个氨基酸(16.68 kDa)组成,包括一个典型的缺乏效应子结构域的“短”LOV蛋白。PmSB-LOV与的MDS_3586具有100%的同一性p . mendocinaNK-01(无花果。S1的PpSB2-LOV(71%)和PpSB1-LOV(69%)的端到端相似性p . putidaKT2440。与FMN形成硫醇加合物所需的保守半胱氨酸残基也出现在53位(指定为Cys)53)的PmSB-LOV蛋白。在PmSB-LOV中也发现了在其他“短”lov中发现的保守的n端n帽和c端j α-螺旋(图2)。S1B)。
我们关注PmlR2和PmSB-LOV,因为它们在基因组中很接近p . mendocina和NBRC 14162可能有很强的功能关系。
光诱导转录p . mendocinaNBRC 14162
我们进行了RNA-seq分析,以初步筛选在蓝光下表达发生变化的基因。在30.5 μ mol s的暗光和蓝光条件下,从液体培养基中培养的细胞中纯化总RNA−1米−2.2倍以上的光诱导基因见表S1.所鉴定的光诱导基因普遍存在于p . putidaKT2440,正如我们之前报道的那样9.
为了确认RNA-seq分析的结果,我们进行了定量RT-PCR (qRT-PCR)。转录pmlR1,phrB,ufaM在光照条件下要高得多(表1).转录的pmlR2而且pmSB-LOV在黑暗和光照条件下都是构成的。因此,许多基因位于侧翼的区域pmlR1以一种依赖于照片的方式强烈转录,而pmlR2在光照条件下略有上调。phrB而且ufaM在p . putidaKT2440同样受到PplRs的调控,这表明这些基因在该细菌中受到PmlR1和PmlR2的调控。
我们根据RNA-seq和qRT-PCR分析的结果预测了转录单位。九个基因(PME6650来PME6610)、两个基因(PME7070而且cryB),和三个基因(folM,folE,folX)可能包含一个多顺子,由第一个基因之前的启动子转录,因为RNA-seq分析检测到它们的转录水平在光照响应下显示出类似的折叠变化(表2)S1).从操纵子的第一个基因开始的每个下游编码序列的假定翻译起始密码子与前一个编码序列的终止密码子重叠或接近。
重要的是要了解在体内的功能pmlR而且pmSB-LOV基因在p . mendocinaNBRC 14162。然而,我们无法利用几种类型的载体,包括宽宿主范围质粒(pBHR1)和基因组整合转座子(pUTminiTn5),通过共轭转移或电穿孔建立转化方法。因此,我们主要对PmlR2和PmSB-LOV进行体外分析。
启动子结构先于光诱导PME6650基因
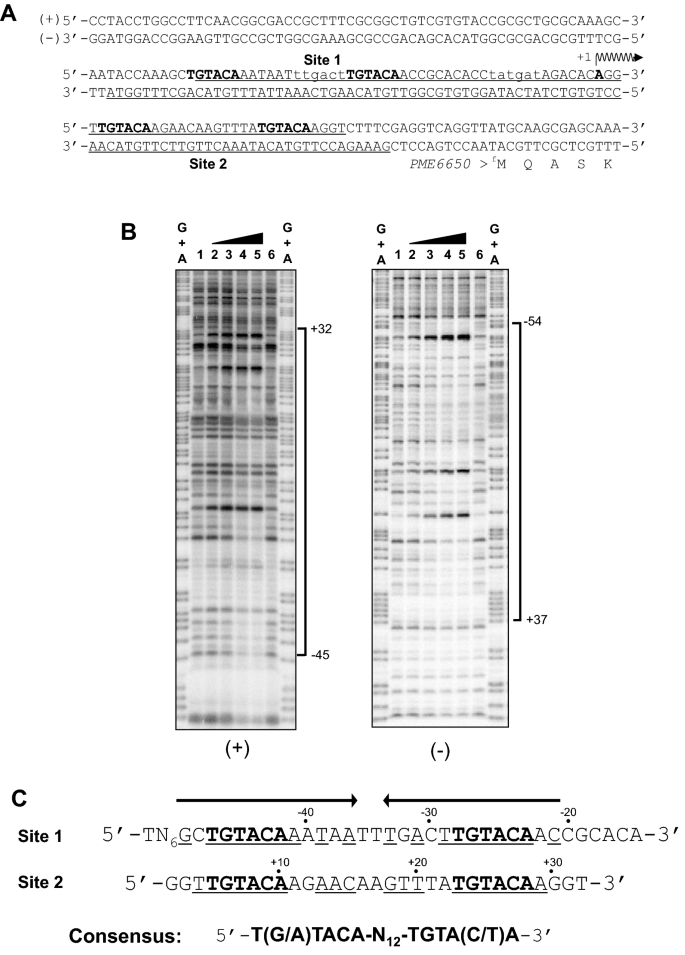
的转录起始位点PME6650发起人(指定P6650)p . mendocinaNBRC 14162和PP0468(指定P468)p . putidaKT2440作为对照启动子,我们进行了5 ' -RACE和CAGE实验。P6650和P468位于ATG翻译起始密码子上游48 bp和51 bp(图。2A). P .的−35和−10区域6650(TTGACTN16TATGAT)与研究的一致序列高度相似大肠杆菌σ70-依赖启动子(TTGACAN16-18TATAAT)26.根据转录起始位点的位置,我们注释了不同的翻译起始密码子PME6650在p . mendocina与NBRC 14162相比p . mendocinaNK-01。
DNase I足迹分析测定pmlr2结合位点。(一个) P .核苷酸序列6650.pmlr2结合位点有下划线。共识(5 ' -T(G/A)TACA和TGTACA)p . putidapplr1绑定用粗体字母表示,包括Site 1和Site 2。P6650,由5 ' -RACE和CAGE决定,由+ 1和排气箭头表示。假定的−10和−35六聚体序列用小写字母表示。(B) dna酶印迹试验。一个pmlr2结合位点在P的正义(+)和反义(−)链上6650是受保护的。重组PmlR2的用量分别为0 pmol(1、6通道)、10 pmol(2通道)、20 pmol(3通道)、40 pmol(4通道)和80 pmol(5通道),以10 kcpm的速度添加到反应混合物中32p标记DNA探针。进行了三个独立的技术重复实验,并给出了具有代表性的数据。全长凝胶的原始和未加工版本包括在无花果中。S10而且S11.(C)两个pmlr2结合位点与p . putidaPplR1共识。
我们首先用凝胶位移法评估了重组PmlR2蛋白的dna结合活性。PmlR2表达为谷胱甘肽- s -转移酶(GST)融合蛋白大肠杆菌并用GST亲和层析法进行纯化。使用序列特异性蛋白酶去除gst标记(见材料与方法),并将无标记PmlR2重组蛋白用于凝胶转移试验等实验。PmlR2结合到的启动子区PME6650而且folM但并没有束缚到rpoD低蛋白浓度的启动子区(图;S2).在这些启动子区域中,P6650被用作探针。因此,我们使用P6650来研究PmlR2的dna结合活性。
利用DNase I足迹分析鉴定P基因正义链和反义链上的pmlr2结合位点6650(无花果。2一个和B).在正义链(+ 32 ~ - 45)和反义链(+ 37 ~ - 54)的单个宽区域受到了DNase I处理的保护,表明P6650含有多个pmlr2结合位点(图;2B).支撑数据如图所示。S3用于分析图中感觉链的结果。2B,与反义链相比,PmlR2与义链的特异性dna结合活性较低。我们将受保护区域的核苷酸序列与一致的PplR1进行了比较9.如图所示。2一个和C5 ' -TGTACA-N核苷酸序列12-TGTACA-3',指定为Site 1和5 ' -TGTACA-N12-TGTACA-3'与共识序列(5 ' -T(G/A)TACA-N)具有很高的相似性,被指定为Site 212-TGTA(C/T)A-3')的PplR19.Site 1与P . 35区域重叠6650的转录起始位点和翻译起始密码子之间PME6650.
PmSB-LOV重组蛋白吸光度光谱的光依赖性变化
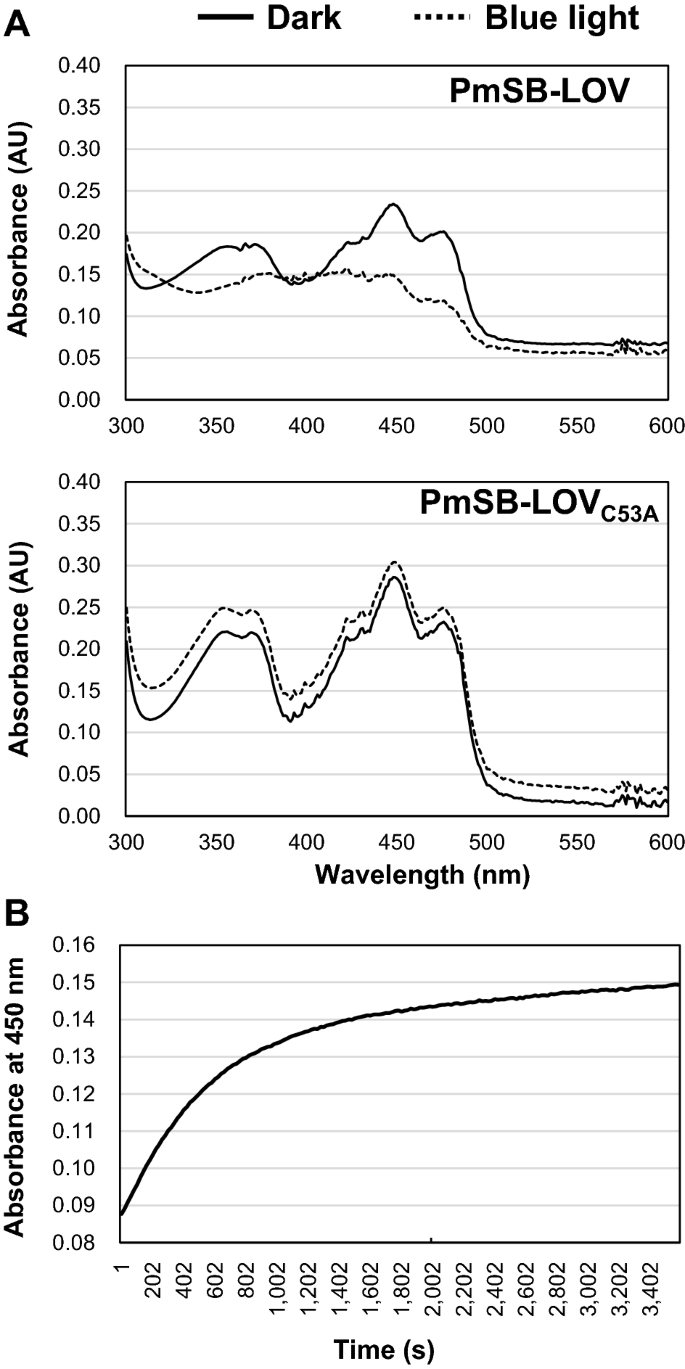
LOV蛋白通常在紫外可见区(300-600 nm)表现出光依赖性的吸收变化(称为光循环),这是由于在光照下,LOV的保守Cys残基与FMN之间形成了巯基加合物。在没有光的情况下,LOV畴吸收能力恢复到暗态(暗态恢复)。为了研究吸收的变化,我们纯化了野生型PmSB-LOV和PmSB-LOVC53A重组蛋白来自大肠杆菌表达式宿主(见材料与方法).两种纯化蛋白都是黄色的,这与它们与黄素衍生物的关系一致,这是LOV蛋白的特征。HPLC分析表明,两种类型的PmSB-LOV蛋白主要与FMN和少量FAD发色团相关(图。S4).这一结果说明了两点:(i) FMN是PmSB-LOV的主要发色团;(ii) Cys53残渣与黄素无关。
然后,我们在25°C的黑暗和光照条件下测量了PmSB-LOV的吸光度光谱(300-600 nm)。暗孵育PmSB-LOV的吸收光谱包含多个峰,类似于典型的LOV谱。PmSB-LOV光谱在100 μmol s 450 nm蓝光照射下发生变化−1米−210 s(图;3.A),而暴露在365、530或660 nm的光下没有影响。可见,PmSB-LOV蛋白具有典型的吸光度谱行为。
我们还分析了PmSB-LOV的光谱C53A来评估保守Cys的参与情况53残留在吸光度的变化。与野生型蛋白PmSB-LOV不同C53A在黑暗和光照条件下都表现出相似的吸收光谱(图。3.A),认为Cys53残基是与FMN形成硫醇加合物所必需的,类似于其他LOV蛋白。
接下来,在450 nm处监测吸光度1小时,以追踪暗态恢复。56分钟后观察到完全恢复到黑暗状态。3.B).这说明PmSB-LOV是一种相对慢回复性的LOV蛋白。
异源PmlR2与PmSB-LOV的蛋白-蛋白相互作用大肠杆菌
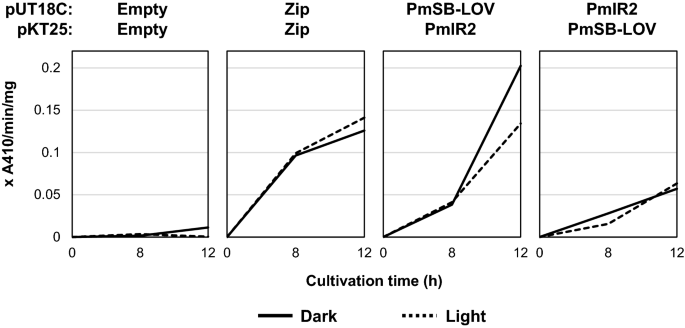
为了获得PmlR2和PmSB-LOV在体内相互作用的初步信息,我们使用PmlR2和PmSB-LOV进行了双杂交分析大肠杆菌作为异源宿主。在每个结构体中,PmlR2和PmSB-LOV在每个分裂cAMP合酶结构域(T18和T25)的c端结构域融合。如图所示。4,大肠杆菌转化株携带pmlR2而且ppSB-LOVβ-半乳糖苷酶活性高于阴性对照(空/空)和阳性对照(zip/zip)。在暗光条件下观察相互作用产生的β-半乳糖苷酶活性。我们对大肠杆菌转化株携带pmlR2而且ppSB-LOV,其中两个基因都与不同对cAMP合酶结构域融合(图。S5).我们假设通过c端或n端与cAMP合酶结构域融合的嵌合结构可以防止光依赖性蛋白-蛋白相互作用。
由PmlR2和PmSB-LOV组成的光依赖复合物
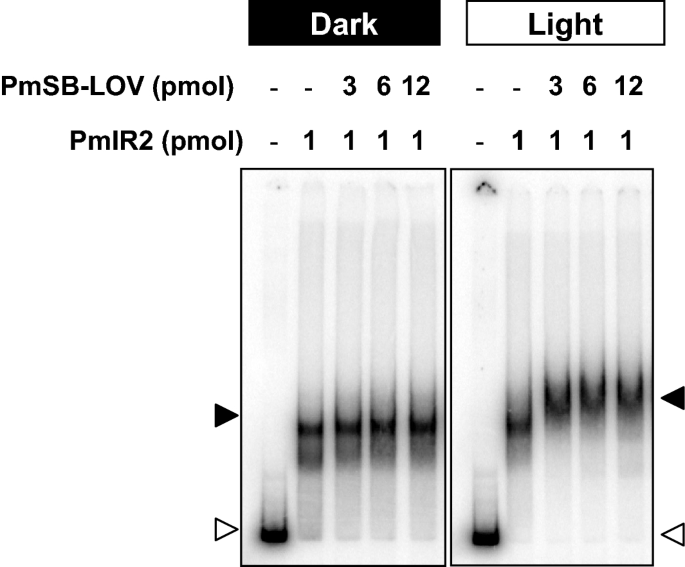
我们在PmSB-LOV存在的情况下进行了凝胶转移试验,以检查PmlR2和PmSB-LOV之间的关联。当P6650使用非发光PmSB-LOV作为探针时,加入非发光PmSB-LOV会引起局部超移(图;5).另一方面,白光处理的PmSB-LOV的加入引起了明显的超位移,并且观察到部分剂量依赖的超位移(图2)。5).这有力地表明,发光的PmSB-LOV与PmlR2直接相互作用,而非发光的PmSB-LOV具有较低的活性。
PmlR2和PmSB-LOV与P6650.在添加总共0.5-5.0 ng的蛋白质之前,在黑暗(左图)或白光(右图)条件下孵育指定数量的蛋白质32P-labeled调查。由PmlR2、PmSB-LOV和探针组成的超位移需要在电泳过程中持续照射聚丙烯酰胺凝胶。开三角形和闭三角形分别表示探针和蛋白质- dna复合物。进行了三个独立的技术重复实验,并给出了具有代表性的数据。全长凝胶的原始和未加工版本包括在图中。S12.
PmlR2和PmSB-LOV之间的光依赖性蛋白-蛋白相互作用
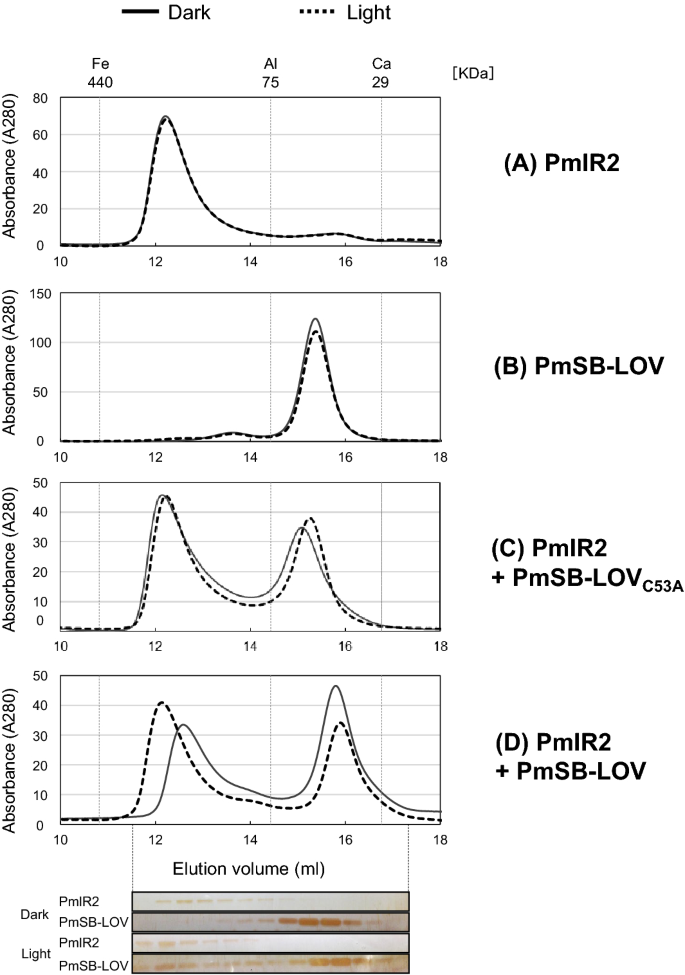
为了证实凝胶转移实验中观察到的PmSB-LOV和PmlR2之间的光诱导相互作用,进行了凝胶过滤色谱分析。用凝胶过滤层析法分离在黑暗或光照条件下孵育的单个蛋白质或蛋白质混合物。当每个蛋白质分别被装载到色谱柱上时,PmlR2和PmSB-LOV以大约226 kDa和51.8 kDa洗脱,而不管它们的照明状态如何(图2)。6A和B),表明PmlR2和PmSB-LOV在溶液中分别以六聚体和三聚体形式存在。
凝胶过滤色谱分析PmlR2和PmSB-LOV之间的蛋白-蛋白相互作用。重组PmlR2 (一个), PmSB-LOV (B), PmSB-LOVC53A和PmlR2 (C),以及PmSB-LOV和PmlR2 (D)在暗(实线)和亮(虚线)条件下孵育,应用于ÄKTA FPLC系统上的Superdex 200 HR 10/30列。PmlR2(200µL;0.43 mg/mL), PmSB-LOV(200µL;0.47 mg/mL), PmSB-LOVC53A(200µL;0.21 mg/mL)重组蛋白。柱用1 × PBS平衡显影,流速为0.25 mL/min。采用分子尺寸标准(Fe:铁蛋白,Al: Conalbumin, Ca:碳酸酐酶,分别为440,75和29 kDa)。白光应用于大约50 μ mol s−1米−2D面板的SDS-PAGE和银染色如图所示。在技术重复中进行了两次独立实验,并给出了具有代表性的数据。全长染色凝胶的原始和未加工版本包括在图中。向.
当PmlR2和PmSB-LOV在黑暗中共同孵育时,它们被洗脱为两个主要的峰(图2)。6D).每个洗脱体积与PmlR2和PmSB-LOV几乎相同。SDS-PAGE和银染色证实,前、后两个峰分别含有PmlR2和PmSB-LOV。因此,在黑暗中孵育不影响PmSB-LOV与PmlR2的相互作用。相比之下,PmlR2和PmSB-LOV的光照混合物产生了一个新的峰。6D)以更高的音量洗脱。新峰包含PmlR2和PmSB-LOV,而后两个峰分别与PmlR2和PmSB-LOV以几乎相同的分数洗脱。因此,PmlR2和PmSB-LOV在光照下相互作用。我们检查了PmSB-LOVC53A用同样的方法。如图所示。6C, PmSB-LOVC53A在黑暗或光照条件下,与PmlR2没有关联。我们的结果表明,PmSB-LOV直接与PmlR2结合,以响应照明,Cys53可能与这种依赖光的关联有关。
PmlR2 n端结构域与PmSB-LOV相互作用
为了确定PmlR2和PmSB-LOV之间的相互作用域,对两个截断的蛋白(i) PmlR2- n蛋白只包含HTH dna结合域和(ii) PmlR2- c蛋白只包含c端结构域)进行凝胶过滤色谱分析。计算得到的PmlR2-N和PmlR2-C重组蛋白分子量分别为12.8 kDa和25.9 kDa。当每个蛋白质分别加载到色谱柱上时,PmlR2-N和PmlR2-C分别以约22 kDa和> 440 kDa洗脱,而不考虑光照状态(图2)。S6而且S7),表明PmlR2-N和PmlR2-C可能分别以二聚体和低聚体的形式存在。当PmlR2-N和PmSB-LOV在黑暗中共孵育时,它们被洗脱为两个主峰。每个洗脱体积与PmlR2-N和PmSB-LOV几乎相同(图2)。S6).相比之下,PmlR2-N和PmSB-LOV的光照混合物产生了一个新的峰(图。S6),以更高的体积洗脱,并含有PmlR2和PmSB-LOV。因此,PmlR2-N与PmSB-LOV在光照下相互作用。我们用同样的方法研究了PmlR2-C和PmSB-LOV之间的相互作用。如图所示。S7在黑暗或光照条件下,PmlR2-C与PmSB-LOV不相关。我们的结果表明,PmlR2-N结构域直接与PmSB-LOV相互作用,以响应照明,也表明PmlR2-N结构域阻止其C端结构域的寡聚。
PmSB-LOV对PmlR2 dna结合活性的光依赖性抑制
我们之前的基因研究p . putidaKT2440表明PplR具有负调控作用,其活性受PpSB-LOV控制。因此,我们通过表面等离子体共振光谱(BIAcore)检测PmSB-LOV存在时PmlR2 dna结合活性是否被抑制,从而研究了PmSB-LOV的功能。
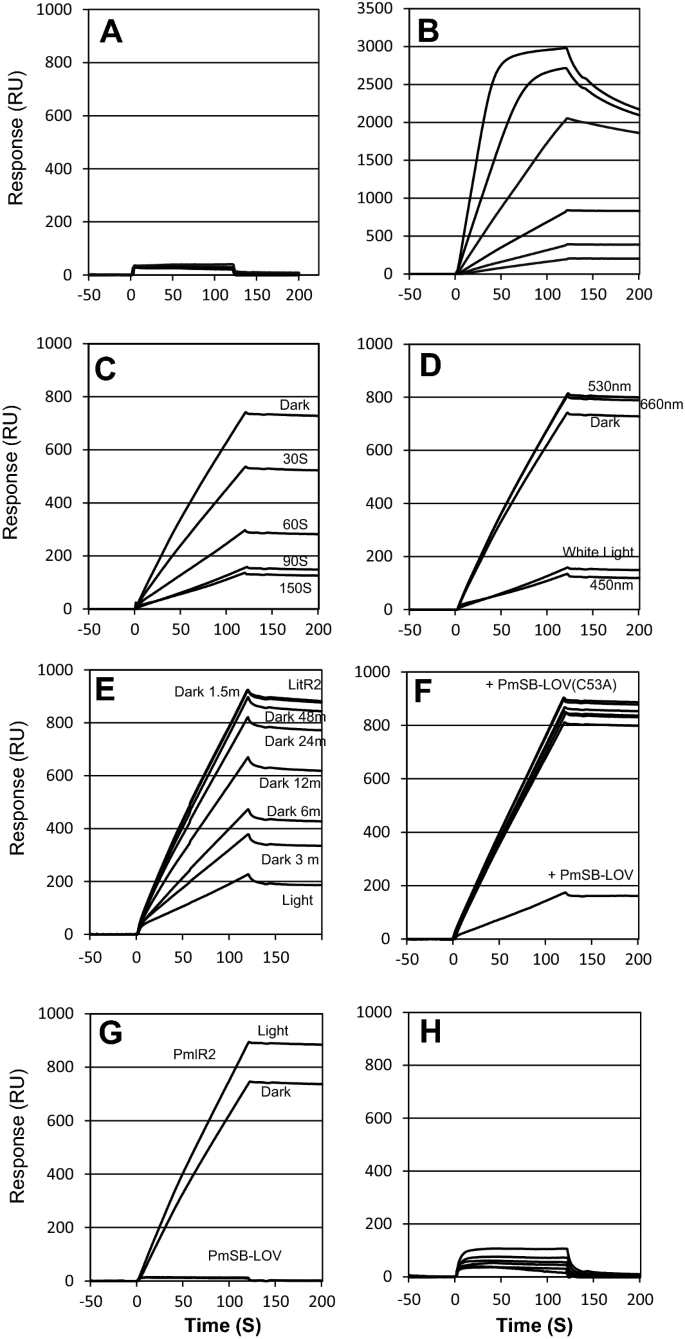
前面的启动子区域PP_0468用作阴性对照,因为PmlR2在凝胶位移试验中不与该区域结合(数据未显示)。一种生物素偶联的寡核苷酸,含有P的pmlr2结合位点26650(−9 ~ + 41,图;2A)固定在传感器芯片SA上。未观察到pmlr2结合反应468固定在传感器芯片上(图;7A)。相反,在固定芯片的Site 2上注入越来越多的PmlR2后,观察到反应单位的剂量依赖性增加(图2)。7B),表示PmlR2与Site 2结合。
BIAcore分析。固定在传感器芯片表面的DNA包括P468作为阴性对照(一个), pmlr2结合位点2 (B) - (G)和突变的pmlr2结合位点2 (H).注射蛋白8.6 ~ 275.2 nM PmlR2 (一个,B而且H)、34.4 nM PmlR2及292.3 nM PmSB-LOV (C- - - - - -E,G)、34.4 nM PmlR2和292.3 nM PmSB-LOVC53A(F).样品用蓝光照射(λ马克斯= 450 nm),绿灯(λ马克斯= 530 nm),红光(λ马克斯= 660 nm)在100 μ mol m−2年代−1为指定的时间。在技术重复中进行了两次独立实验,并给出了具有代表性的数据。
我们检测了PmSB-LOV对PmlR2 dna结合活性的影响(图2)。7C). PmlR2 dna结合活性在发光的PmSB-LOV存在下被抑制。这种抑制作用是由蓝光照射90 s引起的;相比之下,黑暗孵育或365纳米、绿色或红色光的照明没有影响(图。7D).我们还研究了PmSB-LOV在蓝光照射后的暗态恢复。观察到反应单位大幅增加,代表pmlr2在长时间暗潜伏期结合(图。7E).因此,由于PmSB-LOV暗态恢复,暗孵育导致抑制功能丧失。
我们还研究了PmSB-LOV的功能C53A(无花果。7F),不抑制PmlR2的dna结合活性。我们证实光照不影响PmlR2的dna结合亲和力,PmSB-LOV没有dna结合活性(图2)。7我们还证实了PmlR2与Site 2的特异性结合(TGTACA-N)12-TGTACA)。PmlR2没有结合含有突变位点2的dsDNA (AAGCTT-N12-AAGCTT),其中一致性TGTACA序列均被AAGCTT所取代(图2)。7H).我们的数据表明PmlR2的dna结合活性被蓝光激活的PmSB-LOV和Cys抑制53是其抑制功能所必需的。此外,由于暗态恢复,PmSB-LOV的暗孵育消除了抑制功能。
PmSB-LOV可作为PmlR2的抗抑制剂
我们认为PmSB-LOV能感知蓝光,并与PmlR2结合抑制其dna结合活性。为了阐明PmSB-LOV是否在体外直接抑制PmlR2功能,我们使用mRNA合成和调控所需的成分进行了体外转录径流试验。我们使用的RNA聚合酶全酶包含一个基本的sigma因子σ70一种商业上可用的蛋白质大肠杆菌,因为P的-10和- 35序列6650是类似的大肠杆菌σ70端依赖启动子。假单胞菌σ70是不溶的,因此不能用于本研究。
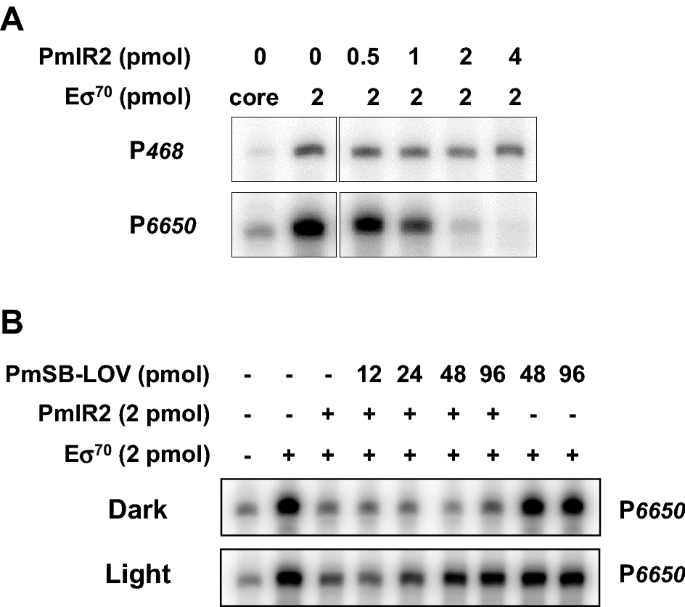
RNA聚合酶全酶从含有P的模板中生成预期长度的转录本6650或P468发起人(无花果。8一个,S8的核心酶大肠杆菌RNA聚合酶产生少量的转录本。为了检验PmlR2是否作为阻遏因子,我们将PmlR2加入到转录反应中。PmlR2剂量依赖性地抑制P6650.当P468被用作模板;可见,PmlR2特异性地抑制了P6650.
然后,我们评估了PmSB-LOV是否对PmlR2反应光的活性具有负调控作用。在PmlR2存在的情况下,将光照或未光照的PmSB-LOV加入转录反应中。光照PmSB-LOV的加入以剂量依赖的方式恢复转录,尽管存在PmlR2(图2)。8B,S8).然而,未照亮的PmSB-LOV的存在并不影响转录效率。在PmSB-LOV存在时未观察到抑制作用。我们的研究结果表明,PmlR2是一种抑制因子,而PmSB-LOV是一种光依赖性的PmlR2活性抗抑制因子。
讨论
细菌“短”LOV蛋白的生化特性已经得到了很好的研究,但它们的效应蛋白还没有被确定。我们的研究发现,MerR家族转录调节因子PmlR2是PmSB-LOV的一个效应蛋白p . mendocinaNBRC14162。这是关于鉴定“短”细菌LOV蛋白的蛋白质伴侣的第一份报告。此外,在细菌光响应系统中,MerR家族调控因子与LOV相互作用的分子机制尚不清楚。因此,本研究确定的调控系统是一种新型的光调控机制。PmlR2和PmSB-LOV同源物的守恒假单胞菌sp .和密切相关的细菌表明光感和反应是在自然界生存所必需的。
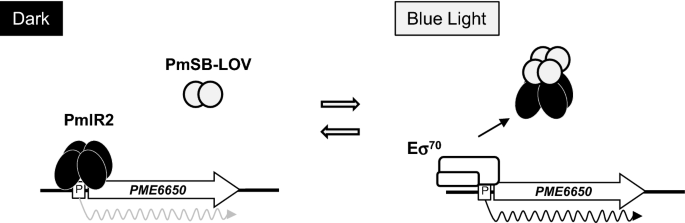
数字9阐述了基于本研究结果的调控模型。在黑暗条件下,PmlR2与蛋白启动子区域的结合位点结合PME6650,阻止RNA聚合酶的进入。在蓝光照射下,fmn结合的PmSB-LOV转化为其激活形式,直接与PmlR2相互作用。这种蛋白-蛋白相互作用消除了PmlR2的dna结合活性,从而允许由RNA聚合酶从光诱导启动子转录。当环境恢复到黑暗状态时,PmSB-LOV因暗态恢复而失活,游离的PmlR2与启动子区域结合,诱导OFF状态。
体外转录流实验清楚地表明,PmlR2的基础功能是一个阻遏因子,它是防止光诱导基因在不需要时表达的基础,类似于许多LitR/CarH家族成员的功能。PmlR2结合到与PplR1相似的一致序列(图2)。2C), PmlR2的n端HTH dna结合域与PplR1高度相似(78%)。在翻译起始位点(5 ' -TGTACA-N)上游95 bp处也发现了共识12-GGTATA-3')为光诱导基因,folM.因此,PmlR2可能与这些序列结合以控制它们的转录。chip-seq等综合分析将允许在未来的研究中对PmlR2结合位点进行更彻底的评估。
PmlR2含有一个内部的B12未知函数的结合域和c端结构域(图;S1A,许多以前的研究表明,B12的依赖光传感器LitRt .酸奶有两个c端B12-绑定域(B12-binding_2和B12-绑定域)调节adb12绑定1,27.后一个域包含一个与B关联所必需的母题12.PmlR2的内部结构域(残基Trp113将低浓缩铀161)与B相似12用an绑定_2域E-value为1.1e-05,而PmlR2的c端结构域与B12的-绑定域栖热菌属LitR。与B相似度低12-binding域表明PmlR2不作为AdoB12绑定域名。与此相一致的是,我们之前对p . putidaKT2440显示B .12PmlR2同源物PplR蛋白的光诱导转录不需要9.基于凝胶过滤分析的结果,我们假设c端是一个寡聚结构域(图2)。S7).
PmSB-LOV主要与FMN相关(图。S4),并表现出典型的LOV畴型特性,如光诱导光谱变化和暗态恢复(图。3.A). PmSB-LOV的加合态寿命与PpSB1-LOV相似p . putidaKT2440 (PpSB1-LOV为109±18 min, PpSB2-LOV为0.5±0.1 min)。在BIAcore分析中也观察到PmSB-LOV的暗态恢复(图2)。7E)。
被充分研究的“短”LOV蛋白的构象在很大程度上受光照的影响:RsLOV的Rhodobacter sphaeroides显示光诱导亚基解离28.相反,PmSB-LOV和PpSB1-LOV16,17,18在溶液中组成二聚体,无论光照条件如何(图。6).ppss1 - lov的轻态晶体结构表明,位于亚基界面的n端n -帽和c端j α-螺旋通过线圈相互作用稳定了平行同型二聚体。n -帽和j α-螺旋在“短”LOV蛋白中是严格保守的,包括PmSB-LOV(图2)。S1B),表明光诱导的构象变化,而不是亚基解离或结合,是PmSB-LOV活性切换所必需的。蛋白质在黑暗和光明下的三维结构将揭示响应照明的功能变化的分子机制。
保守的Cys53残基对于PmSB-LOV的光诱导活化是必不可少的,因为它是光诱导吸收光谱变化所必需的(图2)。3.A),与PmlR2的相互作用(图;6)和PmlR2抑制活性(图2。7,8).相比之下,Cys53残留与FMN没有关联(图。3.一个和S4)或二聚体(图;6).这些结果表明,Cys之间形成了共价键53黄素发色团的4a位置是光诱导PmSB-LOV功能改变的基础,在光激发下产生PmSB-LOV的活性形式。因此,Cys53残基是PmSB-LOV光传感的关键。
我们的分析显示PmlR2的n端HTH结构域与PmSB-LOV相互作用(图2)。S6).海洋细菌EL222蛋白LOV结构域n端短延伸和j α-螺旋Erythrobacter litoralis与c端HTH结构域相互作用29.这表明PmSB-LOV的j α-螺旋结构(图;S1B)与PmlR2的HTH结构域相互作用(图。S1A).需要进一步的分析来描述相互作用背后的分子机制。
我们的结果提供了“短”LOV蛋白和伴侣蛋白之间相互作用的第一个证据,因为凝胶转移试验(图2)。5)和凝胶过滤色谱(图;6)证明了PmlR2和PmSB-LOV之间的光诱导蛋白-蛋白相互作用。此外,我们报道了LOV作为一种抗抑制因子,因为BIAcore分析表明,被照亮的PmSB-LOV抑制PmlR2的dna结合活性(图2)。7),体外转录径流试验表明,蓝光激活的PmSB-LOV可作为PmlR2的抗抑制因子(图2)。8).我们还通过BIAcore分析观察了暗恢复,发现在60 min后,暗恢复所需的时间与吸收光谱相似。我们的研究表明,PmSB-LOV以光诱导的方式直接与PmlR2结合;然而,PmSB-LOV的分子内机制和结构尚不清楚。阐明暗态和光态的三维结构可以阐明精确的光响应机制。
凝胶位移试验和BIAcore分析显示,在光照条件下PmlR2、PmSB-LOV和探针DNA的复合物形成有不同的结果。凝胶转移实验表明形成稳定的PmlR2/PmSB-LOV复合物与P结合6655(图右面板。5),而BIAcore分析显示PmSB-LOV抑制PmlR2 dna结合活性(图。7C).在这两个实验中,PmlR2/PmSB-LOV配合物都是在光照条件下预先形成的,随后分析了与探针的相互作用。在凝胶转移实验中,用外部光源持续照射凝胶进行大约120分钟的电泳。相比之下,在BIAcore分析中,蛋白质复合物与探针接触的时间为1-2分钟。因此,两次实验结果的不一致可能是由于实验条件不同。我们倾向于BIAcore分析的结果,因为它需要更少的分析时间。
本研究也为MerR家族调控者的作用提供了见解。两种参与细菌光反应系统的MerR家族蛋白已被鉴定:(i)光敏感AdoB12基于LitR/CarH族和(ii)大肠杆菌由蓝光受体BluF控制30..我们的研究揭示了一种新的类型,(iii) PmlR2由一个“短”的LOV蛋白调控。LitR/CarH家族同源物的广泛分布使我们假设细菌中存在一种未知的光反应机制的多样性。
材料与方法
细菌菌株、质粒和培养基
假单胞菌mendocinaNBRC 1416222来源于日本和usa NITE (NBRC)生物资源中心。大肠杆菌(大肠杆菌)菌株HST08和Rosetta2(DE3)pLysS (Takara Bio Inc., Shiga, Japan)分别作为宿主进行DNA操作和蛋白表达。pUC118 (Takara Bio)用于一般DNA操作。pMD19 (Takara Bio)用于pcr生成的DNA片段的ta克隆。pGEX-6P-2 (GE Healthcare UK Ltd, Buckinghamshire, England)用于过表达PmlR2和PmSB-LOV大肠杆菌。DNA操作用酶购自Takara Bio。培养条件和遗传操作大肠杆菌由Maniatis等人描述。31大肠杆菌而且p . mendocina在Luria-Bertani (LB)培养基中生长于28°C或30°C31;固体培养基中加入1.5%琼脂(Kokusan, Tokyo, Japan)。最终浓度为:氨苄西林、卡那霉素50 μg/mL;氯霉素15 μg/mL。
光照射实验
照明培养箱(BR-180LF;Taitech,埼玉,日本)配备白色,蓝色和红色荧光灯(20w;东芝,东京,日本)用于摇晃液体培养。纯化后的重组蛋白用LED灯照射,蓝光(λ马克斯= 450 nm),绿灯(λ马克斯= 530 nm),红光(λ马克斯= 660 nm) (Optocode,东京,日本)。亮度用LI-250型光度计(LI-COR公司,Lincoln, NE, USA)测量。
总RNA制备及定量RT-PCR
从中纯化总RNAp . mendocina根据制造商的说明,使用RNeasy保护细菌试剂盒(Qiagen GmbH, Hilden, Germany)。p . mendocina在28°C的LB液体中,在蓝光下(30.5 μ mol s−1米−2)和黑暗的环境。按照制造商的描述,使用SuperScript VILO cDNA Synthesis Kit (Thermo Fisher Scientific)进行cDNA合成。根据制造商说明书和我们之前的研究,用PowerUp SYBR Green Master Mix(赛默飞世尔科学公司)和Applied Biosystems 7500 Real-Time PCR系统(赛默飞世尔科学公司)对合成cDNA的数量进行量化8.寡核苷酸引物总结见表S2.相对基因表达量的定量使用信号dnaA(一种重要的染色体复制启动基因)作为内参和相对定量2−ΔΔCt方法32.所有反应均重复进行三次。
RNA-seq分析
为了测序文库的制备,用以下方法制备1µg总RNA。根据制造商的协议,使用RiboZero细菌试剂盒(Illumina, San Diego, CA, USA)去除核糖体RNA。测序文库由Illumina公司(New England Biolabs, Ipswich, MA, USA)的NEBNext mRNA文库准备试剂盒制备,并进行如下修改。使用随机六聚体引物进行逆转录。在第二链合成后,使用Covaris S2超声系统(Covaris, Woburn, CA, USA)将双链cDNA碎片化到平均长度为300 bp。根据制造商的规格(Illumina),使用HiSeq2500系统进行了100次配对端测序循环。测序反应完成后,使用Illumina分析管道(CASAVA 1.8.0)对原始测序数据进行处理。RNA-Seq reads使用CLC Genomics Workbench ver进行修剪。9.0.1 (Qiagen, Venlo,荷兰),具有以下参数;质量评分:0.05; Removing terminal 15 nucleotides from 5’ end and 2 nucleotides from 3’ end; Trimming read through adaptor: yes; Removing truncated reads less than 30 nucleotides in length. Trimmed reads were mapped to all genes inp . mendocinaNBRC 14162(登录号:nz_bbqc000000000 .1)使用CLC Genomics Workbench ver。9.0.1.使用以下参数;长度分数:0.7;相似度:0.9;读的最大命中数:1。通过计算每个基因的映射reads来计算每个基因的表达水平,并通过计算RPKM值进行归一化。原始序列读取被存入DRA/SRA数据库,登录号为DRA014932。RNA-seq实验中的生物复制为n= 1,因为在本研究中RNA-seq的目的是为了初步筛选光诱导基因。
转录起始位点的测定
转录起始位点通过5 ' -RACE (cDNA末端快速扩增)和CAGE实验确定,使用光照射细胞中纯化的总RNA。对于5 ' -RACE,序列的5 ' -末端的排序PME6650而且PP0468根据制造商的说明,使用5 ' -Full RACE Core Set (Takara Bio)进行转录。所使用的寡核苷酸,ORF4-RT, ORF4-A1, ORF4-A2, ORF4-S1和ORF4-S2用于PME6650、PP468-RT、PP468-A1、PP468-A2、PP468-S1、PP468-S2PP0468表所示S3.扩增的DNA片段被克隆到t载体pMD19中,克隆的插入物由Eurofins Genomics测序。对于CAGE实验,使用DNAFORM (Yokohama, Kanagawa, Japan)进行CAGE文库制备、测序、作图、基因表达和motif发现分析。简而言之,使用生物分析仪(Agilent)评估RNA质量,以确保RNA完整性数(RIN)超过7.0,A260/280和260/230比值超过1.7。第一链cdna转录到有帽rna的5 '端,连接到CAGE“条形码”标签上,并将测序的CAGE标签映射到p . mendocina在丢弃核糖体rna后,使用BWA软件(v0.5.9)基因组。对于标签聚类,CAGE标签计数数据使用CAGEr进行聚类33以及Paraclu算法34使用默认参数。每百万计数(CPM) < 0.2的集群被丢弃。
重组PmlR2、PmSB-LOV和PmSB-LOV的过表达和纯化C53A
为构建PmlR2、PmlR2- n (1-113 aa)、PmlR2-C (95 aa至C端)、PmSB-LOV的蛋白表达载体,分别采用引物R2ex-F/R2ex-R (PmlR2)、R2ex-F/R2ex-RN (PmlR2- n)、R2ex-FC /R2ex-R (PmlR2-C)、lox - f / lox - r (PmSB-LOV) PCR扩增各基因(表2)S3).PmSB-LOV的表达向量C53A,其中半胱氨酸53 (Cys .53)被丙氨酸取代,用两段PCR生成。分别使用引物LOVex-F/C53A-MR和C53A-MF/ lovx - r扩增突变的上游和下游区域。第一轮PCR扩增子作为引物LOVex-F/LOVex-R的第二轮PCR模板。上游第一轮PCR产物的3'端与下游区域互补,从而在第二轮PCR中产生融合结构。扩增的DNA片段被消化Bam你好,生态然后克隆到pGEX-6P-2 (GE Healthcare)的同一位点生成pGEX-PmlR2 (PmlR2)、pGEX-PmlR2- n (PmlR2- n)、pGEX-PmlR2- c (PmlR2- c)、pGEX-LOV (PmSB-LOV)、pGEX-C53A (PmSB-LOV)C53A).使用ABI3100遗传分析仪测序验证了插入。重组基因在异丙基β- d -硫半乳糖苷(IPTG)诱导启动子的控制下编码n端GST蛋白。大肠杆菌携带表达载体的Rosetta2(DE3)pLysS细胞在28℃LB液体培养基中培养,当培养达到0.3 OD时加入IPTG600.诱导3 h后,离心收集细胞,悬浮于1 × PBS缓冲液(含140 mM NaCl, 2.7 mM KCl, 10 mM Na)中2阿宝4KH为1.8 mM2阿宝4).用超声波仪(Astrason XL2020;Misonix, NY, USA)。通过离心去除细胞碎片,将上清转移到ÄKTA explorer 10S (GE Healthcare)上的5ml GSTrap HP色谱柱(GE Healthcare)中。n端GST标记用PreScission蛋白酶(GE Healthcare)在4°C下在GST亲和柱上去除16小时。蛋白洗脱,用1 × PBS透析。被洗脱的重组蛋白在翻译起始密码子之前有一个额外的n端GPLGS序列。用考马斯色或银染色SDS-PAGE验证均匀性后,将纯化的蛋白质分成小体积,在4°C或- 20°C保存,直到进一步使用。吸收光谱由U-2800A(日立高科技科学公司,东京,日本)、Nano Drop 2000 (Thermo Fisher科学公司)或Cary 8454 UV - vis二极管阵列系统(安捷伦技术公司,CA,美国)的紫外光谱法记录。按照Bio-Rad蛋白质测定试剂盒(Bio-Rad Laboratories, CA, USA)说明书所述,以BSA (Thermo Fisher Scientific)为标准,采用Bradford方法测定蛋白质浓度。
发色团的HPLC分析
如前所述,对PmSB-LOV的黄素衍生物发色团进行了分析35.PmSB-LOV的发色团(4.7µg)和PmSB-LOVC53A(12µg)在95°C下通过热变性释放10分钟。然后样品在14000 rpm下离心20分钟。过滤后(0.22µm过滤器),将10µL样品注入配备CAPCELL PAK C18色谱柱的高效液相色谱中(SISEIDO, Tokyo, Japan)。以含70%乙腈的乙酸铵(50 mM, pH 6.0)为溶剂。使用市售黄素单核苷酸(FMN)和黄素腺嘌呤二核苷酸(FAD) (Wako, Tokyo, Japan)作为参比化合物。
光谱分析
用Cary 8454紫外-可见二极管阵列系统记录PmSB-LOV蛋白吸收光谱。用蓝色发光LED灯以大约100 μ mol s照射样品后,记录被照射的PmSB-LOV蛋白的吸光度光谱(300-600 nm)−1米−2持续10秒。以1 × PBS缓冲液为参考,在480 nm处每隔20秒测量1 h的暗态恢复。所有测量均在25°C下10毫米光径石英试管中进行。
Gel-shift化验
凝胶转移试验如前所述进行9.P的探针6650PrpoD和PfolM分别用引物对ORF4-FPF/ORF4-FPR、rpoDF/rpoDR和ORF12F/ORF12R进行PCR扩增(表S3).DNA片段用T4多核苷酸激酶和[γ-]磷酸化32P] ATP。所述反应混合物共含有0.5-5.0 ng的32p标记探针(10,000-20,000 cpm), 0-160 pmol重组PmlR2, 0-12 pmol PmSB-LOV在50µL结合缓冲液中[包含10 mM Tris-HCl (pH 7.0), 50 mM KCl, 1 mM EDTA, 1 mM二硫苏糖醇,10% (vol/vol)甘油,1µg poly(dI-dC)和50µg mL−1在37℃下孵育30分钟。Poly(dI-dC)作为非特异性dna结合蛋白的竞争对手。为了进行超位移分析,PmlR2和PmSB-LOV在光照条件下(42µmol m)孵育−2年代−1) 10分钟,然后32在反应混合物中加入p标记探针和poly(dI-dC)。混合物进一步在光照下孵育30分钟。在电泳期间,原生PAGE持续用42 μ mol m照射−2年代−1室温下的白光。通过暴露于富士成像板(Fuji Film, Tokyo, Japan)来检测干燥凝胶中的放射性,并使用Typhoon 9410或Typhoon FLA 9500图像分析仪(GE Healthcare)扫描图像。
DNase I足迹分析
为了确定PmlR2 dna结合位点,DNase I足迹分析如前所述进行9.为了标记DNA的正义或反义链,一个引物被T4多核苷酸激酶和[γ-]磷酸化32P] ATP。32采用ORF4-FPF*/ORF4-FPR引物PCR扩增p -标记dna片段(表4S3)为义链,ORF4-FPF/ORF4-FPR*为反义链。星号表示[γ-]标记的引物32P] ATP。反应混合物(50 μL)由10 kcpm组成32p标记DNA探针;10-80 pmol PmlR2;25 mM HEPES-KOH, pH 7.9;0.5 mM EDTA-NaOH, pH 8.0;50mm KCl;还有10%的甘油。为了使PmlR2与探针结合,在25℃下孵育30 min。最终浓度为20 μg/ml的DNase I加入,进一步孵育1 min。为了终止反应,100 μL停止液(含100 mM Tris-HCl, pH 8.0;100 mM NaCl;1%的钠N月桂酰sarcosinate;10mm EDTA-NaOH, pH 8.0;添加25 mg/ml三文鱼精子DNA)和300 μL苯酚:氯仿(1:1)。将消化后的探针用乙醇沉淀法浓缩,然后用80%乙醇洗涤,6 μL甲酰胺-染料混合物溶解,在6%聚丙烯酰胺凝胶上运行。如上所述进行自放射显像。为了供参考,我们制备了Maxam-Gilbert测序梯子(G + A和T + C反应)32p标记探针DNA36.
细菌腺苷酸环化酶双杂交(BACTH)体系分析
用BACTH系统试剂盒(EUROMEDEX, Souffelweyersheim,法国)研究了PmlR2和PmSB-LOV之间的蛋白-蛋白相互作用。37根据制造商的说明。用引物R2T-F/R2T-R pcr扩增PmlR2和PmSB-LOV基因S3)pmlR2和LOVT-F/LOVT-R(表S3)pmSB-LOV.扩增子被消化Xba我和Kpn然后克隆到pUT18C和pKT25的同一位点。PmlR2和PmSB-LOV在分裂的腺苷酸环化酶结构域(T18和T25)的c端融合。该核苷酸序列由Eurofins基因公司确认。pUT18C和pKT25衍生物共转化为a自保”缺乏症菌株大肠杆菌BTH101,由制造商提供。β-半乳糖苷酶测定如前所述38.
凝胶过滤色谱法
为了估计重组蛋白的天然分子量,进行了凝胶过滤柱层析。PmlR2 (0.43 mg/mL)、PmSB-LOV (0.48 mg/mL)和PmSB-LOV样品C53A(0.21 mg/mL)按200-µL体积应用于ÄKTA FPLC系统(GE Healthcare)上的Superdex 200 HR 10/30色谱柱,流速为0.3 mL/min,根据制造商说明。为了进行相互作用分析,PmlR2和PmSB-LOV在光照条件下(约50 μ mol s)共孵育−1米−2)静置5分钟后再涂于色谱柱上。以含140 mM NaCl、2.7 mM KCl、10 mM Na2PO4和1.8 mM KH2PO4 (pH 7.2)的冷1 × PBS(流速为0.3 mL/min)平衡显影。凝胶过滤校准试剂盒(GE Healthcare)用于尺寸标准(铁蛋白、醛缩酶、联白蛋白、卵清蛋白、RNase A和蓝葡聚糖,分别对应于440、158、75、43、13.7和2 kDa)。
BIAcore分析
为了检测PmlR2和PmSB-LOV蛋白的功能,在BIAcore 1000 (GE Healthcare)上进行了表面等离子体共振分析。含有野生型pmlr2结合位点2、突变pmlr2结合位点2和P468(表S4)被固定在传感器芯片SA (GE Healthcare)的链霉亲和素表面。制备了hplc级5 ' -生物素偶联意义和无标记反义寡核苷酸(均为50 nt)。以HBS-EP (GE Healthcare)为运行缓冲液,温度为28℃,10 μL/min。注入PmlR2 (8.6-275.2 nM)和PmSB-LOV (292.3 nM)样品(20µL)并记录传感器图。每个蛋白质稀释剂的等效体积应用于非固定表面,以估计背景体折射率。用80µL含2 M NaCl的HBS-EP以100µL/min的速度再生传感器芯片SA表面。PmSB-LOV和PmSB-LOVC53A暴露于100 μ mol m−2年代−1白光、365 nm、450 nm、530 nm和660 nm持续90 s,缔合/解离速率常数和解离常数(Kd)通过BIAevaluation软件(version 4.1.1;通用电气医疗集团)。
体外转录径流试验
体外径流试验如前所述进行3..用4RO-F/4RO-R引物pcr扩增P的启动子和操作子序列模板6650(198 bp)和P的CRO-F/CRO-R468(303 bp)(表S3).将0.5 pmol的模板与2 pmol混合大肠杆菌RNA聚合酶全酶或核心酶(AR Brown, Tokyo, Japan), 100 nmol核糖核苷酸(包括[α-32P] CTP), 0-4 pmol PmlR2, 0-96 pmol PmSB-LOV。样品用42 μ mol m照射−2年代−1用6 M尿素聚丙烯酰胺凝胶电泳分析转录本。标记10 (pBR322/ .Msp我消化)(Nippon Gene,东京,日本)被标记为[γ-32P] ATP,用作分子标记。如上所述进行自放射显像。
意义的声明
“短”LOV蛋白是广泛分布在植物基因组中的蓝光感光元件假单胞菌虽然“短”LOV蛋白的生化功能已经被很好地研究,但任何相关的效应蛋白尚未被确定。我们首次揭示了属于MerR家族的转录调节蛋白PmlR2作为“短”LOV蛋白的伴侣蛋白。PmlR2和PmSB-LOV同源物的守恒假单胞菌sp .和密切相关的细菌表明光感应和反应是在自然界生存所必需的。
数据可用性
在本研究中生成的RNA-seq数据集可在DRA/SRA数据库存储库中获得。DRA014932。
参考文献
Jost, M。et al。基因调控的结构基础12端依赖光感受器。自然526, 536-541(2015)。
高野,H., Obitsu, S., Beppu, T. & Ueda, K.光诱导胡萝卜素发生链霉菌属coelicolorA3(2):一种胞外质功能sigma因子的鉴定,该因子指导类胡萝卜素生物合成基因簇的光依赖性转录。j . Bacteriol。187, 1825-1832(2005)。
Takano, H。et al。LitR是一种腺苷b12结合光敏调节因子芽孢杆菌megateriumQM B1551,类胡萝卜素生产的调节。j . Bacteriol。197, 2301-2315(2015)。
Takano, H。et al。CarA/LitR和CRP/FNR家族转录调节因子参与光诱导的类胡萝卜素产生栖热菌属酸奶.j . Bacteriol。193, 2451-2459(2011)。
Perez-Castano, R。et al。辅酶B12-依赖和独立的光调节的胡萝卜素生成黏球菌。环绕。Microbiol。24, 1865-1886(2022)。
非光养细菌中光诱导生产类胡萝卜素的调节机制。Biosci。Biotechnol。物化学。8451, 1-10(2016)。
库塔,r.j.。et al。a B的光化学机理12依赖的感光蛋白。Commun Nat。6, 7907(2015)。
Sumi, S., Shiratori-Takano, H., Ueda, K. & Takano, H. III类LitR的作用和功能,一种光电传感器同源物伯克multivorans.j . Bacteriol。200, e00285-18(2018)。
烟灰墨,S。et al。光响应假单胞菌putidaKT2440介导的II类LitR,光敏传感器同源物。j . Bacteriol。202, e00146-20(2020)。
贺璐,李志强,李志强,等。细菌光感LOV蛋白的功能、结构及机制研究。Nat. Rev.微生物。9, 713-723(2011)。
Losi, A., Mandalari, C. & Gärtner, W.黄素原核光感受器的进化和功能作用。Photochem。Photobiol。91, 1021-1031(2015)。
盖登科,T. A.,金,T. J.,魏格尔,A. L.,布罗迪,M. S. & Price, C. W.蓝光受体YtvA在环境胁迫信号通路中的作用枯草芽孢杆菌.j . Bacteriol。188, 6387-6395(2006)。
斯沃茨,t.e.。et al。蓝光激活组氨酸激酶:细菌中的双组分传感器。科学317, 1090-1093(2007)。
亚历山大,m.t.a。et al。光感组氨酸激酶的电子和蛋白质结构动力学。生物化学49, 4752-4759(2010)。
Purcell, E. B., Siegal-Gaskins, D., Rawling, D., Fiebig, A. & Crosson, S.光感觉双组分系统调节细菌细胞附着。Proc。国家的。学会科学。104, 18241-18246(2007)。
Rollen, K。et al。从晶体结构和溶液核磁共振谱揭示了短蓝光感光蛋白ppss1 - lov的信号状态。J. Mol.生物学。428, 3721-3736(2016)。
斯塔德勒et al。光激活降低了LOV光感受器的侧链动力学。Biophys。J。110, 1064-1074(2016)。
延奇,K。et al。两种短LOV结构域蛋白通过扩展突变相互交换动力学性质假单胞菌putida.生物化学48, 10321-10333(2009)。
Arinkin, V。et al。的LOV蛋白中加合物寿命的结构决定因素假单胞菌putida.2月J。288, 4955-4972(2021)。
Metz, S., Jäger, a . & Klug, G.短光、氧、电压(LOV)结构域蛋白在蓝光中的作用及单线态氧依赖基因调控Rhodobacter sphaeroides.Microbiol。(N Y)158, 368-379(2012)。
Endres, S。et al。海洋光养细菌LOV短蛋白的结构和功能Dinoroseobacter shibae.BMC Microbiol。15, 30(2015)。
paleroni, N. J., Doudoroff, M., Stanier, R. Y., Solánes, R. E. & Mandel, M.好氧假单胞菌的分类:性质假单胞菌stutzeri组。J. Gen.微生物。60, 215-231(1970)。
粉丝,X。et al。基因组还原增强了聚羟基烷酸和海藻酸低聚糖的生成假单胞菌mendocina.Int。生物。絮凝。163, 2023-2031(2020)。
阿瓦亚,J. D.和杜波伊斯,J. L.鉴定,分离,和分析参与铁获取基因簇假单胞菌mendocinaymp。Biometals21, 353-366(2008)。
Pribat,。et al。FolX和FolM在四氢蝶呤合成中是必不可少的大肠杆菌而且铜绿假单胞菌.j . Bacteriol。192, 475-482(2010)。
石滨,A,岛田,T. &山崎,Y.的转录谱大肠杆菌:基因组SELEX搜索转录因子调控靶点。诊断。酸Res44, 2058-2074(2016)。
Takano, H., Mise, K., Maruyama, T., Hagiwara, K. & Ueda, K.半保守组氨酸残基在mre型光感觉转录调控子LitR光敏感域中的作用。Microbiol。(读)。162, 1500-1509(2016)。
康拉德,K. S., Bilwes, a . M. & Crane, B. R.光诱导亚基解离由光氧电压域光感受器从Rhodobacter sphaeroides.生物化学52, 378-391(2013)。
纳什,A.我。et al。细菌光氧电压/螺旋-旋转-螺旋(LOV-HTH) dna结合蛋白光敏性的结构基础。Proc。国家的。学会科学。美国108, 9449-9454(2011)。
Tschowri, N. Lindenberg, S. & Hengge, R.生物膜调节蓝光信号通路的分子功能和潜在进化大肠杆菌.摩尔。Microbiol。85, 893-906(2012)。
曼尼提斯,T.弗里奇,E. F. &桑布鲁克,J.。分子克隆:实验室手册.(冷泉港实验室出版社,冷泉港,纽约,1982年)。
Livak, K. J. & Schmittgen, T. D.使用实时定量PCR和2(-Delta Delta C(T))方法分析相关基因表达数据。方法25, 402-408(2001)。
Haberle, V., Forrest, A. R. R., Hayashizaki, Y., Carninci, P. & Lenhard, B. CAGEr:用于综合分析的精确TSS数据检索和高分辨率启动子组挖掘。诊断。酸Res。43, e51(2015)。
弗里斯,m.c。et al。哺乳动物基因组中转录起始的编码。基因组Res。18, 1-12(2007)。
曹志刚,李志刚,李志刚,李志刚,李志刚。植物病原菌蓝光诱导双组分信号转导系统的研究两pv。番茄。Biophys。J。94, 897-905(2008)。
Bencini, D. A., O 'Donovan, G. A. & Wild, J. R.快速化学降解测序。生物学技术2, 4-5(1984)。
李志强,李志强,李志强,李志强。一种基于重组信号转导途径的细菌双杂交系统。Proc。国家的。学会科学。95, 5752-5756(1998)。
高野,H.,细野,K., Beppu, T. & Ueda, K. σ的参与H以及相关的sigma因子在糖依赖启动的形态学和生理发育链霉菌属将.基因320, 127-135(2003)。
确认
我们感谢Shoichi Amano的HPLC分析,以及东京农业大学NODAI基因组研究中心生物资源基因组研究合作项目对RNA测序的支持。本研究由日本文部科学省HT科学研究资助基金(B) (No. 22H02248)资助;野田科学研究所;NAGASE科学技术发展基金会;慈善信托荒木医学生化研究纪念基金。
作者信息
作者及隶属关系
贡献
H.T.设计了实验。T.M.进行重组蛋白的过表达和纯化、发色团的HPLC分析、光谱分析和凝胶过滤色谱。S.S.进行总RNA制备、定量RT-PCR、凝胶过滤层析数据分析。M.K.对重组蛋白进行过表达、纯化和凝胶过滤层析。T.E.进行BACTH系统分析。K.Y.和H.Y.进行RNA测序分析。H.T.进行了转录起始位点分析、DNase I足迹分析、凝胶位移分析、BIAcore分析和体外转录径流分析。H.T.写了手稿。H.T.整合了整个研究项目。所有作者讨论了结果并对手稿进行了评论。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
丸山,T,苏美,S,小林,M。et al。II类LitR作为“短”lov型蓝光感受器的效应器假单胞菌mendocina.Sci代表12, 21765(2022)。https://doi.org/10.1038/s41598-022-26254-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-022-26254-3