摘要
病毒适应度的改变不能仅从对分离病毒蛋白的诱变研究中推断出来。迄今为止,还没有进行系统分析来确定提高病毒适应性和降低药物疗效的突变。我们提出了一种评估哪些病毒突变可能降低药物疗效的通用策略,并将其应用于评估SARS-CoV-2的进化如何影响目前批准/候选的小分子抗病毒药物的疗效箴, PL箴,和RdRp。对于每个药物靶点,我们从可用结构中确定了药物相互作用的病毒残基,并从SARS-CoV-2基因组中确定了病毒残基的选择压力。这使得有希望的药物靶点区域和病毒可能产生耐药性的小分子抗病毒药物得以识别。我们利用基因组序列和蛋白质结构数据库中的序列和结构信息的策略可以快速评估任何新出现的病毒变体的适应性,并可以帮助针对未来病原体设计抗病毒药物。
简介
两年多前,严重急性呼吸综合征冠状病毒2型(SARS-CoV-2)引发了一场大流行,造成600万人死亡,经济、教育和社会互动中断,影响了数十亿人的生计。自发现该病毒以来,出现了大量的出版物和预印本,试图(i)找到这种病毒的起源及其进化,(ii)描述病毒的生命周期和发病机制,以及(iii)开发预防性疫苗或治疗方法。然而,很少有人努力阐明SARS-CoV-2蛋白的未来突变是否会使已批准/候选药物的疗效失效。由于病毒适应度的改变不能从对分离的病毒蛋白质的诱变研究中推断出来,因此通过费力扫描病毒突变体发现的一些药物逃逸突变体,如果它们降低了病毒适应度,则可能不存在。另一方面,每天以前无法对病毒基因序列进行大规模分析,但现在可以对SARS-CoV-2进行大规模分析。在这里,我们提出了一种通用策略,以评估哪些病毒突变将降低药物疗效,使用蛋白质编码区病毒基因序列的进化分析,结合病毒蛋白质-药物相互作用的生化/结构数据。我们通过使用这一策略来预测SARS-CoV-2对当前小分子抗病毒药物耐药的近期可能性,来说明这一策略。
尽管COVID-19预防性疫苗非常成功,但事实证明,提供有效的药物来治疗COVID-19要困难得多。针对COVID-19的治疗工作主要集中在开发抑制过度反应的免疫反应的药物或包括小分子、多肽和单克隆抗体(mAbs)的抗病毒药物上。1.迄今为止,美国食品和药物管理局(FDA)已按时间顺序批准了6种抗sars - cov单抗,即(i) casirivimab + imdevimab (REGEN-COV), (ii) bamlanivimab + etesevimab, (iii) sotrovimab, (iv) tocilizumab (Actemra™),(v) tixagevimab + cilgavimab (Evusheld™)和(vi) bebtelovimab。在已发表的许多针对SARS-CoV-2的小分子抗病毒药物中,瑞德西韦(Veklury™)是一种SARS-CoV-2 RNA依赖RNA聚合酶(RdRp或nsp12)的核糖核酸抑制剂。2,3.baricitinib (Olumiant™)是首个获得FDA批准的药物,baricitinib是一种宿主蛋白JAK1和JAK2的选择性抑制剂4.此外,FDA还批准了ritonvir增强的nirmatrelvir (Paxlovid™)和(Lagevrio™)的EUA。与瑞德西韦一样,molnupiravir也针对SARS-CoV-2 RdRp,但与瑞德西韦不同的是,它是一种延迟链终止剂,可以阻止病毒RNA合成5, molnupiravir作为诱变剂,增加病毒突变率,导致病毒拷贝功能失调6.Nirmatrelvir是一种可逆的SARS-CoV-2主要蛋白酶(M箴ritonavir是一种HIV-1蛋白酶抑制剂,通过抑制其细胞色素P450 3a介导的代谢,使nirmatrelvir保持更长时间的活性7.
在进化过程中,病毒会发生突变,促进有益等位基因的传播(积极/多样化选择)或阻碍有害等位基因的传播(消极/净化选择)。8.因此,SARS-CoV-2蛋白的某些突变可能会降低药物疗效,这是一个主要问题。事实上,目前流通的Omicron变体的刺突蛋白的大量突变显著降低了REGEN-COV、bamlanivimab + etesevimab和sotrovimab的疗效,导致这三种单抗疗法在美国停止使用,而bebtelovimab对Mu变体的疗效降低。人们已经尝试确定SARS-CoV-2峰值的突变三聚物的通过产生突变体,表达突变体,并确定它们是否影响原生病毒折叠和功能,如果不影响,它们如何影响抗体结合,从而逃避中和抗体的糖蛋白9,10,11.结果取决于(i)给定病毒蛋白的所有可能的氨基酸(aa)突变的覆盖范围,(ii)表达系统是否以其功能性、原生低聚物/糖基化状态表达病毒蛋白,以及(iii)结合测定的敏感性。由于需要在一个孤立的实验室设备中生产大量的病毒突变蛋白,很少有这样的研究已经完成。此外,某种SARS-CoV-2蛋白的突变可能会影响其与其他病毒蛋白的相互作用,从而影响SARS-CoV-2的适应性。
此外,在硅研究几个12,13,14,15,16,17,18利用序列分析、SARS-CoV-2变体结构建模、分子动力学/对接模拟等工具,预测了a的突变具体的病毒蛋白,可能改变其结构/灵活性,从而对某些药物的敏感性。然而,据我们所知,目前还没有进行系统分析来评估SARS-CoV-2突变是否存在正/负选择,这将以不同的方式改变药物疗效:如果给定病毒蛋白的药物相互作用残基突变处于负选择,则它们将恢复以防止伤害病毒;因此,这种替代在短期内可能无法逃脱电流抑制。相反,如果它们处于正向选择,它们将受到极大关注,因为它们将提高病毒的适应性,并可能否定药物作用。
在这里,我们提出了一种策略,通过从3D结构中确定药物相互作用的病毒残基,并使用来自基因组序列的进化信息对其选择压力进行分类,来评估哪些病毒突变可能会降低药物疗效。如果一个残差的变异速度比中性漂移所预期的要快(或慢),那么它就被认为处于正选择(或负选择)下。然后,我们应用我们的策略来预测病毒对科学文献中目前批准/候选的针对SARS-CoV-2蛋白的小分子药物产生耐药性的可能性。这是及时的,因为可以获得丰富的SARS-CoV-2基因组序列和许多SARS-CoV-2蛋白/抑制剂复合物的3D结构。我们的研究结果有助于阐明当前SARS-CoV-2对已批准/候选小分子药物的耐药潜力。由于大多数国家在COVID-19大流行后建立了大规模基因调查能力,我们的通用策略可用于帮助选择针对其他病毒的候选抗病毒药物用于临床开发。
方法
小分子的选择批准/候选
为了获得小分子SARS-CoV-2抑制剂,我们使用以下关键词搜索PubMed数据库:“SARS-CoV-2,药物,靶标或蛋白质”。截至2021年9月,该研究共发表了约10,000篇论文和预印本。我们通过排除所有针对宿主蛋白或生物制剂(如多肽和单克隆抗体)的已批准/候选药物或包含已知和未知化合物(如植物叶子和其他传统药物元素)混合物的候选药物的论文来减少这一数字。此外,我们排除了病毒蛋白靶点未知或对病毒蛋白靶点或整个病毒的影响尚未经过实验验证的候选药物,例如仅通过硅质筛选的候选药物。然而,我们没有判断完成的实验的质量,但认为直接的病毒抑制和实验分析显示抑制剂与病毒蛋白靶标的相互作用是足够的。最后,我们只保留了那些经实验验证的小分子抑制剂,它们与病毒蛋白残基的相互作用是从晶体/停靠结构中已知的。同样,我们没有判断用于识别此类药物相互作用残基(如病毒蛋白抑制剂结构的质量)的方法。补充表S1列出最终的候选药物及其病毒蛋白靶点。我们并不声称这个列表是全面的,因为由于大量的出版物,一些候选药物可能会被省略;此外,新的候选药物也在不断地被报道。
进化分析
人类宿主病毒蛋白及其来自RefSeq和GenBank全基因组的编码序列19使用NCBI数据集服务从NCBI获得(2022年1月11日)。随着每天序列数量的快速增长,对全序列数据集进行分析变得不可行的。因此,对于给定的病毒药物靶蛋白,我们随机采样了20,000个不同的病毒蛋白序列,使用MaffT v7.487对其进行比对20..在多蛋白序列比对的指导下,我们使用HyPhy 2.5.32 (MP)包中的msa-密码子工具对病毒药物靶蛋白的编码序列进行比对21.得到的多核序列比对提供给IQTree 2.1.322为病毒蛋白靶标建立系统发育树。用于估计树的模型由IQTree在优化搜索过程中选择。为了分析病毒蛋白靶标每个位点的选择压力,我们采用HyPhy 2.5.32 (MP)包中的固定效应似然(FEL)方法21,23,该方法估计了每个站点的非同义和同义替换率。默认p值(p < 0.1)被用作阈值,将选择分为消极或积极。没有对重组进行分析,因为研究发现了适度的重组事件证据,一些重组事件可能是另一种解释24,25.为了确认上述程序得到的结果的稳定性,我们对原始数据库进行了共10轮的采样。
结构分析
我们在蛋白质数据库(Protein Data Bank, PDB)中以最佳分辨率从蛋白质-药物结构中提取了药物相互作用病毒残基。26,或者,如果这种结构不存在,则从候选药物已被停靠的已发表的停靠结构到已知的蛋白质实验结构。由于缺乏单个残基对药物结合的绝对自由能贡献的实验数据,我们没有试图对药物相互作用病毒残基的重要性进行排名。为了以一种可访问的方式向从事药物设计的研究人员展示进化分析结果,我们使用SIFTS将我们基于序列的负选择和正选择数据映射到相应蛋白质的晶体结构27.对药物结合残基采用PDB残基编号。
结果
通过调查PubMed数据库,我们确定了149种经实验验证的小分子抑制剂,其SARS-CoV-2药物靶点和药物相互作用病毒残基已知。它们包括fda批准的药物瑞德西韦,以及eua批准的nirmatrelvir,但不包括molnupiravir,因为没有molnupirvir结合的SARS-CoV-2 RdRp结构。补充表S1列出了每个病毒蛋白靶点、候选药物、病毒蛋白/抑制剂复合体的PDB代码以及药物相互作用的SARS-CoV-2残基。
大多数候选药物在补充表中S1针对特定的病毒蛋白。然而,其中一些可以与同一蛋白质的多个位点结合。例如,临床试验中的抗癌药物YM155在木瓜蛋白酶样蛋白酶(PL箴)在SARS-CoV-2 PL晶体结构中箴-YM155复杂28.6种候选药物;即苏拉明,槲皮素,化合物7而且13, ebselen和disulfiram针对多个SARS-CoV-2蛋白。苏拉明是一种带高度负电荷的分子,已被用于治疗非洲昏睡病和河盲症,它与SARS-CoV-2 M结合箴和RdRp。它被认为作用于M箴引起构象变化,改变蛋白酶活性29.它也可以结合到RdRp活性位点,阻断RNA模板和引物链的结合30..槲皮素,被鉴定为SARS-CoV-2 M箴竞争性抑制剂通过活性为基础的实验筛选,结合到M箴催化部位31以及刺突受体结合域32.它对蛋白酶稳定性表现出剂量依赖的不稳定效应,并抑制刺突与人血管紧张素转换酶2之间的相互作用32.化合物7而且13通过基于药效团的虚拟筛选发现,它们是M箴和PL箴以及人类的糠醛蛋白酶33.Ebselen和disulfiram是Zn2 +-喷射化合物,可以同时针对活性半胱氨酸(游离或锌2 +-结合)在多个SARS-CoV-2非结构蛋白(nsps)中,包括复制转录复合体,复制并产生编码辅助蛋白和结构蛋白的亚基因组mrna34,35,36.值得注意的是,ebselen与M箴,如2.05 Å晶体结构的ebselen结合到M箴37.
结果见补充表S1表明开发SARS-CoV-2抗病毒药物的努力集中在(i) nsp5 M箴(ii) nsp3 PL箴(iii) nsp12 RdRp催化结构域。两米箴和PL箴通过自身的蛋白水解活性从病毒多蛋白(pp1a和pp1ab)中切除。对于这3种药物靶蛋白,我们在下面从蛋白质数据库(PDB)的可用结构中概述了病毒蛋白的功能、总体结构和不同的结合位点/基序。26.然后,我们描述了药物配体的结合位置和药物结合残基的选择压力,这些残基根据各自的PDB结构而不是编码序列进行编号。我们强调SARS-CoV-2 M箴, PL箴阳性选择下的RdRp残基,因为它们可能根据其报告的作用影响药物疗效。
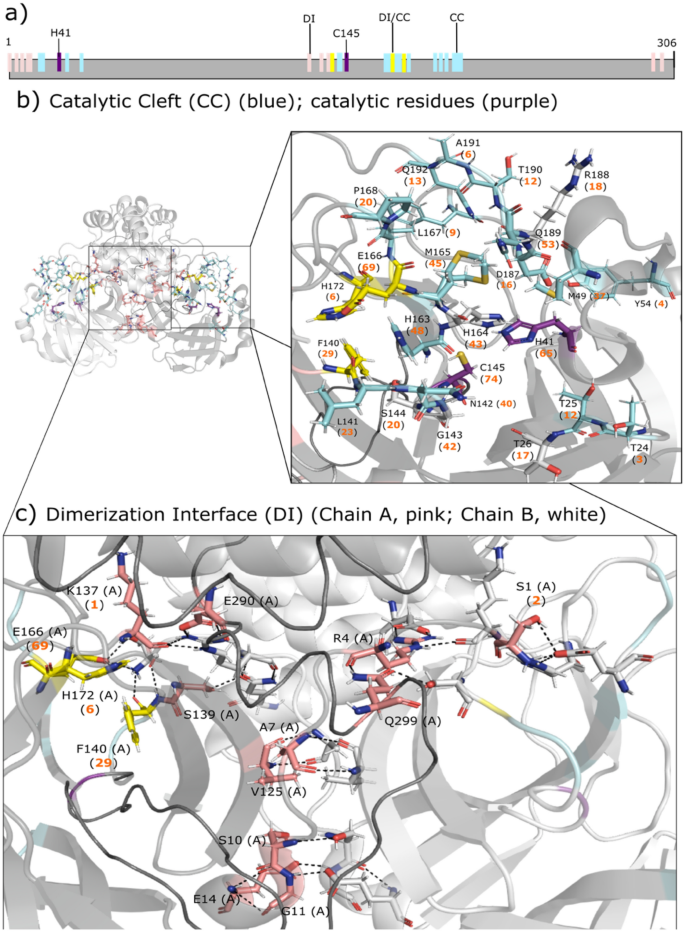
SARS-CoV-2米箴(3氯箴或nsp5)
主要蛋白酶(M箴),也称为3-凝乳胰蛋白酶样蛋白酶(3CL .箴)或nsp5是一种半胱氨酸蛋白酶,它将两种病毒多蛋白裂解为16个组成NSPS,这对病毒复制和成熟至关重要。它是最受欢迎的SARS-CoV-2 nsp药物靶点,因为(i)它在病毒复制中起着先决条件作用,(ii)它没有人类同源物,但在冠状病毒中保守,以及(iii)它具有独特的裂解特异性,与已知的人类半胱氨酸蛋白酶不同,它在Gln后裂解序列38,39,40,41.因此,针对M箴会减少脱靶活动,从而减少副作用吗42.
单体的米箴由一个n端指(残基1-7)和三个结构域组成:糜蛋白酶样结构域I(残基8-101),小核糖核酸病毒3C蛋白酶样结构域II(残基102-184)和结构域III(残基201-306)43.M需要二聚体箴由于两个原聚体之间的相互作用,特别是一个原聚体的n端S1与另一个启动子的E166之间的相互作用,使酶保持活性构象38.因此,n端手指,E166和独特的催化C145-H41二元体在蛋白水解活性中起着至关重要的作用。米箴有两个不同的结合区域(图;1):(i)一个底物结合位点,包含催化C145-H41二元体,位于结构域i和结构域II之间的裂口;(II)二聚化界面,包括来自n端指、催化裂口和结构域III的残基40,44,45,46.
数字1b c表示M的个数箴括号中的抑制剂靶向(i)催化C145-H41二分体(紫色),(ii)底物结合残基(浅蓝色),(iii)二聚体界面残基(粉红色),以及(iv)催化裂口和二聚体界面共有的残基(黄色)。所有94个抑制剂均以M箴包括eua批准的药物nirmatrelvir (PF-07321332)。它们最常见的目标是催化C145-H41二元体(74和65个化合物)以及E166(69个化合物),这对二聚化很重要。然而,94种候选药物中的3种(奥美拉唑、punicalagin和chebulagic acid)也靶向二聚体界面上的两个残基(S1和K137)。金芩苷和车布酸也是M箴酶活性29,47.
数字2描述的是SARS-CoV-2 M箴在十轮抽样中的任何一轮中显示负选择(蓝色)或正选择(红色)证据或无负/正选择证据(白色)的残基。例如,在10轮抽样中,催化C145在4轮中显示出负选择的证据,但在其他轮中没有正/负选择的证据。大部分残基被M箴抑制剂45,46;即T25、T26、H41、Y54、K137、F140、L141、N142、S144、C145、H163、H164、E166、L167、P168、H172、D187、R188、Q189、T190、Q192为负选择。其他药物相互作用残基(S1, T24, M49, G143, M165)没有阴性/阳性选择的证据,但高度保守。正选择下的残基不直接与M相互作用箴除A191外的抑制剂。
A191在10轮抽样中有2轮显示出阳性选择的证据。它由6种药物靶向;即,PF-00835231,埃弗尼地平,奈非那韦,双吲哚基马来酰亚胺IX,以及化合物2而且151.PF-00835231是一种酮基共价抑制剂,与A191骨架形成范德华相互作用48.然而,由于其口服生物利用度低,它已被口服药物PF-07321332 (eua批准的nirmatrelvir)所取代,后者在正选择压力下不与任何残留物相互作用。有趣的是,G15、K90和P132在当前的SARS-CoV-2变体中经常发生突变43,都是正选择。由于K90突变Arg有望改善二聚43,它可能会影响靶向二聚体界面的化合物。
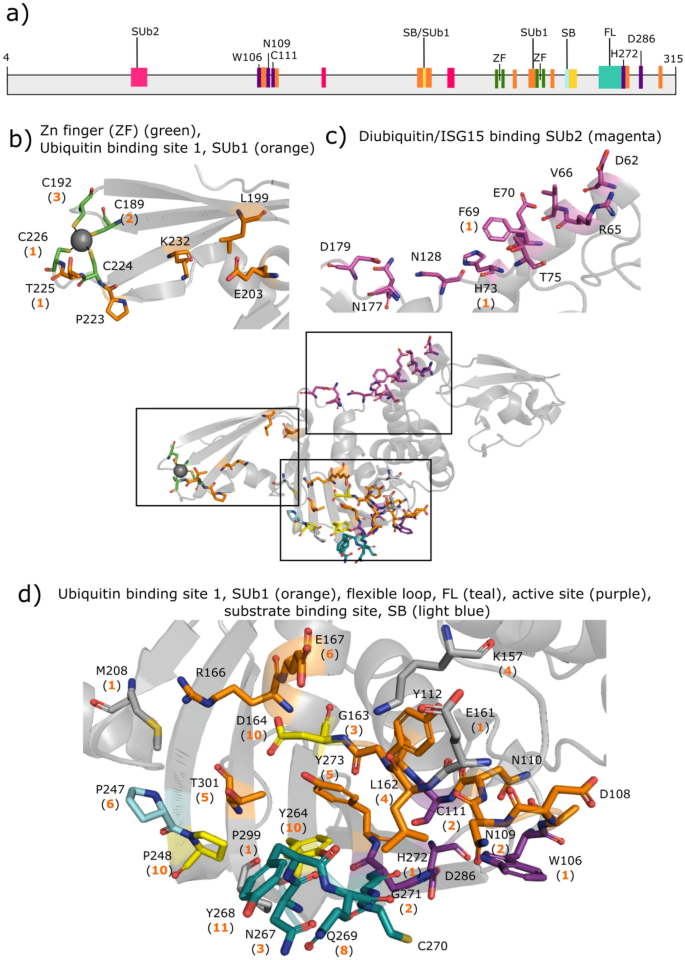
SARS-CoV-2 PL箴
SARS-CoV-2 nsp3编码PL箴蛋白酶也是一种流行的药物靶点,因为它参与病毒复制和宿主免疫反应抑制,并且在冠状病毒中是保守的41,49.该蛋白酶在病毒多蛋白的nsp1/2、nsp2/3和nsp3/4边界以及宿主泛素和干扰素刺激基因15 (ISG15)的c端识别LXGG↓(X)切割motif50.因此,除了裂解病毒底物,PL箴也能切割宿主蛋白质上的翻译后修饰以逃避抗病毒免疫反应51.不像米箴, PL箴使用催化剂三合会(C111-H272-D286),具有催化活性单体.PL箴由n端类泛素子结构域和右手拇指-手指-手掌催化单元组成49.它有四个结合位点(图;3.a): a Zn2 +-结合位点,病毒底物结合通道,以及两个宿主泛素/ isg15结合子位点SUb1和SUb228,41,43,44,49,52.的锌2 +-结合位点,由4个保守的半胱氨酸排列(图;3.B),对结构完整性和蛋白酶活性至关重要53.SUb2子位点由D62、R65-V66、F69-E70、H73、T75、N128、N177和D179组成。3.c). SUb1子站点由W106-Y112、E161-D164、R166-E167、L199、E203、P223、T225、K232、P248、Y264、Y268-G271、Y273、T301组成。3.d)54.值得注意的是,W106和N109有助于稳定肽水解的氧阴离子过渡态41而L162和E167参与了与宿主ISG15的相互作用55.SUb1子位点部分重叠于含有C111-H272-D286催化三联体、G163-D164、P247-P248、Y264和一个称为BL2的柔性环的病毒底物结合通道(残基267-271)。41,43,44,49,52.BL2环很重要,因为它可以识别病毒蛋白之间的LXGG基序,并在底物/抑制剂结合时关闭52.
SARS-CoV-2 PL箴域结构和绑定位点。(一个图中显示了活性位点催化残基(紫色为W106, C111, H272, D286),底物结合残基(SB,浅蓝色),柔性环上的残基267-271 (FL,蓝绿色),SUb1和SUb2的残基(品红),活性位点裂口和SUb1的残基共有的残基(黄色)。载波PL的1.90-Å晶体结构箴(PDB 7 d7k)28以及针对Zn中残留的药物数量(括号中)2 +-绑定站点(b)、SUb2子网站(c),以及与SUb1子站点(d).
大部分PL箴抑制剂靶向活性位点裂缝,3个化合物靶向Zn2 +在sub2结合位点上只发现了一个化合物(YM155)。3.).大多数候选药物针对的是sub1结合位点上与底物结合有关的残基。其中Y268是最常见的药物靶向残留(11种化合物),其次是D164、P248和Y264(各10种化合物),Q269(8种化合物)。两种化合物,VIR250和VIR251,共价键与催化C11156.
无花果的比较。2而且4表明PL中存在较多的正选择残基(红色残基)箴比在M箴.几乎所有正选择下的药物相互作用残基都位于SUb1子位点,该子位点结合宿主泛素和ISG15蛋白。这些残基包括Y268、Y264、G271和T225,它们分别被11、10、2和1个抑制剂靶向。值得注意的是,BL2环上的Y268可以与候选药物形成氢键和/或π-堆叠相互作用;因此,其突变可以影响BL2环构象,减弱药物相互作用。事实上,SARS-CoV-2 PL的突变箴Y268对Thr或Gly显著降低了非共价抑制剂GRL-0617的抑制作用51.另一个阳性选择下的药物相互作用残基是P299,它只与一种候选药物XR8-24形成疏水接触57.有趣的是,SARS-CoV-2 PL . 2.1 Å晶体结构箴-YM155配合物(PDB 7D7L)表明,YM155与Zn中的(i) C192、Q195、T225和C226形成了范德华斯键或氢键相互作用2 +(ii)病毒底物结合通道中的P248、Y264、Y268和Y273; (iii) SUb2子位点中的F69和H7328.虽然C192和H73是负选择,但相邻的G193和Y71分别是正选择。由于G193、T225、Y264、Y268和Y71为正选择,其突变可能减弱YM155与这3个位点的结合。
除Y71和G193外,其他几个正选择残基也在药物相互作用残基附近。带正电的K232靠近带负电的Zn2 +网站(无花果。3.b),其对存在于SARS-CoV-2 γ变异(K232Q)中的Gln的突变增强了体外泛素切割,这可能会影响感染细胞中的宿主免疫反应54.R166靠近两个常见的药物相互作用的酸性残基D164和E167,而(V159, G160), (Y207, G209, T210)和K297分别与E161, M208和P299相邻,它们都只与一种抑制剂相互作用(图2)。3.d).令人惊讶的是,即使D286是催化三联征的一部分,它也处于正选择状态。D286通过与H272侧链形成氢键,使H272在催化过程中成为一般的酸/碱43.D286的这种作用可能由M箴,它缺乏第三种催化残留物。
RNA依赖RNA聚合酶(nsp-12)
nsp12 RdRp是另一个关键的药物靶点,因为它负责病毒RNA的合成,并且在冠状病毒中高度保守,没有已知的哺乳动物同源物16.nsp12亚基由三个结构域组成:n端nidvirus RdRp相关核苷酸转移酶结构域(NiRAN,残基Q117-A250),界面结构域(残基L251-R365)和手指-手掌-拇指RdRp催化结构域(残基L366-L932)41.nsp12本身表现出很少或没有聚合酶活性,这需要nsp7和nsp8辅助因子的帮助来增加nsp12与模板引物RNA的结合5.两个守恒Zn2 +-binding motif (H295, C301, C306, C310和C487, H642, C645, C646)保持RdRp的结构完整性5.除了两个Zn2 +-结合位点,7个保守结构基序(标记一个- - - - - -G)参与RNA模板链和引物链和/或进入的核苷酸的结合。在模板定向RNA合成过程中,单链RNA模板沿由基序夹紧的凹槽通过F(T538-V560)和G(K500-R513)并进入由motif组成的活性位点模拟58.图案一个(N611-M626)和C(F753-N767)含有催化618DX4D623而且759SDD761基序,其中保守的酸性残基参与调节催化活性和结合两个催化Mg2 +离子58.主题B(T680-T710)包含一个柔性环(S682-T686),参与新生dsRNA的模板绑定和易位58.主题E(H810-K821)与引物RNA链相互作用5,而母题D(L775-E796)和F与进入的NTP磷酸基相互作用58.
几乎所有已确定的nsp12候选药物,包括fda批准的瑞德西韦,都靶向包含nsp12催化结构域保守结构基的残基。它们最常与基序F中带正电的R555相互作用,该基序接触引物链RNA的+ 1碱基,在催化中带负电的D623618DX4D623基序B中的S682和N691(见图。5).所有nsp12候选药物均未与两个Zn结合2 +-sites or motif;
SARS-CoV-2 RdRp结构域结构和结合位点。(一个nsp12中7个保守基序(A-G)和低温电镜结构(PDB 7aap)的图解。(b)插图显示了针对属于7个保守基序(a - g)之一的残基的药物数量(括号中)58.
大多数药物相互作用的残基,特别是759SDD761催化残基处于负选择状态(图;6).值得注意的是,在瑞德西韦延迟链终止机制中起关键作用的S861处于负选择状态。然而,包括瑞德西韦在内的SARS-CoV-2 RdRp抑制剂最常靶向的R555没有显示出阴性/阳性选择的证据。另一方面,体外进化研究已经确定了三种nsp12突变体,即S759A、V792I和E802(A/D),可以对瑞德西韦产生耐药性17,18,59.但是,S759包含759SDD761催化基序和V792都存在负选择,表明它们的突变会降低SARS-CoV-2的适应度。尽管高度保守的E802没有显示出阴性/阳性选择的证据,但在体外实验中,E802(A/D)突变体相对于野生型SARS-CoV-2 nsp12降低了病毒复制,这表明E802突变具有适应度成本59.
没有药物相互作用的SARS-CoV-2 RdRp残基处于阳性选择状态;然而,有些是正选择下的近残基。例如,T324在所有10轮采样中都显示出阳性选择的证据,它旁边的两个脯氨酸(P322和P323)被预测会与抑制剂taroxz -104相互作用60.另一个正选择残基T582接近于A580,在与苏拉明结合的SARS-CoV-2 RdRp (PDB 7d4f)晶体结构中与苏拉明存在填料相互作用。
讨论
COVID-19大流行的一个重要副产品是,大多数国家已经建立了扩展基因组调查能力,以监测和分析病毒基因组的变化。这些调查能力可应用于未来的流行病/大流行。因此,我们建议使用从基因组数据库获得的信息来支持抗病毒药物的设计。在此,我们通过从SARS-CoV-2基因序列的大规模分析中提取进化趋势,说明如何将这些数据纳入抗病毒药物设计的早期阶段。这使我们能够确定良好的SARS-CoV-2药物靶点和具有高概率抗病毒耐药性的候选药物短术语。与我们提出的策略相反,以前的研究通常采用整个冠状病毒家族的保守作为替代,以确定良好的病毒药物靶点,并将高频突变与抗病毒耐药性的可能性相关联;例如,米箴最容易发生突变的残基被认为是潜在的耐药位点46.然而,在非结构蛋白中看到的突变频率并不能提供直接证据,证明突变对病毒是有益的而不是有害的。我们在病毒序列库的每个剩余位置都观察到了一些变异,因此我们可以计算不提高病毒适应性的突变。
对SARS-CoV-2药物靶点和候选药物的影响
在三种最受欢迎的SARS-CoV-2药物靶点中,M箴的残基数量最少,显示正选择,而PL箴(比较无花果。2,4而且6).因此,针对M箴或RdRp活性位点具有比靶向PL更多的进化支持箴活跃的站点。我们的结果进一步表明,有希望的药物靶点区域包括负选择下的残基,而不是在空间上靠近正选择下的残基。例如图中RdRp的结果。6在负选择条件下,指明两个包含残基的相邻区域(494IVNNLDKS501而且840AGCFVDDIV848),正选择下最接近的余数为> 8 Å。
尽管正选择下的残基可能不会与给定的药物直接相互作用,但其突变可能调节药物的变构相互作用,并可能产生耐药性。因此,药物靶向的残基/区域显示负选择多个与针对单一病毒蛋白的药物相比,必需病毒蛋白可以更好地对抗新突变带来的危险。事实上,锌2 +-引射药物(ebselen, disulfiram)已被证明同时靶向催化和/或Zn2 +5种SARS-CoV-2蛋白中的结合半胱氨酸;即,米箴61, PLpro34, nsp10 (nsp14和nsp16的辅因子)34, nsp13 RNA解旋酶/5 ' -磷酸酶35,以及nsp14外切酶结构域35.与依布色林/双硫仑相反,拟肽候选药物不能同时作用于M箴和PL箴同时,由于这两种病毒蛋白酶具有相当不同的底物特异性50;因此,它们各自的抑制剂必须联合使用。为了最大限度地降低耐药出现的风险和最大限度地提高药效,我们建议将多靶向临床安全的ebselen/disulfiram与靶向M的强效抑制剂联合使用箴和/或在负选择下的RdRp残基。的确,ebselen/disulfiram联合靶向nsp3 PL箴, nsp5 M箴remdesivir靶向nsp12 RdRp后,nsp10, nsp13和nsp14被证明是有效的表现为协同作用在Vero E6细胞中抑制SARS-CoV-2复制35.
限制
由于缺乏单个病毒残基对药物结合的自由能贡献的实验数据,我们无法评估药物相互作用残基或其aa变化对药物结合的影响。注意,阳性选择位点上的aa变化对药物结合的影响并不相同,因为一些变化可能完全取消药物的作用,而另一些变化可能只对药物结合有轻微的影响。此外,药物靶蛋白中所有aa变化的集体效应决定了耐药性。请注意图中的结果。2,4,6基于当前SARS-CoV-2基因序列(截至2022年1月)。尽管在负选择下的位点发生突变,并可能导致耐药病毒62在当前数据所描述的病毒适应度格局下,这些自然发生的变异可能不那么适合。然而,当更多的抗病毒药物被批准并广泛使用时,SARS-CoV-2可能会发生突变,对抗病毒治疗产生耐药性。尽管迄今为止,患者缺乏对nirmatrelvir的耐药性,但在体外传代SARS-CoV-2时,在nirmatrelvir浓度增加的情况下,产生了耐药病毒63,64.当患者出现耐药变异时,新的病毒基因序列和病毒蛋白结构可以使用本文提出的方法重新计算病毒残基的选择压力。总之,通过对病毒残基的选择压力进行分类,我们为抗病毒药物的开发/筛选提供了一个有用的工具,以评估给定病毒的进化是否会降低药物疗效。
数据可用性
作者声明,支持本研究结果的数据可在文章和补充表中获得S1文件。
参考文献
Carvalho, T, Krammer, F. & Iwasaki, A. COVID-19的前12个月:免疫学见解的时间表。Nat. Rev. Immunol。21, 245-256(2021)。
Gordon, d.e., Jang, g.m.和Bouhaddou, m.e.a. SARS-CoV-2蛋白相互作用图揭示药物再利用的靶点。自然583, 459 - 468。https://doi.org/10.1038/s41586-020-2286-9(2020)。
白格尔,j.h.。et al。瑞德西韦治疗covid -19最终报告。心血管病。j .地中海。383, 1813-1826(2020)。
Akbarzadeh-Khiavi, M., Torabi, M., Rahbarnia, L. & Safary, A. Baricitinib联合治疗:重新利用Janus激酶抑制剂治疗严重SARS-CoV-2感染的叙事回顾。感染50, 295-308(2022)。
阴,W。et al。瑞德西韦抑制SARS-CoV-2 RNA依赖RNA聚合酶的结构基础科学368, 1499-1504(2020)。
Kabinger F。et al。molnupirvir诱导SARS-CoV-2突变机制研究。Nat。结构。摩尔。杂志。28, 740 - 746。https://doi.org/10.1038/s41594-021-00651-0(2021)。
Ullrich, S., Ekanayake, K. B., Otting, G. & Nitsche, C. SARS-CoV-2变体的主要蛋白酶突变体仍然对nirmatrelvir敏感。Bioorg。地中海,化学。列托人。62, 128629(2022)。
佩奇,R. D. M.霍姆斯,E. C。分子进化:系统发育方法(Blackwell Science, 1998)。
李,问。et al。SARS-CoV-2突变对病毒传染性和抗原性的影响细胞182, 1284 - 1294。e9(2020)。
斯塔尔,t.n.et al。逃离用于治疗COVID-19的抗体的病毒突变的前瞻性图谱。科学371, 850 - 854。https://doi.org/10.1126/science.abf9302(2021)。
哈维,w。T。et al。SARS-CoV-2变体、spike突变和免疫逃逸。Nat. Rev.微生物。19, 409 - 424。https://doi.org/10.1038/s41579-021-00573-0(2021)。
克罗斯,t.j.。et al。SARS-CoV-2主要蛋白酶临床相关变异的序列表征和分子建模。生物化学59, 3741-3756(2020)。
乌格瑞尔,o.m.et al。评估fda批准的药物对野生型和突变型SARS-CoV-2解旋酶(Nsp13)的效力。Int。生物。絮凝。163, 1687-1696(2020)。
Krishnamoorthy, N. & Fakhro, K.针对SARS-CoV2主要蛋白酶的突变抗性冷点的鉴定。IUBMB生活73, 670-675(2021)。
马丁,R。et al。全球流行的SARS-CoV-2分离株和最近出现的人类和水貂变异株的RNA复制复合物的遗传守恒表明,对瑞德西韦的耐药性很小。小红伞。Res。188, 105033(2021)。
Yazdani, S。et al。SARS-CoV-2口袋组的遗传变异性。J.蛋白质组。20., 4212-4215(2021)。
西米尔,理学博士。et al。瑞德西韦耐药的体外选择表明SARS-CoV-2的进化可预测性。公共科学图书馆Pathog。17, e1009929。https://doi.org/10.1371/journal.ppat.1009929(2021)。
史蒂文斯,L. J.。et al。SARS-CoV-2依赖RNA的RNA聚合酶的突变通过不同的机制赋予了对瑞德西韦的耐药性。科学。Transl。地中海。https://doi.org/10.1126/scitranslmed.abo0718(2022)。
塞耶斯,e.w.。et al。国家生物技术信息中心数据库资源。核酸测定。50, D20-D26。https://doi.org/10.1093/nar/gkab1112(2022)。
Katoh, K. & Standley, D. M. MAFFT多序列比对软件版本7:性能和可用性的改进。摩尔。杂志。另一个星球。30., 772 - 780。https://doi.org/10.1093/molbev/mst010(2013)。
庞德,S. L.,弗罗斯特,S. D. &缪斯,S. V. HyPhy:使用系统发育的假设检验。生物信息学21, 676 - 679。https://doi.org/10.1093/bioinformatics/bti079(2005)。
Nguyen, L. T., Schmidt, H. A., von Haeseler, A. & Minh, B. Q. IQ-TREE:估算最大似然系统发育的快速有效随机算法。摩尔。杂志。另一个星球。32, 268 - 274。https://doi.org/10.1093/molbev/msu300(2015)。
科萨可夫斯基池塘,S. L.和弗罗斯特,S. D.毕竟没有那么不同:在选择中检测氨基酸位点的方法的比较。摩尔。杂志。另一个星球。22, 1208 - 1222。https://doi.org/10.1093/molbev/msi105(2005)。
Pollett, S。et al。人类冠状病毒的比较重组分析及其对SARS-CoV-2大流行的影响科学。代表。11, 17365年。https://doi.org/10.1038/s41598-021-96626-8(2021)。
VanInsberghe, D., Neish, A. S., Lowen, A. C.和Koelle, K.重组SARS-CoV-2基因组在大流行的第一年以低水平循环。病毒的另一个星球。https://doi.org/10.1093/ve/veab059(2021)。
Berman, H., Henrick, K. & Nakamura, H.宣布全球蛋白质数据库。Nat。结构。摩尔。杂志。10, 980(2003)。
丹娜,j。M。et al。SIFTS:更新的结构集成功能,分类和序列资源,使基于结构的蛋白质注释的覆盖范围增加40倍。核酸测定。47, D482-D489。https://doi.org/10.1093/nar/gky1114(2019)。
赵,Y。,Du, X。,段,Y。,,X &, Y s E。。高通量筛选确定现有药物为SARS-CoV-2 PLpro抑制剂。蛋白细胞12, 877 - 888。https://doi.org/10.1007/s13238-021-00836-9(2021)。
艾伯利,r.j.。et al。转用药物苏拉明和阿那平在体外协同抑制SARS-CoV-2 3CLpro。病毒13, 873年。https://doi.org/10.3390/v13050873(2021)。
阴,W。et al。苏拉明抑制SARS-CoV-2 RNA聚合酶的结构基础。Nat。结构。摩尔。杂志。28, 319 - 325。https://doi.org/10.1038/s41594-021-00570-0(2021)。
阿扁,O。et al。通过实验筛选SARS-CoV-2 3CLpro的结构稳定性及槲皮素抑制剂的鉴定Int。生物。絮凝。164, 1693 - 1703。https://doi.org/10.1016/j.ijbiomac.2020.07.235(2020)。
Kaul R。et al。天然黄酮类化合物对SARS-CoV-2靶点的抗病毒活性:系统综述Int。理学。22, 11069年。https://doi.org/10.3390/ijms222011069(2021)。
爱尔斯金尼,美国。et al。有前景的抗sars - cov -2药物,有效地双重靶向病毒和宿主蛋白酶。Bioorg。地中海,化学。列托人。43, 128099年。https://doi.org/10.1016/j.bmcl.2021.128099(2021)。
萨尔基扬,K。et al。临床安全锌喷射器多靶向多个保守SARS-CoV-2结构域的功能半胱氨酸。化学。科学。11, 9904-9909(2020)。
陈,T。et al。双硫仑/依布selen和瑞德西韦协同抑制SARS-CoV-2复制ACS制药。Transl。科学。4, 898-907(2021)。
Mazmanian, K., Chen, T., Sargsyan, K. & Lim, C.从半胱氨酸反应的量子衍生原理到对抗COVID-19大流行。电线第一版。摩尔。科学。12, e1607(2022)。
张晓明,王晓明,王晓明,王晓明,等。ebselen及其衍生物对SARS-CoV-2主要蛋白酶的抑制机制。Commun Nat。12, 3061年。https://doi.org/10.1038/s41467-021-23313-7(2021)。
张,L。et al。SARS-CoV-2主蛋白酶的晶体结构为改进α-酮酰胺抑制剂的设计提供了基础。科学368, 409−412(2020)。https://science.sciencemag.org/content/early/2020/03/20/science.abb3405.
辛格,E。et al。综合综述了通过各种计算方法鉴定出的针对SARS-CoV-2主要蛋白酶的有前途的抗病毒治疗候选药物。j .麝猫。Eng。Biotechnol。18, 69年。https://doi.org/10.1186/s43141-020-00085-z(2020)。
Roe, m.k, Junod, n.a., Young, a.r., Beachboard, d.c.和Stobart, c.c.在COVID-19时代靶向冠状病毒蛋白酶nsp5 (3CLpro, Mpro)的新结构和功能特征。维罗尔将军。102, 001558年。https://doi.org/10.1099/jgv.0.001558(2021)。
严伟,郑勇,曾晓霞,何斌,程伟,新冠病毒的结构生物学:开启新疗法的大门。钙信号。目标。其他。7, 26(2022)。
孟ist, H. M., Dilnessa, T. & Jin . .针对SARS-CoV-2主要蛋白酶的潜在抑制剂的结构基础。前面。化学。9, 622898年。https://doi.org/10.3389/fchem.2021.622898(2021)。
Lv, Z。et al。针对SARS-CoV-2蛋白酶开发COVID-19抗病毒药物。前面。化学。9, 819165年。https://doi.org/10.3389/fchem.2021.819165(2022)。
苏,H。et al。针对SARS-CoV-2的小分子药物发现的分子洞察。Angew。化学。Int。艾德。60, 9789-9802(2021)。
赵,Y。et al。SARS-CoV-2主要蛋白酶与蛋白酶抑制剂PF-07321332配合物的晶体结构蛋白质的细胞。https://doi.org/10.1007/s13238-021-00883-2(2021)。
Mótyán, J. A., Mahdi, M., Hoffka, G. & Tozsér, J. SARS-CoV-2主蛋白酶(Mpro)对蛋白酶抑制剂的潜在耐药性:从HIV-1蛋白酶中吸取的教训。Int。理学。23, 3507年。https://doi.org/10.3390/ijms23073507(2022)。
杜,R。et al。新型SARS-CoV-2变构抑制剂chebulic acid和punicalagin的发现。小红伞。Res。190, 105075年。https://doi.org/10.1016/j.antiviral.2021.105075(2021)。
霍夫曼,r.l.。et al。发现冠状病毒3CL蛋白酶的酮基共价抑制剂,可用于COVID-19的潜在治疗。医学。化学。63, 12725-12747(2020)。
高,X。et al。SARS-CoV-2木瓜蛋白酶的晶体结构。Acta制药。罪。Bhttps://doi.org/10.1016/j.apsb.2020.08.0(2020)。
墨守成规,W。et al。抑制剂结合SARS- CoV-2木瓜蛋白酶样蛋白酶的活性谱和晶体结构:抗covid -19药物设计的框架科学。睡觉。6, eabd4596(2020)。
Shin D。et al。木瓜蛋白酶样蛋白酶调控SARS-CoV-2病毒传播和先天性免疫。自然587, 657 - 662。https://doi.org/10.1038/s41586-020-2601-5(2020)。
Osipiuk, J。et al。SARS-CoV-2木瓜蛋白酶的结构及其与非共价抑制剂的配合物。Commun Nat。12, 743(2021)。
Barretto, N。et al。严重急性呼吸综合征冠状病毒木瓜蛋白酶具有去泛素活性。j .性研究。79, 15189-15198(2005)。
帕奇特。et al。一种分子传感器确定SARS-CoV-2木瓜蛋白酶样蛋白酶的泛素底物特异性。细胞的代表。36, 109754(2021)。
傅,Z。et al。GRL0617和SARS-CoV-2 PLpro的复杂结构揭示了抗病毒药物的发现热点。Commun Nat。12, 488年。https://doi.org/10.1038/s41467-020-20718-8(2021)。
Narayanan, A., Toner, S. A. & Jose, J.基于结构的抑制剂设计和重新利用临床药物来靶向SARS-CoV-2蛋白酶。物化学。Soc。反式。50, 151 - 165。https://doi.org/10.1042/BST20211180(2022)。
沈,Z。et al。利用结合协同作用设计用于COVID-19抗病毒治疗的SARS-CoV-2 PLpro抑制剂医学。化学。65, 2940 - 2955。https://doi.org/10.1021/acs.jmedchem.1c01307(2022)。
高,Y。et al。COVID-19病毒RNA依赖RNA聚合酶的结构。科学368, 779-782(2020)。
甘地,S。et al。免疫功能低下患者在治疗持续性SARS-CoV-2感染期间重新出现瑞德西韦耐药突变:一例报告。Commun Nat。13, 1547年。https://doi.org/10.1038/s41467-022-29104-y(2022)。
发现Taroxaz-104:首个有效的SARS-CoV-2 VOC-202012/01菌株解毒剂。J. Mol.结构。1246, 131106年。https://doi.org/10.1016/j.molstruc.2021.131106(2021)。
金,Z。et al。M的结构箴COVID-19病毒及其抑制剂的发现。自然582, 289 - 293。https://doi.org/10.1038/s41586-020-2223-y(2020)。
莫加达西,美国。et al。对临床蛋白酶抑制剂耐药的传染性SARS-CoV-2变种。bioRxiv。https://doi.org/10.1101/2022.08.07.503099(2022)。
Iketani, S。et al。SARS-CoV-2对nirmatrelvir耐药的多种途径。自然https://doi.org/10.1038/s41586-022-05514-2(2022)。
Jochmans D。et al。通过体外蛋白酶抑制剂筛选SARS-CoV-2 3CLpro蛋白的L50F、E166A和L167F取代体,获得对nirmatrelvir的耐药性。bioRxiv。https://doi.org/10.1101/2022.06.07.495116(2022)。
确认
本研究由台湾科技部(MOST-107-2113- m -001-018)和中央研究院(AS-IA-107-L03)资助。
作者信息
作者及隶属关系
贡献
方法:K.S.和K.M.,分析:K.M.和C.L.,写作-初稿(K.S.和K.M.),编辑(C.L.)和审查(所有三个作者)。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
Sargsyan, K., Mazmanian, K. & Lim, C.一种评估小分子药物潜在抗病毒耐药性和应用于SARS-CoV-2的策略。Sci代表13502(2023)。https://doi.org/10.1038/s41598-023-27649-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-023-27649-6