摘要gydF4y2Ba
缺陷干扰(DI)颗粒在病毒繁殖过程中产生,以亲本病毒的复制和包装为条件,并干扰病毒的扩张。开发DIs作为抗病毒药物引起了很大的兴趣。在这里,我们描述了SARS-CoV-2在高感染多样性下连续传代后产生的DI颗粒。已确定的主要DIs已丢失了约84%的SARS-CoV-2基因组,并能够降低亲本病毒滴度。人工合成的DI基因组变体也可以干扰感染,并可以用作有条件的基因传递载体。此外,DI基因组编码一种能够减弱病毒复制的Nsp1-10融合蛋白。这些结果确定了自然选择的缺陷病毒基因组,在亲代病毒存在时出现并稳定繁殖。gydF4y2Ba
简介gydF4y2Ba
贝塔冠状病毒是具有重大医学和经济重要性的病原体。在过去20年里,它们造成了三次重大病毒疫情:2003年SARS-CoV和2012年MERS-CoV疫情,以及目前的SARS-CoV-2大流行。冠状病毒是阳性意义的单链RNA病毒,其基因组为26-32 kb。冠状病毒基因组的前三分之二由两个大的开放阅读框(orf)组成,编码在病毒复制中起作用的非结构蛋白。病毒基因组的其余部分包括orf,通过编码结构蛋白和辅助蛋白的不连续转录过程转录成亚基因组mrna。5 '和3 '未翻译区域(utr)包含复制所必需的结构特征gydF4y2Ba1克ydF4y2Ba.gydF4y2Ba
正如其他正链RNA病毒所指出的那样,冠状病毒显示出很高的重组率。复制引起的错误,加上冠状病毒基因组之间的重组,增加了病毒库的遗传多样性,导致了新的病毒变体的出现gydF4y2Ba2gydF4y2Ba,并产生含有大量缺失的缺陷病毒基因组(DVGs)gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.有时,在辅助病毒存在的情况下,会出现能够保持复制和被包装的dvg,这些被称为缺陷干扰(DI)颗粒。自然选择的DI基因组已被鉴定为几种冠状病毒成员,包括小鼠肝炎病毒gydF4y2Ba5克ydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,传染性肠胃炎病毒gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,牛冠状病毒gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,以及传染性支气管炎病毒gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba.此外,最近设计了基于人类229E和SARS-CoV-2冠状病毒基因组的合成DIs,并证明能够在体外和体内降低病毒基因组RNA (gRNA)水平或病毒滴度gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba;可能是与亲代病毒竞争限制细胞和/或病毒资源的结果gydF4y2Ba27gydF4y2Ba.对自然产生的DIs的描述特别有见解,因为它们定义了在亲本病毒存在的情况下扩增突变基因组时施加的选择压力的后果,从而深入了解与亲本病毒稳定共繁殖所需的遗传信息。本文报道了SARS-CoV-2 DI颗粒的分离和功能表征。gydF4y2Ba
结果gydF4y2Ba
SARS-CoV-2 dvg的分离与鉴定gydF4y2Ba
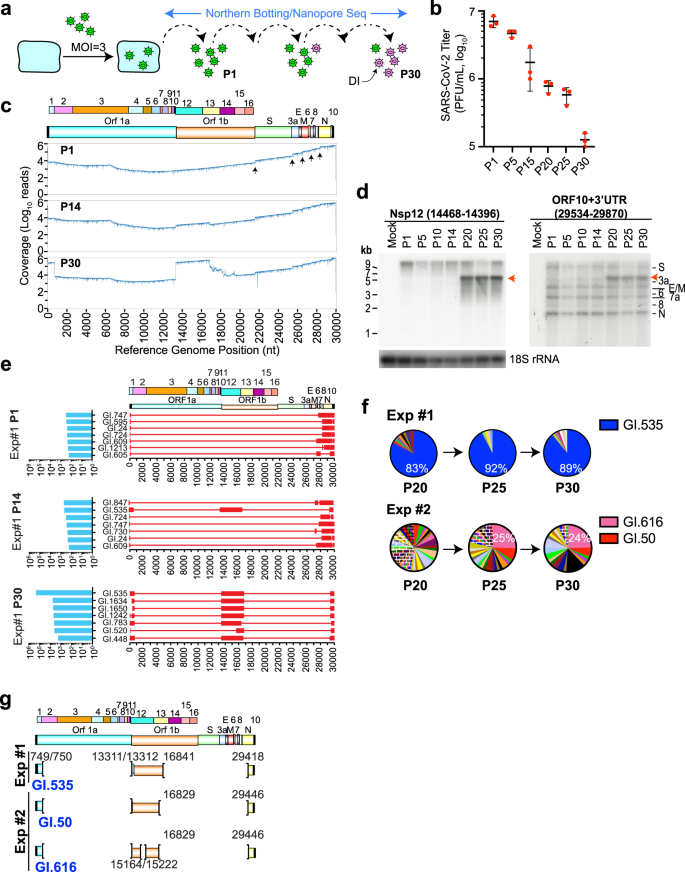
为了选择自然形成的DIs,将SARS-CoV-2以3 pfu/细胞的速度连续传代30次(图2)。gydF4y2Ba1gydF4y2Ba).传代过程中病毒滴度评估显示感染性病毒产量下降;与P1相比,P30病毒显示出55倍的减少(图3)。gydF4y2Ba1 bgydF4y2Ba).从P1-、P14-和P30感染的细胞中分离出的总RNA的直接RNA测序(DRS)显示,到P30,在连续传代过程中出现的dvg的显著比例保留了横跨Nsp10-Nsp12 (~ 13.3-16.8 kb)的基因组片段(图3)。gydF4y2Ba1 cgydF4y2Ba).从受感染细胞的不同传代中分离出的RNA的Northern blot分析发现,从P20到P30,约5 kb的DVGs保留了Nsp12和ORF10/3'UTR区域(图2)。gydF4y2Ba1 dgydF4y2Ba).由DRS信息构建的转录本模型,保留5 '和3 '端序列(因为这些区域包含重要的复制信号)gydF4y2Ba28gydF4y2Ba),表明在p1感染的细胞中SARS-CoV-2基因组损失了很大一部分(>27 kb)。gydF4y2Ba1 egydF4y2Ba).到P14时,出现了约4.7 kb的DVG (GI.535),保留核苷酸13,311/13,312-16,841。到了P30, GI.535和两个相关基因组,GI.1634和GI.1650(仅在5 '端起始位置有差异)在DVG群体中占主导地位(图3)。gydF4y2Ba1 egydF4y2Ba及补充资料gydF4y2Ba1克ydF4y2Ba).为了验证GI.535的存在,针对5 '和3 '端(A1和A2)的引物对(补充图。gydF4y2Ba1gydF4y2Ba及补充资料gydF4y2Ba2gydF4y2Ba)用于远程(LR) pcr扩增dvg(补充图。gydF4y2Ba1 bgydF4y2Ba),这些基因被克隆和测序。GI.535端口:(i) Nsp1-10帧内融合,(ii)跨越Nsp11、移码信号和Nsp12的序列,以及(iii) Nsp13和N个ORF的最后116个nts之间的帧外融合(补充图。gydF4y2Ba1gydF4y2Ba).我们追踪了GI.535中识别的上游和下游连接(USJ和DSJ)的出现,发现这些连接似乎在串行传代过程中共同出现(补充图)。gydF4y2Ba1 cgydF4y2Ba).GI.535是P20-P30中占主导地位的DVG(~83%),表明该基因组具有稳定的长期繁殖能力(图2)。gydF4y2Ba1 fgydF4y2Ba, Exp #1)。gydF4y2Ba
一个gydF4y2Ba用于分离SARS-CoV-2 DIs的策略。使用Northern blotting和纳米孔DRS来评估连续传代过程中突出的dvg的出现。亲代病毒颗粒以绿色显示,新出现的DIs以洋红色显示。gydF4y2BabgydF4y2Ba从指定的传代中获得的病毒滴度定量。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。gydF4y2BacgydF4y2BaP1、P14和P30的纳米孔RNA测序数据的基因组覆盖。“阶跃”变化(P1中向上的箭头表示)发生在S、3a、E、6和N sgrna的5′边界。参考基因组位置(nt)显示在底部。gydF4y2BadgydF4y2Ba对从sars - cov -2感染细胞的指示传代中分离的RNA进行了Northern blot分析。红色箭头突出了在通道后期出现的突出dvg。模拟未感染的细胞。探针标识显示在印迹上方。gydF4y2BaegydF4y2Ba从P1、P14和p30感染的Exp #1细胞中获得的7个最丰富的DVGs结构保留了5 '和3 '端序列。右图:DVG架构。打开的红色方框是保留的序列,细线对应删除的序列。左:转录模型对应的读取计数。顶部显示了SARS-CoV-2参考基因组,以及编码的多肽。参考基因组的核苷酸位置如下所示。gydF4y2BafgydF4y2Ba饼图显示了实验1和实验2中P20、P25和P30中DVGs的相对丰度。gydF4y2BaggydF4y2Ba从感染细胞P30分离出的最常见dvg的基因组结构。核苷酸位置基于SARS-CoV-2武汉-胡-1分离物(NC_045512.2)。gydF4y2Ba
为了确定GI.535中识别的结构特征是否可以独立地重新分离,我们在高MOI (Exp #2)下从Exp #1中使用的相同病毒原液中重复了SARS-CoV-2的连续传殖。实验2中P30发现病毒滴度降低了21倍(补充图2)。gydF4y2Ba2gydF4y2Ba).使用一系列探针(a -G)对SARS-CoV-2基因组进行Northern印迹平铺,发现在第14传代出现了约6和7 kb的显著dvg,保留了5 '近端序列(探针a检测到)、横跨Nsp12的区域(探针B)和3 '末端序列(探针G检测到)(补图)。gydF4y2Ba2 bgydF4y2Ba).到P20,我们观察到大约5 kb dvg的出现,并稳定维持了10个额外的传代(补充图。gydF4y2Ba2摄氏度gydF4y2Ba).DRS显示P15的DVG结构池与第一个实验中看到的不同(比较补充图。gydF4y2Ba3gydF4y2Ba, P15到图。gydF4y2Ba1 cgydF4y2Ba好)。在本实验中,P15处两个最突出的dvg分别是GI.464 (7.2 kb)和GI.384 (5.7 kb)。gydF4y2Ba3 bgydF4y2Ba),它们的大小与我们在P14中通过Northern blotting检测到的基因组物种相对应(补充图。gydF4y2Ba2 bgydF4y2Ba,探针B和G,分别用+和*表示)。在实验2的P29中发现的流行dvg在结构上与实验1的P30中发现的dvg相似(比较补充图。gydF4y2Ba3gydF4y2Ba(P29)到图。gydF4y2Ba1 cgydF4y2Ba(e))。P29中数量最多的前7个dvg均含有GI.535中记录的相同的Nsp1-10连接(补充图)。gydF4y2Ba3 bgydF4y2Ba).利用LR-PCR扩增P15和P29的基因组,然后进行Sanger测序,确认了GI.464、GI.384、GI.616和GI.50的结构(补充图)。gydF4y2Ba3 gydF4y2Ba及补充资料gydF4y2Ba1克ydF4y2Ba).Nsp12框架内19个氨基酸缺失存在于GI.616中,并将其与GI.50区别开来(补充图)。gydF4y2Ba4gydF4y2Ba).LR-PCR显示,大约7 kb基因组出现在P11和P16之间,大约5 kb基因组出现在P20和P30之间。gydF4y2Ba4 bgydF4y2Ba).我们评估了GI.616和GI.50在P20、P25和P30中的丰度,发现GI.616是P25和P30中存在的主要DVG(25%)。gydF4y2Ba1 fgydF4y2BaExp #2)。总的来说,这两个实验中相对适合度最高的dvg的结构(i)保留了5 '和3 '端序列,(ii)拥有相同的Nsp1-10结断点,(iii)保留了病毒移码位点Nsp11和Nsp12编码区(图2)。gydF4y2Ba1克gydF4y2Ba).gydF4y2Ba
晚期传代SARS-CoV-2 dvg的复制依赖于辅助病毒gydF4y2Ba
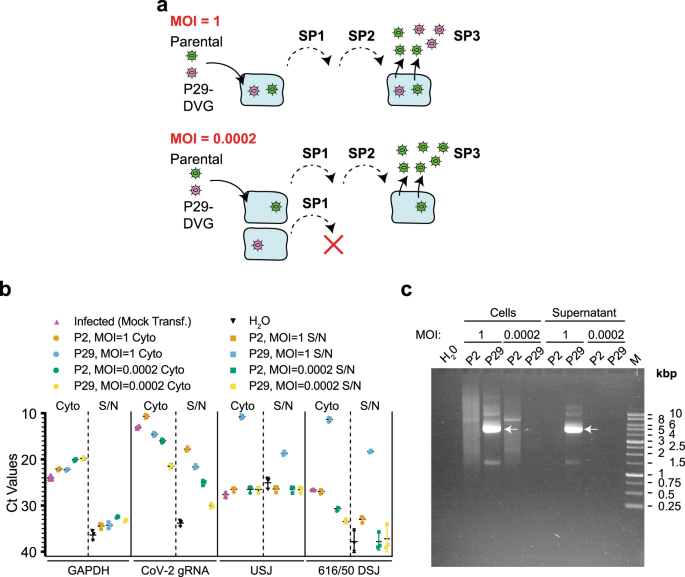
DIs的一个显著特征是它们依赖亲代病毒进行繁殖。为了确定自然选择的dvg是否也是如此,我们以MOI为1或0.0002连续用Exp #2中的P2或P29株感染Vero E6细胞。在第三次感染后,评估感染细胞和培养基的基因组含量(图。gydF4y2Ba2gydF4y2Ba).在MOI为0.0002时,来自P29的dvg更有可能进入未感染的细胞,因此,在随后的传代中应该从种群中丢失。然而,当MOI为1时,DIs更有可能进入病毒感染的细胞,并且在传代过程中应该保持(图1)。gydF4y2Ba2gydF4y2Ba).采用RT-qPCR方法分析第三个连续传代(SP3)的细胞和培养基制备的RNA (S/N)。GAPDH转录本仅在细胞RNA制剂中检测到。gydF4y2Ba2 bgydF4y2Ba).SARS-CoV-2 gRNA存在于P2或P29稀释后的细胞和培养基中。在P29感染的培养基和细胞的RNA中很容易检测到GI.616和GI.50共有的USJ和DSJ (MOI = 1)。相比之下,USJ或DSJ RT-qPCR产物在P29感染细胞的RNA中未检测到(MOI = 0.0002)(图。gydF4y2Ba2 bgydF4y2Ba).此外,P2株的感染表明,从SP3细胞裂解液或培养基中分离出的RNA中不存在含有USJ或DSJ的DVGs(图2)。gydF4y2Ba2 bgydF4y2Ba).dvg仅从感染P29 (MOI = 1)的细胞和培养基中恢复(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).在MOI = 0.0002感染细胞的提取物或培养基中未检测到DVGs。因此,突出的dvg只在辅助病毒存在的情况下传播,并且在串行传代后检测到它们也表明它们是打包的。因此,我们将这些dvg称为SARS-CoV-2 DI颗粒。gydF4y2Ba
一个gydF4y2Ba显示dvg依赖于亲代病毒复制和传播的示意图。在MOI = 1时,亲本基因组和DI基因组都有望在序列传代中保持。在MOI较低(0.0002)时,亲代基因组和DI进入不同的细胞,DI将在连续的传代中丢失。gydF4y2BabgydF4y2BaRT-qPCR分析感染指示病毒传代和MOI的细胞或上清中分离的RNA。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。gydF4y2BacgydF4y2Ba在MOI为1或0.0002时,从Vero细胞裂解液和已被指示病毒储存感染的培养基中分离DIs。使用A1和A2引物扩增30个周期。在0.8%琼脂糖/TAE凝胶上分析产物。白色箭头表示恢复了5 kb dvg。M;1 kb DNA阶梯。gydF4y2Ba
人工合成的重组DI基因组具有长期稳定性,并能减弱SARS-CoV-2的复制gydF4y2Ba
为了正式证明我们分离的DI基因组负责减弱SARS-CoV-2的复制,我们将GI.50和GI.616对应的序列置于T7 RNA聚合酶启动子的控制下,并添加一个3 ' poly(a)尾巴(图7)。gydF4y2Ba3gydF4y2Ba).在体外合成DI rna和RLuc mRNA(阴性对照)。Vero E6细胞感染SARS-CoV-2 8小时后,将RLuc、GI.50和GI.616 RNA转染到细胞中。22小时后,收集病毒并连续传代4次(图。gydF4y2Ba3gydF4y2Ba).斑块分析显示,感染后接受重组DI RNA的细胞中病毒滴度降低了10 - 20倍,而接受RLuc mRNA的细胞中病毒滴度没有明显降低(图2)。gydF4y2Ba3 bgydF4y2Ba).通过LR-PCR从sp4感染细胞的RNA中恢复了完整的合成DI基因组(图2)。gydF4y2Ba3 cgydF4y2Ba).用α-Nsp1抗体探测细胞裂解液,发现病毒感染细胞中存在Nsp1。gydF4y2Ba3 dgydF4y2Ba从GI.616转染细胞中接受病毒的细胞也表达了一种免疫反应蛋白,其分子质量与Nsp1-10融合产物一致(图2-4与1通道比较)。gydF4y2Ba3 cgydF4y2Ba通过RT-qPCR分析来自P0、SP2和SP4感染的RNA是否存在SARS-CoV-2 gRNA和DI基因组(图2)。gydF4y2Ba3 egydF4y2Ba[原始Ct值]和补充图。gydF4y2Ba5gydF4y2Ba[数据归一化为GAPDH mRNA水平,并相对于CoV-2 gRNA水平表达])。正如预期的那样,RLuc mRNA在P0转染的细胞中存在,但在SP2-或sp4感染的细胞中不存在。gydF4y2Ba3 egydF4y2Ba和补充图。gydF4y2Ba5gydF4y2Ba).所有感染细胞均可检测到SARS-CoV-2 gRNA。USJ和DSJ是DI基因组的独特特征,存在于转染的(P0)细胞以及SP2-和sp4感染的细胞中(图2)。gydF4y2Ba3 egydF4y2Ba).GAPDH mRNA和18 S rRNA水平在样品中相似(补充图。gydF4y2Ba5 bgydF4y2Ba).在没有SARS-CoV-2病毒的情况下,GI.50和GI.616基因组在SP2样本中不存在,这与DI复制依赖于亲本病毒一致(补充图)。gydF4y2Ba6gydF4y2Ba).这些结果表明,合成的重组DI RNA可以在亲本SARS-CoV-2存在的情况下有条件地稳定繁殖,并在感染后削弱病毒复制。gydF4y2Ba
一个gydF4y2Ba用于合成DI颗粒的实验流。将Vero E6细胞在MOI = 1时感染SARS-CoV-2后,转染体外合成的RLuc、GI.50或GI.616 RNA。22小时后收集培养基,澄清,用于Vero E6细胞连续感染(4传代)。gydF4y2BabgydF4y2Ba从指定的DIs中获得的病毒滴度在SP4的定量。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。ns无意义的,gydF4y2BapgydF4y2Ba> 0.9(双向方差分析)。gydF4y2BacgydF4y2BaRT/LR-PCR显示从sp4感染细胞中恢复了DI基因组。gydF4y2BadgydF4y2BaWestern blot检测提取物α-Nsp1或α-actin抗体。分析的裂解物来自未感染的(模拟)Vero E6细胞(通道1)或接受未转染细胞(−)(通道2)、RLuc mrna转染细胞(通道3)或GI.616 rna转染细胞(通道4)SP2的Vero细胞。虚线箭头表示Nsp1,红色箭头表示Nsp1 - 10融合。gydF4y2BaegydF4y2BaRT-qPCR分析P0、SP2-和sp4感染细胞的RNA。每个寡核苷酸对靶向的rna显示在底部。显示获取的Ct值。gydF4y2BangydF4y2Ba= 2个生物独立实验,黑柱表示均值。gydF4y2BafgydF4y2Ba含有EMCV/RLuc或TRS/RLuc表达盒的DI基因组示意图。gydF4y2BaggydF4y2BaRLuc活性在SP4得到。gydF4y2BangydF4y2Ba= 4个生物独立实验±SD。gydF4y2BahgydF4y2Ba从指定的DIs中获得的病毒滴度在SP4的定量。gydF4y2BangydF4y2Ba= 4个生物独立实验±SD。gydF4y2Ba我gydF4y2BaRT-PCR显示,在转染GI.616-TRS/RLuc的sars - cov -2感染细胞中存在一个包含5 ' TRS-L位点上游序列的RLuc sgRNA。gydF4y2Ba
为了评估DIs是否可以作为有条件的基因传递载体,一个emcv驱动gydF4y2BaRenillagydF4y2Ba将荧光素酶(EMCV/RLuc)或转录调控序列(TRS/RLuc)盒式报告基因插入GI.616的DSJ(图。gydF4y2Ba3 fgydF4y2Ba).用SARS-CoV-2感染Vero E6细胞,进行1 hpi DI RNA转染。接着是四篇连载文章。在接受RLuc转染的SP4病毒的细胞中,仅检测到背景水平的荧光素酶活性(图2)。gydF4y2Ba3 ggydF4y2Ba).相反,转染GI.616-EMCV/RLuc的SP4病毒感染的细胞产生显著的荧光素酶活性(图2)。gydF4y2Ba3 ggydF4y2Ba).然而,GI.616-TRS/RLuc样品的荧光素酶水平最高,比含有GI.616-EMCV/RLuc DIs的细胞高320倍(图2)。gydF4y2Ba3 ggydF4y2Ba).重组GI.616-EMCV/RLuc或GI.616-TRS/RLuc基因组的存在,分别使SARS-CoV-2滴度降低了15倍和30倍。gydF4y2Ba3 hgydF4y2Ba).我们使用针对TRS-L和renilla ORF的引物,通过RT-PCR证实GI.616-TRS/RLuc产生了包含病毒5 ' TRS-L末端序列的亚基因组mRNA(图2)。gydF4y2Ba3我gydF4y2Ba综上所述,这些结果表明,本文鉴定的DI基因组的合成版本可以用作抑制SARS-CoV-2复制的条件基因传递载体。gydF4y2Ba
重组DI基因组干扰SARS-CoV-2复制gydF4y2Ba
接下来,我们试图查询GI.616限制病毒复制的机制。在感染和转染Vero E6细胞后,病毒连续传代三次,并在SP2和SP3时测定培养基和细胞中的病毒RNA水平(图。gydF4y2Ba4gydF4y2Ba).在p2感染的细胞中,与RLuc对照相比,GI.616降低了SARS-CoV-2 gRNA水平(图2)。gydF4y2Ba4 bgydF4y2Ba).然后比较p2感染细胞中SARS-CoV-2 gRNA相对于SP3培养基中存在的病毒粒子的水平。GI.616的存在并不影响SARS-CoV-2 gRNA从细胞中包装或释放(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).此外,GI.616被包装并以与SARS-CoV-2 gRNA相同的效率释放(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).GI.616的存在不影响SARS-CoV-2 gRNA的传播(图。gydF4y2Ba4 dgydF4y2Ba).然而,GI.616基因组的传播速度是SARS-CoV-2 gRNA的四倍(图2)。gydF4y2Ba4 dgydF4y2Ba).综上所述,这些结果表明,在感染早期(4小时)GI.616的强劲复制与感染过程中SARS-CoV-2 gRNA水平的降低有关。gydF4y2Ba
一个gydF4y2Ba评估GI.616对SARS-CoV-2复制影响的实验设计示意图。从P2、P3细胞及SP3上清中分离出SARS-CoV-2和GI.616基因组。gydF4y2BabgydF4y2Ba在GI.616(+)或RLuc(−)存在下繁殖的亲代病毒的生长速率(相对于4小时数量的绝对gRNA水平)。给出了四个独立实验的数据。gydF4y2BacgydF4y2Ba在GI.616存在或不存在的情况下,SARS-CoV-2繁殖时封装基因组的百分比。gydF4y2BangydF4y2Ba= 4个独立的生物学实验±SD。Ns,不显著(gydF4y2BapgydF4y2Ba= 0.25)。gydF4y2BadgydF4y2BaSARS-CoV-2和GI.616的传播效率。gydF4y2BangydF4y2Ba= 4个独立生物实验±SD。gydF4y2Ba
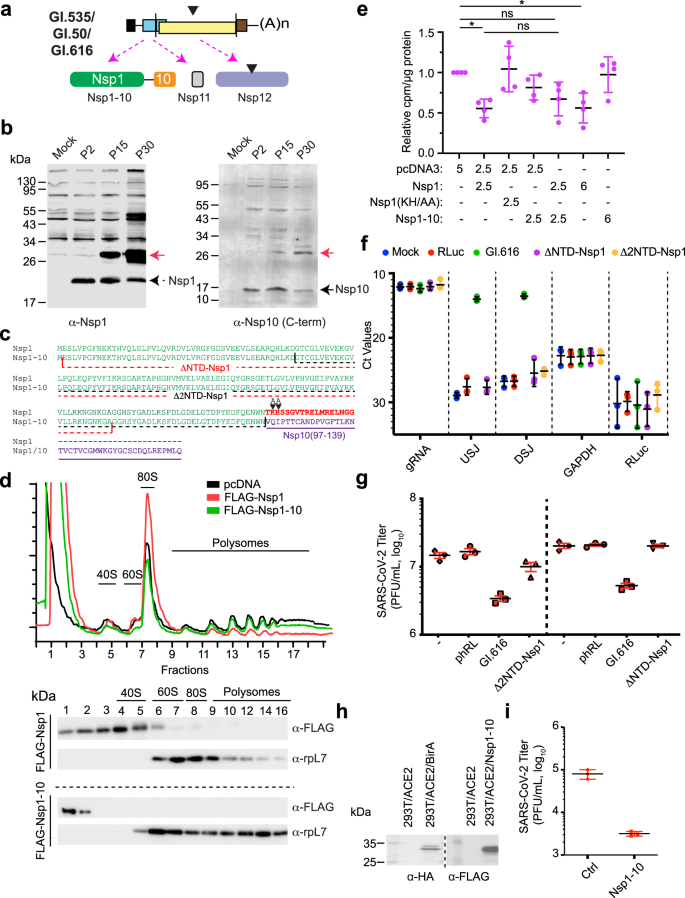
SARS-CoV-2 DIs编码Nsp1-10融合,抑制病毒复制gydF4y2Ba
SARS-CoV-2 Nsp1是一种多功能蛋白,涉及阻断宿主翻译、细胞mRNA降解和抑制核细胞质mRNA输出gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba.为了确定Nsp1-10是否能在感染细胞中检测到(图;gydF4y2Ba5gydF4y2Ba),对P2、P15或P30株感染的Vero E6细胞提取物进行Western blot。这些实验结果表明,Nsp1 (~20 kDa)存在于感染细胞中(图2)。gydF4y2Ba5 bgydF4y2Ba(左侧面板黑色箭头),而在P15-和p30感染的细胞(红色箭头)中存在一种更大的~30 kDa蛋白,与Nsp1抗体交叉反应。这种蛋白也可以使用针对Nsp10的c端结构域的抗体检测到,该抗体显示了约30 kDa的免疫反应蛋白(图。gydF4y2Ba3 bgydF4y2Ba,右面板红色箭头)。由突出的DIs编码的Nsp1-10融合蛋白在未感染细胞中过表达时主要局限于细胞质(补充图)。gydF4y2Ba7一个gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba黑色三角形表示GI.616中19个氨基酸缺失。gydF4y2BabgydF4y2Ba用α-Nsp1(左)或α-Nsp10 c端结构域抗体(右)检测提取物的Western blot。分析的裂解物来自未感染的(模拟)Vero E6细胞或感染P2、P15和P30 (Exp #2)病毒的Vero E6细胞。虚线箭头为Nsp1,填充箭头为Nsp10,红色箭头为Nsp1 - 10融合。gydF4y2BacgydF4y2BaNsp1和Nsp1 - 10融合的星团欧米茄对齐。红色表示的氨基酸对应于对翻译抑制至关重要的Nsp1 c端区域。紫色表示的氨基酸来自Nsp10。虚线表示两个Nsp1缺失突变体的程度。显示了突变为AA的Nsp1 KH氨基酸的位置。gydF4y2BadgydF4y2Ba用表达载体(20µg)转染的293个T细胞的多体分析。转染24 h后制备细胞质提取物,用蔗糖梯度沉淀法分析多体。然后对从单个多聚体部分获得的蛋白质样本进行Western blot分析。Western blot检测的抗体显示在右侧。gydF4y2BaegydF4y2BaNsp1-10的异位表达不抑制293 T细胞的翻译。293个T细胞转染一定量的表达载体。24小时后,细胞被代谢标记gydF4y2Ba35gydF4y2BaS-Met/Cys 15分钟。使用TCA沉淀来确定纳入蛋白质的放射性标签量,计数归一化为提取物中的总蛋白质含量,并相对于接受空载体(pcDNA3;设置为1)。gydF4y2BangydF4y2Ba= 4个生物独立实验±SD。ns,gydF4y2BapgydF4y2Ba> 0.05;*, 0.01 >gydF4y2BapgydF4y2Ba> 0.05 (Dunnett多重比较检验)。gydF4y2BafgydF4y2BaRT-qPCR分析P2细胞被指定DIs感染的RNA。每个寡核苷酸对靶向的RNA制剂显示在底部。显示获取的Ct值。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。gydF4y2BaggydF4y2Ba在P4用指定DIs获得的病毒滴度定量。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。gydF4y2BahgydF4y2BaWestern blot检测稳定表达BirA (Ctrl, control)或Nsp1-10的293 T/ACE2细胞。gydF4y2Ba我gydF4y2Ba293 T/ACE2/BirA或293 T/ACE2/ Nsp1-10细胞中获得的SARS-CoV-2病毒滴度。gydF4y2BangydF4y2Ba= 3个生物独立实验±SD。gydF4y2Ba
Nsp1 c端结构域对于阻断翻译至关重要,因为它与mRNA入口通道相互作用以抑制细胞蛋白质合成gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba.然而,这个结构域在Nsp1-10融合蛋白中是不存在的。gydF4y2Ba5度gydF4y2Ba).因此,Nsp1,而不是Nsp1 - 10,通过多体分析评估,抑制细胞翻译(图。gydF4y2Ba5 dgydF4y2Ba).与Nsp1不同,Nsp1 - 10不以多体梯度与40s核糖体共迁移(图2)。gydF4y2Ba5 dgydF4y2Ba).这些数据与我们之前所描述的不阻断转译的Nsp1(KH/AA)突变体的观察结果一致(补充图)。gydF4y2Ba7 bgydF4y2Ba)gydF4y2Ba31gydF4y2Ba.Nsp1,但Nsp1 - 10和Nsp1(KH/AA)不受抑制gydF4y2Ba35gydF4y2BaS-Met/Cys掺入新生多肽链(图。gydF4y2Ba5 egydF4y2Ba,补充图。gydF4y2Ba7 cgydF4y2Ba).Nsp1-10无法挽救nsp1介导的细胞翻译抑制(图。gydF4y2Ba5 egydF4y2Ba)或体外培养(补充图。gydF4y2Ba7 d, egydF4y2Ba).gydF4y2Ba
为了评估Nsp1-10区域对DI复制的需求,我们在GI.616中生成了两个缺失突变体ΔNTD-Nsp1和Δ2NTD-Nsp1。gydF4y2Ba5度gydF4y2Ba).在与亲本病毒共传代两代后,突变DIs的繁殖受到影响(图2)。gydF4y2Ba5 fgydF4y2Ba[原始Ct值]和补充图。gydF4y2Ba8gydF4y2Ba[数据归一化到GAPDH mRNA水平,并相对于CoV-2 gRNA水平表达])和亲本病毒滴度没有显著影响(图。gydF4y2Ba5克gydF4y2Ba).在293 T/ACE2细胞中异位表达Nsp1-10可使SARS-CoV-2滴度降低25倍(图2)。gydF4y2Ba5 h,我gydF4y2Ba).Nsp1-10的作用是选择性的,因为它没有降低登革2型病毒的滴度(补充图。gydF4y2Ba9gydF4y2Ba).虽然我们的数据不能让我们得出关于Nsp1-10在DI复制中的作用的确切结论,但它确实支持这样的结论,即Nsp1-10是一种有效的SARS-CoV-2复制抑制剂。gydF4y2Ba
GI.616编码的Nsp12 (Δ19aa)对聚合酶活性无活性gydF4y2Ba
最后,我们研究了GI.616中Nsp12 (Δ19aa)突变可能对Nsp12活性的影响。根据Nsp12的结构,缺失19个氨基酸有望缩短该蛋白的指区和掌区之间的距离,改变RNA结合(补充图。gydF4y2Ba10gydF4y2Ba).使用聚合酶延伸试验,其中使用4-mer引物评估活性,我们监测14-nts产物的外观(补充图)。gydF4y2Ba10 bgydF4y2Ba之前描述的Nsp12 (SNN)活性位点突变体在本试验中不活跃(比较13-18和1车道)gydF4y2Ba36gydF4y2Ba.同样,Nsp12(Δ19aa)突变体也降低了聚合酶活性(比较19-24车道和1车道)。在含有WT RdRP的反应中,Nsp12(SNN)(2-6车道)或Nsp12(Δ19aa)(8-12车道)的存在不会降低RdRP活性,证明缺乏主导-阴性行为。gydF4y2Ba
讨论gydF4y2Ba
目前正在探索将DI颗粒用于几种重要的人类病原体(包括甲型流感病毒)的抗病毒治疗gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,寨卡病毒gydF4y2Ba40gydF4y2Ba以及基孔肯雅病毒gydF4y2Ba41gydF4y2Ba.虽然已经从冠状病毒中分离出了几种自然产生的DIs(见导言),但我们的研究记录了SARS-CoV-2中自然产生的DIs的特征。冠状病毒DIs中存在的一个显著特征是基因组5 '和3 '端序列的保留——这一发现与这些位置的茎环对复制至关重要一致gydF4y2Ba1克ydF4y2Ba.一个常见的,保守的上游连接元件,融合Nsp1到Nsp10存在于80-90%的晚期传代dvg中。由于在实验#1和#2中使用了相同的病毒种子库,我们不能正式排除在初始种子库中存在含有Nsp1-10的缺陷基因组,尽管在P1中RT-qPCR没有检测到它(补充图2)gydF4y2Ba1 cgydF4y2Ba).尽管如此,无论是出现在P0还是在传播过程中产生,它在两个独立的实例(Exp #1和#2)的串行传递中显然是被积极选择的。gydF4y2Ba
Gribble等人。gydF4y2Ba4gydF4y2Ba绘制了SARS-CoV-2基因组重组的模式和频率,并报告>50%的重组频率发生在26个位置,其中13个位点映射到TRS位置。值得注意的是,这些位点都没有映射到我们所识别的Nsp1-10连接上。我们无法发现Nsp1和Nsp10之间的主要序列相似性/互补性,这可以很容易地解释Nsp1 - 10融合的出现是不连续转录或重组的结果。驱动这种结融合形成的机制细节有待更好地理解蛋白质- rna相互作用组和在冠状病毒复制期间驱动重组的元素。gydF4y2Ba
虽然Nsp1-10融合出现在干扰素缺乏的Vero细胞中,但其功能并不局限于此,因为Nsp1-10在表达ace2的293 T细胞中的表达减弱了病毒复制(图2)。gydF4y2Ba5我gydF4y2Ba).因此,Nsp1-10蛋白似乎代表了一种di编码蛋白,可以减弱辅助病毒的复制。与Nsp1不同的是,Nsp1 - 10不干扰细胞翻译。gydF4y2Ba5 d, egydF4y2Ba).Nsp1是一种多功能蛋白,也与宿主mRNA的切割和mRNA输出的阻断有关gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba目前的研究旨在评估这些过程是否受到Nsp1-10的影响。gydF4y2Ba
目前已有两篇合成设计SARS-CoV-2 DIs的报道。gydF4y2Ba25gydF4y2Ba合成了RNA DIgydF4y2Ba1克ydF4y2Ba其中,有789个5 '端序列融合到Nsp14 (nt 19,674), Nsp15融合到3 ' UTR的最后1426个nt。核苷酸19,674-20,340被纳入DI设计gydF4y2Ba1克ydF4y2Ba因为基因组的这个区域被认为是包装信号的港湾gydF4y2Ba44gydF4y2Ba.迪gydF4y2Ba1克ydF4y2Ba在一次病毒传代后能够与SARS-CoV-2共同繁殖gydF4y2Ba25gydF4y2Ba.多次注射注入gydF4y2Ba1克ydF4y2Ba没有报道,所以我们不知道这个基因组是否稳定,可以长期繁殖。我们的结果清楚地表明19,674-20,340核苷酸不是包装所必需的,因为我们的主要DIs都没有这个区域。查图维迪等人。gydF4y2Ba26gydF4y2Ba报道了第二组合成DIs,他们称之为治疗性干扰粒子1和2 (TIP1和TIP2)。TIP1只包含5 '端(1-450)和3 '端(361个核苷酸),而TIP2从病毒5 '端(约1.5 kb)融合到3 '端(约713个核苷酸)。TIP1和TIP2在连接断点处都嵌入了EMCV IRES/mCherry。携带TIP1的颗粒可以从被TIP1 RNA核感染并随后被SARS-CoV-2感染的Vero细胞上清液中回收,证明了TIP1在辅助病毒存在下被包装的能力。TIP1和TIP2的合成基因组能够在体内和体外降低病毒滴度gydF4y2Ba26gydF4y2Ba.这些结果表明Nsp1-10融合并不是DIs降低亲本病毒滴度的特有特征。值得注意的是,在我们的实验中,我们没有通过Northern blotting检测到长度<5 kbp的显著稳定缺陷病毒基因组(图2)。gydF4y2Ba1 dgydF4y2Ba和补充图。gydF4y2Ba2 b, cgydF4y2Ba)也不是两个实验后期的纳米孔测序数据中普遍存在的转录模型(图。gydF4y2Ba1 egydF4y2Ba和补充图。gydF4y2Ba3 bgydF4y2Ba).这些结果表明,在亲代病毒存在的情况下,我们所描述的约5 kb基因组能够稳定地长期繁殖。gydF4y2Ba
目前尚不清楚为什么大多数DI基因组在传代后期保留了横跨Nsp10-Nsp13的序列。由于GI.616编码缺乏聚合酶活性的Nsp12突变体,因此功能性Nsp12蛋白对于DI繁殖不是必需的。gydF4y2Ba10gydF4y2Ba),但在实验#2 P30中DI含量最高(补充图。gydF4y2Ba3 bgydF4y2Ba).是否最近发现了移码位点周围的远程RNA相互作用gydF4y2Ba45gydF4y2Ba是平衡翻译和复制事件所必需的,这仍有待研究。gydF4y2Ba
先前的研究表明,有缺陷的干扰粒子可以导致病毒滴度的周期性变化,因为它们不仅与亲本病毒竞争,而且依赖于它们进行繁殖。在DI存在的情况下,亲本病毒水平将下降并达到局部最小值,DI水平随后也会如此,因为它们依赖于亲本病毒进行复制。这反过来又导致亲代病毒水平达到峰值,因为可供竞争的DI颗粒最小。随着传代数的增加,这导致亲代病毒水平和DI水平的周期性变化,其中DI水平的峰值与亲代病毒水平的低谷重叠,反之亦然gydF4y2Ba14gydF4y2Ba,gydF4y2Ba46gydF4y2Ba对野生型脊髓灰质炎病毒与脊髓灰质炎DI基因组共复制之间动力学的深入分析表明,DI基因组复制和包封是影响wt病毒结果的两个最关键参数gydF4y2Ba27gydF4y2Ba.考虑到控制这些事件用于临床应用的明显复杂性,更可靠的治疗策略可能是将编码DI颗粒的RNA经鼻内递送,事实上,这已被用作在小鼠模型中抑制SARS-CoV-2感染的成功策略gydF4y2Ba26gydF4y2Ba,gydF4y2Ba47gydF4y2Ba.gydF4y2Ba
我们的研究的一个局限性是,所描述的DIs是从干扰素缺乏的Vero E6细胞中分离出来的,在产生干扰素的细胞系中扩展这些研究将是重要的。我们的结果值得在临床前小鼠模型中进一步扩展和验证,以评估本文披露的合成基因组或Nsp1-10融合蛋白是否能发挥预防或治疗的作用。gydF4y2Ba
方法gydF4y2Ba
产生有缺陷的干扰粒子gydF4y2Ba
Vero E6细胞(10gydF4y2Ba6gydF4y2Ba在6孔板中培养的细胞/孔)在MOI为3的条件下感染SARS-CoV-2 60 min。病毒被清除,用2毫升新鲜的完全DMEM培养基替换。每天监测细胞,在细胞致病效应(CPE)出现24 - 48小时后收集病毒上清。将含有病毒1传代(P1)的上清液的50%用于感染新的一批Vero E6细胞,剩余的P1病毒在−80°C下冷冻成两个等体积的等份。感染1小时后,将病毒去除,用2 ml新鲜的完全DMEM培养基替换。每天监测细胞,CPE出现24 - 48小时后收集上清液。这是病毒的第二代(P2)。重复同样的过程,直到到达第30段(P30)。提取每个传代的总细胞RNA,并在- 80°C保存以备将来使用。实验1和实验2均使用相同的病毒种料。 The Institutional Biosafety Committee of the University of Alberta approved the protocol used in these studies.
斑块化验gydF4y2Ba
Vero E6细胞镀于24孔板(10gydF4y2Ba5克ydF4y2Ba细胞/孔),37°C孵育过夜。连续稀释含病毒培养基(10gydF4y2Ba−1gydF4y2Ba-10年gydF4y2Ba−6gydF4y2Ba)用DMEM介质制成96孔板。在24孔板的Vero E6细胞中各取100 μL,在5% CO中37℃孵育gydF4y2Ba2gydF4y2Ba1小时,每15分钟摇晃一次。孵育1小时后,除去含病毒的培养基,每孔加入1ml预热的空斑培养基(含2%胎牛血清和0.75%甲基纤维素的MEM培养基)。培养皿在37℃、5% CO中孵育gydF4y2Ba2gydF4y2Ba3天让斑块形成。第3天,甲基纤维素覆盖物被轻轻去除,细胞通过在每个孔中加入1ml 4%多聚甲醛PBS固定。室温孵育30 min后,取出固定液,用dH冲洗gydF4y2Ba2gydF4y2BaO,每孔加1ml 1% (w/v)结晶紫,20%甲醇。30分钟后取出结晶紫溶液,用水清洗盘子。仅在有5-30个斑块的孔中计数。gydF4y2Ba
免疫印迹(Nsp1-10融合)gydF4y2Ba
感染SARS-CoV-2传代P2、P15和P30的Vero E6细胞在感染后24小时收获。细胞用PBS洗涤三次,然后用含有新鲜蛋白酶抑制剂和1单位苯并酶(Millipore;Burlington, MA, USA)。蛋白质在95°C下变性10分钟,SDS-PAGE分离,转移到聚偏二氟乙烯(PVDF)膜上进行免疫印迹。使用Odyssey®CLx成像系统(LI-COR Biosciences;林肯,东北,美国)。gydF4y2Ba
Renilla荧光素酶测定gydF4y2Ba
Vero E6细胞(10gydF4y2Ba5克ydF4y2Ba以MOI = 1感染SARS-CoV-2 1 h后,分别用体外合成的RLuc、GI.616-EMC/RLuc和GI.616-TRS/RLuc RNA转染24孔板。在感染后24小时收集上清,澄清,50%的病毒用于Vero E6细胞的连续感染。每传代感染24 h后,取出培养基,PBS洗涤细胞,100 μL renilla luciferase assay lysis buffer (Promega, #E2810)室温裂解15 min。收集裂解液并在- 80°C保存,直到进一步使用。荧光素酶测定,每传代取20 μL加入白色96孔微孔板(Greiner bio-one),一式两份gydF4y2BaRenillagydF4y2Ba荧光素酶活性测定。每孔加入100微升renilla荧光素酶测定试剂(Promega, #E2810),并立即使用Synergy HTX平板阅读器(Biotek;Winooski, VT, USA)。除了在连续感染中使用的病毒上清外,收集剩余的病毒进行滴定。此外,还提取来自每个传代细胞的总细胞RNA,并在−80°C下存储以备将来使用。gydF4y2Ba
RNA转染gydF4y2Ba
Vero E6细胞在DMEM中以2 × 10密度添加10% FBS (Gibco, #12483-020)、1%青霉素-链霉素(Wisent, # 450-200-EL)和1×非必需氨基酸(Wisent, # 321-011-EL)的24孔板中培养gydF4y2Ba5克ydF4y2Ba细胞/。第二天,介质改为200 uL Opti-MEM (Gibco, 31985070)。用Lipofectamine™3000 (Invitrogen, #L3000015)转染细胞,并按照制造商推荐的方法制备转染混合物。基本上,将500 ng cap-1 mRNA添加到模拟或sars - cov -2感染细胞的每孔中,并在下游处理前孵育22小时。gydF4y2Ba
质粒构建gydF4y2Ba
本研究中使用的所有质粒序列在补充数据中提供gydF4y2Ba3 gydF4y2Ba.gydF4y2Ba
有帽mrna的体外合成gydF4y2Ba
携带DI基因组的质粒用BsmBI或Esp3I线性化。用苯酚-氯仿萃取纯化DNA,用HgydF4y2Ba2gydF4y2BaO,通过1 mL Sephadex™g -50超细珠柱(GE Healthcare, #17-0041-01),用100%乙醇沉淀,用70%乙醇洗涤,并以1µg/µL的浓度在RNase-free水中重新悬浮。使用线性化质粒作为模板,根据制造商的说明,使用T7 RiboMAX™Express Large Scale RNA生产系统(Promega, P1320)在体外合成mRNA。然后使用牛痘封盖系统(NEB, # M2080S)和mRNA Cap 2′- o -甲基转移酶(NEB, #M0366S)一步反应封盖RNA。RNA清除是通过上文所述的苯酚-氯仿萃取来清除线性化的DNA。浓度由NanoDrop 1000 (Thermo Scientific)定量测量。Cap 1 mRNA与ssRNA阶梯(NEB, # N0362S)一起在1%琼脂糖-甲醛变性凝胶上进行分析,以确认大小和质量。gydF4y2Ba
远程-qPCRs (LR)gydF4y2Ba
为了制备lr - pcr的全长cDNA,根据制造商的说明,使用Superscript™IV逆转录酶(Invitrogen, #18090010)和针对SARS-CoV-2的3 ' UTR的基因特异性引物:(5'CTCCTAAGAAGCTATTAAAATCAC3 ')对总RNA进行逆转录。通过RNase H (NEB, #M0297S)处理获得单链DNA,将得到的cDNA稀释10倍。采用LA-Taq DNA聚合酶热启动版本(TaKaRa, #RR042B)和2µL稀释cDNA作为模板进行lr - pcr。循环条件按照制造商的建议执行。引物在补充数据中说明gydF4y2Ba2gydF4y2Ba.以LR-PCR产物为模板,生成更小的产物,直接进行Sanger测序。gydF4y2Ba
RT-qPCRsgydF4y2Ba
互补DNA由M-MuLV逆转录酶(NEB, M0253L)或SuperscriptTM IV VILO™Mastermix (ThermoFisher, #11756050)使用随机六聚物引物生成。cDNA被稀释10倍,并使用SsoFast™Evagreen®Supermix (Bio-Rad, #1725201)作为qPCR模板。循环条件包括初始变性95°C/30秒,98°C/5秒,60°C/5秒(40次循环),65°C到95°C以0.5°C/min的速度递增。用于熔化曲线采集。使用的引物对在补充数据中列出gydF4y2Ba2gydF4y2Ba.gydF4y2Ba
确定病毒传代中dvg的相对丰度gydF4y2Ba
使用Superscript™IV逆转录酶(Invitrogen, #18090010)根据制造商的协议对实验#1和#2中P20、P25和P30的细胞RNA进行逆转录。得到的cDNA用RNase H (NEB, #M0297S)处理并稀释10倍。以稀释后的cDNA为模板,使用A1/A2引物对和LA-Taq DNA聚合酶热启动版本(TaKaRa, #RR042A)进行LR-PCR。主要长程产品经凝胶纯化,并根据制造商说明,使用pGEM®-T Easy Vector System (Promega, # A1360)通过TA克隆到pGEM-T Easy中,或使用EcoRV通过钝器连接到pBluescript II KS(+)中。用碱性裂解法对每个克隆进行微制备,获得质粒DNA,然后进行Sanger测序。gydF4y2Ba
Northern blot分析gydF4y2Ba
对于所有的Northern印迹,总RNA使用NanoDrop 1000 (Thermo Scientific)进行量化。每个样本的RNA在1.2%的甲醛-琼脂糖凝胶上电泳。电泳后,切除RNA梯道,用SYBR™金核酸凝胶染色(Invitrogen)进行染色。用10× SSC在Hybond N +膜上进行Northern印迹转移。转移后,膜以1.2 × 10进行uv交联gydF4y2Ba5克ydF4y2BaµJ /厘米gydF4y2Ba2gydF4y2Ba.用杂交缓冲液(50%甲酰胺、10%硫酸右旋糖酐、0.8 M NaCl、5× Denhardt’s溶液、50 mM Tris 7.5、0.1%热磷酸钠、100µg/mL鲑鱼精子DNA、0.5% SDS)在42℃下预杂交16 h,然后用放射性探针在42℃下杂交16 h。分别用0.1% SDS/2× SSC、0.1% SDS/1× SSC、0.1% SDS/0.5× SSC在65℃下洗涤2次,每次25 min。通过将膜暴露于x射线胶片(BioMax XAR, Kodak)来获得放射自显影。gydF4y2Ba
确定SARS-CoV-2和dip的生长速度、包装效率和传播效率gydF4y2Ba
以MOI = 1感染Vero E6细胞,感染8 h后转染DI mRNA。转染22小时后,所产生的上清连续传代3次,每24小时1次。在P2感染后的指定时间点(4、8和24小时)和P3感染后4小时提取细胞RNA。P2上清24 h提取RNA。RT-qPCR后,用2gydF4y2Ba−ΔCtgydF4y2Ba方法,以GAPDH为对照。结果值归一化到4小时时间点。gydF4y2Ba
为了计算基因组包装百分比和传播效率,采用RT-qPCR方法,根据重组RNA标准建立的标准曲线确定DI和SARS-CoV-2 RNA拷贝数。将DI或WT病毒mRNA打包百分比计算为:100 × (P2上清液mRNA拷贝数)/(P2上清液mRNA拷贝数+ P2细胞mRNA拷贝数)。gydF4y2Ba
透射率计算如下:100 × (P3细胞在4小时时的mRNA拷贝数)/(P2细胞在24小时时的mRNA拷贝数)。gydF4y2Ba
差别洗涤剂分馏gydF4y2Ba
HEK-293T细胞以10的密度在6孔板中播种gydF4y2Ba6gydF4y2Ba细胞/。在每个孔中,细胞用磷酸钙转染,使用5µg空pcDNA3.1或表达指示蛋白的pcDNA3.1。转染24小时后,在冷PBS中刮取细胞,在4°C下300 ×离心10 min制粒gydF4y2BaggydF4y2Ba.细胞在100µL洋地黄苷提取缓冲液(10 mM PIPES (pH 6.8), 300 mM蔗糖,100 mM NaCl, 3 mM MgCl中裂解gydF4y2Ba2gydF4y2Ba, 5 mM EDTA, 0.015%洋地黄苷,1 mM PMSF)冰封10分钟,裂解液在4℃下480 ×离心10分钟gydF4y2BaggydF4y2Ba.上清液保留为胞浆部分。将洋地黄苷不溶性颗粒放入相同体积的洋地黄苷提取缓冲液中洗涤一次,以480 ×的速度纺丝gydF4y2BaggydF4y2Ba10分钟。废弃上清液后,将颗粒重悬于相同体积的Triton-X-100萃取缓冲液(10 mM PIPES (pH 6.8), 300 mM蔗糖,100 mM NaCl, 3 mM MgCl中gydF4y2Ba2gydF4y2Ba, 5 mM EDTA, 0.5% Triton-X-100, 1 mM PMSF),冰静置15分钟,5000 ×离心gydF4y2BaggydF4y2Ba10分钟。4°C。废弃上清液(膜/细胞器部分),triton不溶颗粒在100µL的1x样品缓冲液中裂解,得到核部分。在10%的SDS-PAGE凝胶上分离相同的细胞质和核组分,并通过western blotting分析蛋白质。gydF4y2Ba
多核糖体分馏gydF4y2Ba
HEK-293T细胞在10cm培养皿中以5 × 10的速率培养gydF4y2Ba6gydF4y2Ba在DMEM中添加10% BGSS、1% Pen-Strep和1% l -谷氨酰胺(Wisent)。第二天,每个质粒用20µg磷酸钙转染细胞。清洗细胞,转染后6-8小时重施新鲜培养基。转染24小时后,在含有100µg/ml环己亚胺的冰冷PBS中收集细胞。细胞在4℃制粒,在425µL低渗裂解缓冲液(5 mM Tris-Cl (pH 7.5), 2.5 mM MgCl中裂解gydF4y2Ba2gydF4y2Ba, 1.5 mM KCl)。将环己酰亚胺(100µg/ml)、DTT (2 mM)、Triton-X-100(0.5%)和脱氧胆酸钠(0.5%)分别加入到指定的最终浓度中,并使样品短暂涡旋。以16000 ×离心清除裂解物gydF4y2BaggydF4y2Ba在4°C下加热2分钟。裂解液(400µL)分层到10-50%蔗糖梯度上。梯度在217,000 ×离心gydF4y2BaggydF4y2Ba在SW40贝克曼转子4°C下加热2小时。使用Teledyne ISCO Foxy R1收集器收集馏分,同时监测UV 254剖面。蛋白质用10%三氯乙酸沉淀,16000 × g离心30 min收集。4°C。用500µL丙酮清洗颗粒,4°C离心10 min。16000 ×gydF4y2BaggydF4y2Ba,并在真空下干燥(Eppendorf Vacufuge)。蛋白颗粒重悬于1× SDS样品缓冲液中,并在10% SDS- page凝胶上进行分析。溶解蛋白在4°C转移到PVDF膜(Bio-Rad)上,并通过免疫印迹检测。gydF4y2Ba
抗体gydF4y2Ba
本研究中使用的抗体有:抗nsp1 (GeneTex, GTX135612)、抗nsp10 (Pro-Sci Inc, #9179)、抗flag (Sigma-Aldrich, #F1804)、抗rpl7 (Novus Biologicals, #NB100-2268)、抗gapdh (Abcam, #ab8245)、抗β-actin (Abcam, #ab8226)、抗eef2 (CST, #2332)和抗hnrnpa1 (CST, #8443)。gydF4y2Ba
体外和细胞内蛋白质合成的测定gydF4y2Ba
对于体外翻译,使用SP6 RNA聚合酶(NEB, #M0207S)体外转录FF/EMCV/Ren双子报告子mRNA,并用m共转录封头gydF4y2Ba7gydF4y2BaGpppG RNA帽类比物(NEB, #S1404S)。用核酸酶处理的兔网织红细胞裂解液(Promega, #L4960)表达20 ng/µL mRNA,并添加相应的重组蛋白。重组蛋白与裂解物在30°C下预孵育5分钟,然后加入mRNA。在30°C下1 h后,将反应置于冰上,加入10µg/mL环己亚胺停止反应。荧光素酶活性在Berthold Lumat LB 9507光度计上测定。gydF4y2Ba
对于[gydF4y2Ba35gydF4y2BaS]-蛋氨酸/半胱氨酸标记,HEK-293T细胞以10的密度在6孔板中播种gydF4y2Ba6gydF4y2Ba在DMEM中添加10% BGSS、1% Pen-Strep和1% l -谷氨酰胺。转染后6-8小时,用指定的pcDNA3.1表达质粒转染细胞,用PBS洗涤三次,胰酶化,并在24孔板中播种,技术重复。转染24小时后,将培养基与不含蛋氨酸、半胱氨酸的DMEM (Gibco, #21013-024)补充10% FBS交换45分钟,然后用22 μ Ci的gydF4y2Ba35gydF4y2Ba每孔添加s -蛋氨酸/半胱氨酸蛋白标记混合物(Perkin Elmer, #NEG772007MC)。在37°C/5% CO下标记15分钟gydF4y2Ba2gydF4y2Ba细胞在RIPA缓冲液(20 mM Tris-Cl (pH 8.0), 100 mM NaCl, 1 mM EDTA, 1 mM EGTA, 1% NP-40, 0.5%脱氧胆酸钠,0.1% SDS, 0.01 mg/mL抑肽蛋白,0.002 mg/mL leupeptin, 2.5µM pepstatin和1 mM PMSF)中裂解。裂解物被标记在3MM Whatman纸上,蛋白质用10%三氯乙酸(TCA)沉淀。通过闪烁计数对tca不溶性放射性标记的蛋白质进行定量,并将计数归一化至每个样品的DC蛋白测定(Bio-Rad, #5000112)测定的总蛋白质量。裂解物也被SDS-PAGE分解,并转移到PVDF膜(Bio-Rad)时,western blot必须进行。gydF4y2Ba
重组蛋白纯化gydF4y2Ba
重组他的gydF4y2Ba6gydF4y2Ba从表达pet15b表达载体的BL21 (DE3)细胞中纯化得到Nsp1、Nsp1 (K164A/H165A)和Nsp1 - 10蛋白。采集单个菌落,培养20 mL过夜。以1 L LB/amp(100µg/mL)接种,IPTG (0.5 mM)诱导培养gydF4y2Ba600gydF4y2Ba达到0.8,此时培养物被移至18°C 16 h。离心20分钟,将细胞制成颗粒,再悬浮于20 mL Nsp1超声缓冲液(50 mM HEPES-KOH(pH 7.6), 500 mM KCl, 5 mM MgCl)中gydF4y2Ba2gydF4y2Ba, 40 mM咪唑,10%甘油,1 mM PMSF, 0.01 mg/mL抑肽酶,0.002 mg/mL leupeptin, 2.5 μ M pepstatin, 0.5 mM二硫苏糖醇),超声裂解(热系统超声;10个脉冲@ 1个脉冲/秒)。裂解物在4°C离心45分钟,48,000 ×gydF4y2BaggydF4y2Ba.蛋白质在Ni-NTA琼脂糖珠(Qiagen, #30210)上纯化,用Nsp1超声缓冲液洗涤两次,用Nsp1洗脱缓冲液(50 mM HEPES-KOH (pH 7.6), 500 mM KCl, 5 mM MgCl洗脱gydF4y2Ba2gydF4y2Ba, 300 mM咪唑,10%甘油)。洗脱蛋白部分在Nsp1透析缓冲液(40 mM HEPES-KOH (pH 7.6), 200 mM KCl, 4 mM MgCl)中在4℃下透析过夜gydF4y2Ba2gydF4y2Ba, 10%甘油,1mm二硫苏糖醇)。gydF4y2Ba
RNA纳米孔直接测序gydF4y2Ba
测序前,用“Qubit RNA高灵敏度”定量试剂盒(Q32855;(ThermoFisher Scientific),其质量在“高灵敏度RNA筛选带”(5067-5579;安捷伦科技公司)。只有高质量的样本被测序。总RNA在MinION流细胞(FLO-MIN106;Oxford Nanopore Technologies)使用“直接RNA测序”文库制备试剂盒(SQK-RNA002;牛津纳米孔技术公司)。我们遵循牛津纳米孔技术公司(简称ONT)提供的SQK-RNA002库制备协议(版本DRS_9080_v2_revM_14Aug2019),并进行了以下修改。文库制备开始时,实验1的第1、14和30段总RNA为2µg,实验2的第1和29段总RNA为1µg,实验2的第15段总RNA为3µg。gydF4y2Ba
在起始材料为1µg总RNA的情况下,我们使用以下方案。文库制备试剂盒的第一个适配器在15 μ l溶液中与以下成分连接在RNA上:3 μ l NEBNext快速连接反应缓冲液(原液:5×;B6058;NEB),总RNA 1µg,重组RNA酶抑制剂0.5µl(原液:40单位/µl;2313年;Takara), 1 μ l RT适配器(RTA;安大略省的);1.5 μ l T4 DNA连接酶(原液:2 M U/mL;M0202;New England Biolabs),将溶液加到15 μ l无核酸酶水。 This solution was incubated at room temperature for 20 min and subsequently mixed with a 23 µl solution named “reverse transcription master mix” that had the following components: 9 µl of nuclease-free water, 2 µl of 10 mM dNTPs (N0447S; NEB), 8 µl of 5× SSIV RT buffer (18090010; ThermoFisher Scientific), 4 µl of 0.1 M DTT (18090010; ThermoFisher Scientific). Next, 2 µl of SuperScript IV reverse transcriptase (18090010; ThermoFisher Scientific) were added, and the whole reaction was incubated at 50 °C for 2 h and 50 min, then at 70 °C for 10 min, and then the sample was brought to 4 °C before proceeding with a 1.8× volume of “RNAClean XP” beads cleanup (A63978; Beckman Coulter) and one wash of 150 µl with 70% EtOH. The material was then eluted from the beads with 20 µl of nuclease-free water, and the second adaptor was ligated in a 40 µl solution containing the following: 20 µl of reverse-transcribed RNA, 8 µl of NEBNext Quick Ligation Reaction Buffer (stock: 5×; B6058; NEB), 6 µl of RNA Adapter (RMX; ONT), 2.5 µl of nuclease-free water, 0.5 µl of Recombinant RNase Inhibitor (stock: 40 Units/µl; 2313 A; Takara), 3 µl of T4 DNA Ligase (stock: 2 M U/mL; M0202; NEB). The reaction was incubated at room temperature for 20 min followed by 1× volume of “RNAClean XP” beads cleanup (A63978; Beckman Coulter) and two washes of 150 µl with the Wash Buffer (WSB; ONT). The material was eluted, from the beads, in 21 µl of Elution Buffer, and 1 µl of the solution was quantified with the “Qubit 1X dsDNA high sensitivity” kit (Q3323; ThermoFisher Scientific). Approximately 200–250 ngs of RNA/cDNA hybrid were recovered. After priming the MinION flow-cell as per the ONT protocol we loaded the following solution: 20 µl of prepped RNA/cDNA hybrid in Elution Buffer, 17.5 µl of nuclease-free water, 37.5 µl of RRB buffer (ONT). The duration of the sequencing run was up to 72 h or until no pores were available for sequencing.
在起始材料为2或3µg总RNA的情况下,我们使用以下方案。将文库制备试剂盒的第一个适配器连接在含有以下成分的30 μ l溶液中的RNA上:6 μ l NEBNext快速连接反应缓冲液(原液:5×;B6058;NEB), 2或3µg总RNA, 1µl重组RNA酶抑制剂(原液:40单位/µl;2313年;Takara), 1 μ l RT适配器(RTA;安大略省的);3 μ l T4 DNA连接酶(原液:2 M U/mL;M0202 NEB),加无核酸酶水至30 μ l。溶液在室温下孵育20分钟,随后与46 μ l名为“逆转录主混合物”的溶液混合,其中含有以下成分:18 μ l无核酸酶水,4 μ l 10 mM dNTPs (N0447S; New England Biolabs), 16 µl of 5× SSIV RT buffer (18090010; ThermoFisher Scientific), 8 µ of 0.1 M DTT (18090010; ThermoFisher Scientific). Next, 4 µl of SuperScript IV reverse transcriptase (18090010; ThermoFisher Scientific) were added, and the whole reaction was incubated at 50 °C for 2 h and 50 min, then at 70 °C for 10 min, and the sample was brought to 4 °C before proceeding with a 1.8× volume of “RNAClean XP” beads cleanup (A63978; Beckman Coulter) and one wash of 300 µl with 70% EtOH. The material was then eluted from the beads with 40 µl of nuclease-free water, and the second adaptor was ligated in a 80 µl solution containing the following: 40 µl of reverse-transcribed RNA, 16 µl of NEBNext Quick Ligation Reaction Buffer (stock: 5×; B6058; New England Biolabs), 6 µl of RNA Adapter (RMX; ONT), 11 µl of nuclease-free water, 1 µl of Recombinant RNase Inhibitor (stock: 40 Units/µl; 2313 A; Takara), 6 µl of T4 DNA Ligase (stock: 2 M U/mL; M0202; New England Biolabs). The reaction was incubated at room temperature for 20 min followed by 1× volume of “RNAClean XP” beads cleanup (A63978; Beckman Coulter) and two washes of 150 µl with the Wash Buffer (WSB; ONT). The material was eluted, from the beads, in 38.5 µl of Elution Buffer, and 1 µl of the solution was quantified with the “Qubit 1X dsDNA high sensitivity” kit (Q33230; ThermoFisher Scientific). Approximately 400–750 ngs of RNA/cDNA hybrid were recovered. After priming the MinION flow-cell as per the ONT protocol we loaded the following solution: 37.5 µl of prepped RNA/cDNA hybrid in Elution Buffer, 37.5 µl of RRB buffer (ONT). The duration of the sequencing run was up to 72 h or until no pores were available for sequencing.
纳米孔数据分析管道和分析文件gydF4y2Ba
我们的纳米孔分析管道的详细描述在补充说明中提出gydF4y2Ba1克ydF4y2Ba.作为补充数据,提供了直接RNA纳米孔运行的汇总统计表gydF4y2Ba4gydF4y2Ba,并附在“补充说明”内的说明部分gydF4y2Ba2gydF4y2Ba和补充图中的附图。gydF4y2Ba13gydF4y2Ba.来自纳米孔分析管道的中间和最终文件作为补充数据提供gydF4y2Ba5克ydF4y2Ba.zip格式,并在补充说明中列出每个文件的说明gydF4y2Ba2gydF4y2Ba.gydF4y2Ba
统计和再现性gydF4y2Ba
使用GraphPad Prism软件(版本6.01;GraphPad软件公司,La Jolla, CA, USA)。结果以均数±标准差表示。平均比较使用双尾学生进行gydF4y2Bat -gydF4y2Ba检验或方差分析。为每个实验进行的生物独立重复的数量在图例中显示。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
分析中使用的所有数据和材料可供任何研究人员用于复制或扩展分析。未裁剪和未编辑的凝胶/印迹显示在补充图中。gydF4y2Ba11gydF4y2Ba而且gydF4y2Ba12gydF4y2Ba.所有主要和补充数据的数字源数据均在补充数据中提供gydF4y2Ba6gydF4y2Ba.本研究中使用的质粒(补充数据gydF4y2Ba3 gydF4y2Ba)已沉积在Addgene的以下登录代码:phiint1k GI.50 (191822), phiint1k GI.616 (191864), phiint1j GI.616/EMCV-Ren (191865), phiint1j GI.50/EMCV-Ren (191866), phiint1k GI.616/ΔNTD-Nsp1 (191867), phiint1k GI.616/Δ2NTD-Nsp1 (191868), phiint1k GI.616- trs Ren (191869), phRL-polyA (191870), pcDNA3.1 3xFLAG-Nsp1(191871)和pcDNA3.1 3xFLAG-Nasp1-10(191872),并可根据材料转让协议(MTA)获得。所有其他数据均可根据合理要求从通讯作者处获得。有关要求可向J.P. (jerry.pelletier@mcgill.ca)提出。原始测序数据的fastq文件以BioProject ID保存在公共存储库- sequence Read Archive数据库中gydF4y2BaPRJNA850004gydF4y2Ba48gydF4y2Ba.gydF4y2Ba
代码的可用性gydF4y2Ba
用于纳米孔序列分析的管道可在补充说明中获得gydF4y2Ba1克ydF4y2Ba及补充资料gydF4y2Ba5克ydF4y2Ba. zip文件。gydF4y2Ba
参考文献gydF4y2Ba
王,Y., Grunewald, M. & Perlman, S.冠状病毒:其复制和发病机制的最新概述。gydF4y2Ba方法分子生物学。gydF4y2Ba2203gydF4y2Ba, 1-29(2020)。gydF4y2Ba
Banerjee, A, Mossman, K. & Grandvaux, N. SARS-CoV-2变异的分子决定因素。gydF4y2BaMicrobiol趋势。gydF4y2Ba29gydF4y2Ba, 871-873(2021)。gydF4y2Ba
金,D.等人。SARS-CoV-2转录组结构。gydF4y2Ba细胞gydF4y2Ba181gydF4y2Ba, 914-921 e910(2020)。gydF4y2Ba
格里布尔等人。冠状病毒校对外核糖核酸酶介导广泛的病毒重组。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba17gydF4y2Ba, e1009226(2021)。gydF4y2Ba
van der Most, R. G., Bredenbeek, P. J. & Spaan, W. J.聚合酶基因3 '端结构域对冠状病毒缺陷干扰rna的封装至关重要。gydF4y2Baj .性研究。gydF4y2Ba65gydF4y2Ba, 3219-3226(1991)。gydF4y2Ba
Kim Y. N, Lai, m.m., Makino, S.通过RNA重组和可能的编辑生成和选择具有大开放阅读框的冠状病毒缺陷干扰RNA。gydF4y2Ba病毒学gydF4y2Ba194gydF4y2Ba, 244-253(1993)。gydF4y2Ba
Furuya, T., Macnaughton, t.b., La Monica, N.和Lai, m.m .冠状病毒缺陷干扰RNA的自然进化涉及RNA重组。gydF4y2Ba病毒学gydF4y2Ba194gydF4y2Ba, 408-413(1993)。gydF4y2Ba
林玉娟,赖敏敏。小鼠肝炎病毒干扰RNA缺陷的缺失图谱揭示了复制需要一个内部的不连续序列。gydF4y2Baj .性研究。gydF4y2Ba67gydF4y2Ba, 6110-6118(1993)。gydF4y2Ba
林玉娟,张旭,吴荣昌,赖敏敏,冠状病毒RNA的3 '非翻译区是由缺陷干扰RNA转录亚基因组mRNA所必需的。gydF4y2Baj .性研究。gydF4y2Ba70gydF4y2Ba, 7236-7240(1996)。gydF4y2Ba
牧野,S., Shieh, C. K., Keck, J. G. & Lai, m.m.小鼠冠状病毒缺陷干扰粒子:缺陷病毒rna的合成机制。gydF4y2Ba病毒学gydF4y2Ba163gydF4y2Ba, 104-111(1988)。gydF4y2Ba
Goebel, s.j., Miller, t.b., Bennett, c.j., Bernard, k.a.和Masters, p.s.小鼠冠状病毒基因组3 '顺式作用元件中的高变量区域对于RNA合成不是必需的,但会影响发病机制。gydF4y2Baj .性研究。gydF4y2Ba81gydF4y2Ba, 1274-1287(2007)。gydF4y2Ba
金,Y. N. &牧野,S.小鼠冠状病毒缺陷干扰RNA内部顺式作用复制信号的特征。gydF4y2Baj .性研究。gydF4y2Ba69gydF4y2Ba, 4963-4971(1995)。gydF4y2Ba
牧野,S, Fujioka, N. & Fujiwara, K.小鼠肝炎病毒缺陷干扰颗粒细胞内缺陷病毒rna的结构。gydF4y2Baj .性研究。gydF4y2Ba54gydF4y2Ba, 329-336(1985)。gydF4y2Ba
Makino, S., Yokomori, K.和Lai, m.m.对小鼠冠状病毒有效包装的缺陷干扰rna的分析:可能的rna包装信号的定位。gydF4y2Baj .性研究。gydF4y2Ba64gydF4y2Ba, 6045-6053(1990)。gydF4y2Ba
Mendez, A., Smerdou, C., Izeta, A., Gebauer, F. & Enjuanes, L.传染性胃肠炎冠状病毒缺陷干扰基因组的分子特征:包装和异质性。gydF4y2Ba病毒学gydF4y2Ba217gydF4y2Ba, 495-507(1996)。gydF4y2Ba
Izeta, A.等人。传染性胃肠炎冠状病毒合成小基因组的复制和包装。gydF4y2Baj .性研究。gydF4y2Ba73gydF4y2Ba, 1535-1545(1999)。gydF4y2Ba
张,R. Y., Hofmann, M. A., Sethna, P. B. & Brian, D. A.在缺陷干扰RNA复制中冠状病毒领导者的顺式作用功能。gydF4y2Baj .性研究。gydF4y2Ba68gydF4y2Ba, 8223-8231(1994)。gydF4y2Ba
张,R. Y.和Brian, D. A. cis牛冠状病毒干扰RNA复制缺陷中n特异性蛋白序列的需求。gydF4y2Baj .性研究。gydF4y2Ba70gydF4y2Ba, 2201-2207(1996)。gydF4y2Ba
Williams, G. D., Chang, R. Y. & Brian, D. A.冠状病毒RNA复制中系统发育保守的发夹型3 '非翻译区假结功能。gydF4y2Baj .性研究。gydF4y2Ba73gydF4y2Ba, 8349-8355(1999)。gydF4y2Ba
Raman, S., Bouma, P., Williams, G. D.和Brian, D. a . 5 '非翻译区的Stem-loop III是牛冠状病毒缺陷干扰RNA复制的顺式作用元件。gydF4y2Baj .性研究。gydF4y2Ba77gydF4y2Ba, 6720-6730(2003)。gydF4y2Ba
Brown, C. G., Nixon, K. S., Senanayake, S. D.和Brian, D. a .牛冠状病毒nsp1编码区内的RNA茎环是干扰RNA复制缺陷的顺式作用元件。gydF4y2Baj .性研究。gydF4y2Ba81gydF4y2Ba, 7716-7724(2007)。gydF4y2Ba
Penzes, Z.等人。禽冠状病毒传染性支气管炎病毒复制和包装缺陷RNA的特性。gydF4y2Ba病毒学gydF4y2Ba203gydF4y2Ba, 286-293(1994)。gydF4y2Ba
道尔顿等人。参与拯救IBV缺陷RNA CD-91的序列元件。gydF4y2Ba实验、医学、生物学。gydF4y2Ba440gydF4y2Ba, 253-257(1998)。gydF4y2Ba
Thiel, V. Siddell, S. G. & Herold, J. HCV 229E复制子的复制和转录。p6。gydF4y2Ba实验、医学、生物学。gydF4y2Ba440gydF4y2Ba, 109-113(1998)。gydF4y2Ba
Yao, S., Narayanan, A., Majowicz, S. A., Jose, J. & Archetti, M.干扰SARS-CoV-2的合成缺陷。gydF4y2BaPeerJgydF4y2Ba9gydF4y2Ba, e11686(2021)。gydF4y2Ba
查图维迪,S.等。一种治疗性干扰颗粒的鉴定——具有高耐药屏障的单剂量SARS-CoV-2抗病毒干预。gydF4y2Ba细胞gydF4y2Bahttps://doi.org/10.1016/j.cell.2021.11.004gydF4y2Ba(2021)。gydF4y2Ba
Shirogane, Y.等。脊髓灰质炎病毒和有缺陷的干扰基因组之间相互作用的实验和数学见解。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba17gydF4y2Ba, e1009277(2021)。gydF4y2Ba
吴海燕等。2组冠状病毒之间存在常见的RNA复制信号:动物和人类冠状病毒分子在体内重组的证据。gydF4y2Ba病毒学gydF4y2Ba315gydF4y2Ba, 174-183(2003)。gydF4y2Ba
芬克尔,Y.等。SARS-CoV-2采用多管齐下的策略来阻碍宿主蛋白质合成。gydF4y2Ba自然gydF4y2Ba594gydF4y2Ba, 240-245(2021)。gydF4y2Ba
张,K.等。SARS-CoV-2的Nsp1蛋白破坏mRNA输出机制,抑制宿主基因表达。gydF4y2Ba科学。睡觉。gydF4y2Bahttps://doi.org/10.1126/sciadv.abe7386gydF4y2Ba(2021)。gydF4y2Ba
舒伯特等人。SARS-CoV-2 Nsp1结合核糖体mRNA通道抑制翻译。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba27gydF4y2Ba, 959-966(2020)。gydF4y2Ba
Tidu, A.等人。病毒蛋白NSP1作为核糖体看门人,关闭宿主翻译并促进SARS-CoV-2翻译。gydF4y2Ba核糖核酸gydF4y2Bahttps://doi.org/10.1261/rna.078121.120gydF4y2Ba(2020)。gydF4y2Ba
托马斯等人。SARS-CoV-2 Nsp1蛋白翻译关闭和免疫逃避的结构基础gydF4y2Ba科学gydF4y2Ba369gydF4y2Ba, 1249-1255(2020)。gydF4y2Ba
袁,S.等。SARS-CoV-2的非结构蛋白1是一种有效的致病因子,可将宿主蛋白质合成机制定向于病毒RNA。gydF4y2Ba摩尔。细胞gydF4y2Ba80gydF4y2Ba, 1055-1066 e1056(2020)。gydF4y2Ba
Lapointe, c.p.等。SARS-CoV-2 NSP1与人核糖体上mRNA的动态竞争抑制翻译启动。gydF4y2Ba国家科学院学报美国gydF4y2Bahttps://doi.org/10.1073/pnas.2017715118gydF4y2Ba(2021)。gydF4y2Ba
戈登,C. J.等。瑞德西韦是一种直接作用的抗病毒药物,可高效抑制严重急性呼吸综合征冠状病毒2的RNA依赖RNA聚合酶。gydF4y2Ba生物。化学。gydF4y2Ba295gydF4y2Ba, 6785-6797(2020)。gydF4y2Ba
Noble, S. & Dimmock, N. J.从受甲型流感病毒/WSN (H1N1)致命呼吸道感染保护的小鼠肺部分离出的假定缺陷干扰(DI) A/WSN rna的特征:接种体DI rna的一个子集。gydF4y2Ba病毒学gydF4y2Ba210gydF4y2Ba, 9-19(1995)。gydF4y2Ba
丁莫克,n.j,雷恩斯福德,e.w,斯科特,P. D.和马里奥特,a.c.流感病毒保护RNA:一种有效的预防和治疗抗病毒药物。gydF4y2Baj .性研究。gydF4y2Ba82gydF4y2Ba, 8570-8578(2008)。gydF4y2Ba
杨,Y.等。缺陷干扰粒子/基因组的抗病毒和抗肿瘤作用及其机制。gydF4y2Ba前面。Microbiol。gydF4y2Ba10gydF4y2Ba, 1852(2019)。gydF4y2Ba
Rezelj, V. V.等。缺陷病毒基因组作为治疗性干扰粒子对抗哺乳动物和蚊子宿主的黄病毒感染。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 2290(2021)。gydF4y2Ba
李维,L. I.等。基孔肯雅病毒的缺陷病毒基因组是广谱抗病毒药物,可防止病毒在蚊子中传播。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba17gydF4y2Ba, e1009110(2021)。gydF4y2Ba
黄,C.等。SARS冠状病毒nsp1蛋白诱导模板依赖的mrna内核裂解:病毒mrna对nsp1诱导的RNA裂解具有抗性。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba7gydF4y2Ba, e1002433(2011)。gydF4y2Ba
Kamitani, W., Huang, C., Narayanan, K., Lokugamage, K. G. & Makino, S.一种双管策略抑制SARS冠状病毒Nsp1蛋白的宿主蛋白合成。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba16gydF4y2Ba, 1134-1140(2009)。gydF4y2Ba
Alhatlani, B. Y.在SARS-CoV-2基因组中保守顺式作用RNA元件的硅鉴定。gydF4y2Ba未来的性。gydF4y2Ba15gydF4y2Ba, 409-417(2020)。gydF4y2Ba
齐夫,O.等人。SARS-CoV-2的短期和远程RNA-RNA相互作用组。gydF4y2Ba摩尔。细胞gydF4y2Ba80gydF4y2Ba, 1067-1077 e1065(2020)。gydF4y2Ba
黄晓明,黄晓明,黄晓明,等。有缺陷的干扰颗粒引起水泡性口炎病毒的循环生成。gydF4y2Baj .感染。说。gydF4y2Ba129gydF4y2Ba, 402-410(1974)。gydF4y2Ba
肖,杨,等。一个有缺陷的病毒基因组策略引起广泛的呼吸道病毒的保护性免疫。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 6037-6051 e6014(2021)。gydF4y2Ba
Leinonen, R., Sugawara, H., Shumway, M. &国际核苷酸序列数据库,C.序列读取存档。gydF4y2Ba核酸测定。gydF4y2Ba39gydF4y2Ba, d19-d21(2011)。gydF4y2Ba
确认gydF4y2Ba
我们要感谢Patrick Sénéchal, Emma Woolner和Dana kocinccova出色的技术支持。这项工作的资金来自:科学基金会爱尔兰基因组学数据科学研究培训中心(a.d.f.: 18/CRT/6214)、加拿大卫生研究所(m.g.: # 170343;J.p, p.b., t.c.h.: # vr3-172647;t.c.h.: cihr hal-201803;和J.R.: CIHR CoVaRR-Net),加拿大基因组基因组技术平台拨款(J.R.),加拿大创新基金会4014,41012,35444 (J.R.),以及理查德和伊迪丝·施特劳斯加拿大基金会(J.P.)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
概念化:S.G, Z.X P.V.B。,j, r T.C.H,和摩根大通(J.P. Metholodology: S.G, Z.X, S.O。j。r A.D.F, E.P.T, C.J.G,和摩根大通(J.P.调查:S.G, Z.X, S.O, A.D.F, E.P.T, C.J.G, T.M.S,和摩根大通(J.P.可视化:S.G, Z.X, S.O, A.D.F, T.M.S,和摩根大通(J.P.监督:M.G, n, P.V.B。j。r T.C.H,和摩根大通(J.P.原创作品:草案S.G, Z.X, S.O, T.C.H,和摩根大通(J.P. Writing-review和编辑:S.G Z.X。,S.O A.D.F, E.P.T, C.J.G, T.M.S, M.G, n, P.V.B, T.C.H,摩根大通(J.P. j。rgydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明了以下相互竞争的利益:s.g., Z.X, s.o., a.d.f., p.b., j.r., t.c.h.和J.P.是一项多肽干扰冠状病毒复制专利申请的共同发明人。其余作者声明没有竞争利益。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
通信生物学gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。首席处理编辑:Gene Chong。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
吉吉斯,徐,泽,奥伊科诺波洛斯,S。gydF4y2Baet al。gydF4y2Ba自然产生的SARS-CoV-2缺陷干扰粒子的进化。gydF4y2BaCommun杂志gydF4y2Ba5克ydF4y2Ba, 1140(2022)。https://doi.org/10.1038/s42003-022-04058-5gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s42003-022-04058-5gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba